Modelo Brusselator de Reação-Difusão: mudanças entre as edições
| Linha 150: | Linha 150: | ||
==Implementação== | ==Implementação== | ||
'''Código completo no GitHub''' | '''Código completo no GitHub<ref name='github'>GitHub RicGary</ref>''' | ||
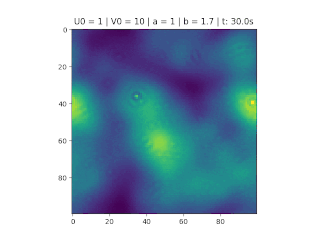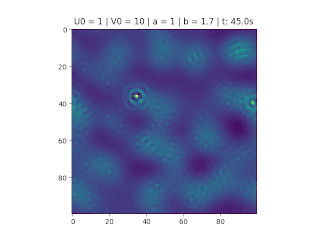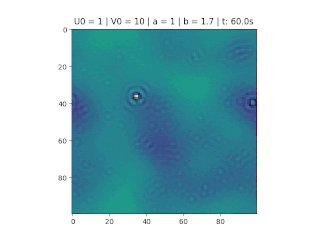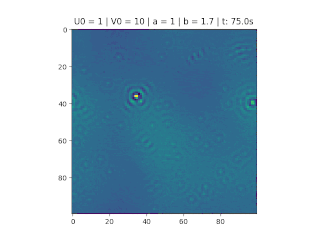
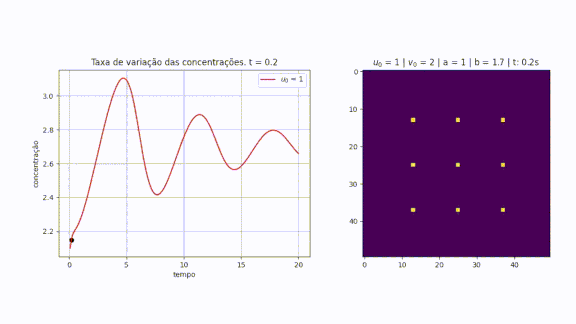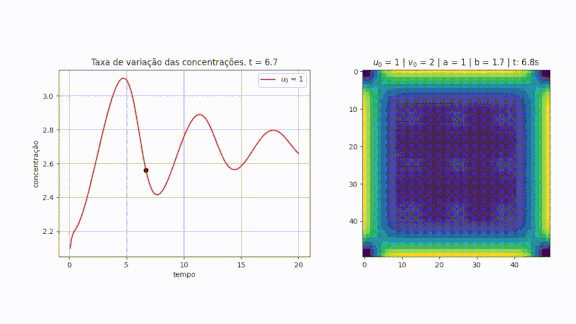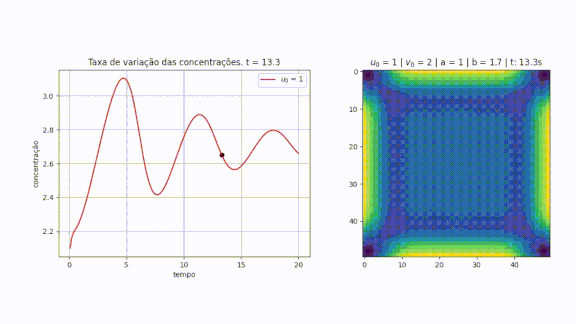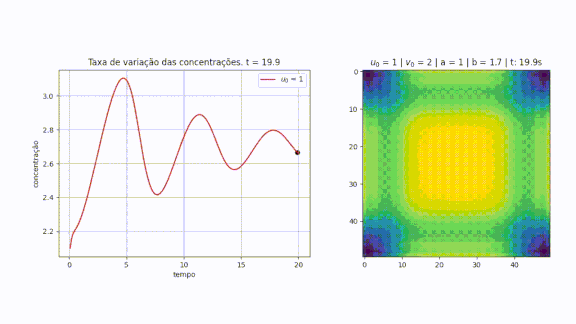
O método foi implementado em Python, considerando <math>\Delta t = 0.1, \Delta s = 1, N_x = N_y = 50</math>, variando as constantes <math>a</math> e <math>b</math> e as condições iniciais do problema. | O método foi implementado em Python, considerando <math>\Delta t = 0.1, \Delta s = 1, N_x = N_y = 50</math>, variando as constantes <math>a</math> e <math>b</math> e as condições iniciais do problema. | ||
Edição das 00h33min de 11 de março de 2022
Grupo: Carolina Lenzi, Eric Naiber e Vitória Xavier
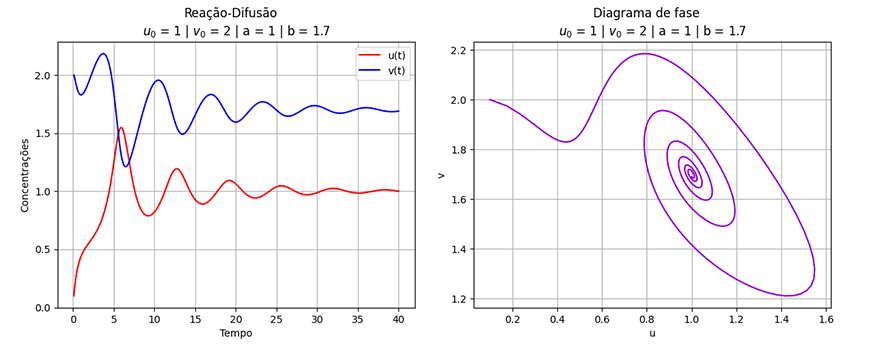
O objetivo deste trabalho é implementar o modelo de reação-difusão Brusselator em duas dimensões, frequentemente utilizado para estudar sistemas complexos químicos e biológicos. O modelo é um sistema não linear de equações diferenciais parciais e foi proposto em 1970 por Ilya Prigogine e seus colaboradores da Universidade Livre de Bruxelas. Desde então tem sido aplicado para analisar reações oscilatórias e autocatalíticas. O método computacional utilizado para implementar o modelo foi o método FTCS (Forward Time Centered Space).
Modelo de Brusselator
O estudo de sistemas químicos e biológicos frequentemente requer o uso de modelos que caracterizam reações de reação-difusão. Um dos modelos mais utilizados é o modelo de Brusselator, que é utilizado para descrever o mecanismo químico de reação-difusão com oscilações não lineares. [J. Tyson, Some further studies of nonlinear oscillations in chemical systems, J. Chem. Phys. 58 (1973) 3919.] Turing [ref?] observou que quando determinadas reações são associadas a difusão, é possível obter um padrão espacial estável, e isso leva a teoria de morfogênese. Além de processos de reação-difusão, o modelo Brusselator é observado em reações enzimáticas e na física de plasma e de lasers.
O mecanismo de Brusselator proposto por Prigogine (1970) é dado por [I. Prigogine, R. Lefever, Symmetries breaking instabilities in dissipative systems II. J. Phys. Chem. 48, 1695–1700 (1968)]:
- (1.a)
- (1. b)
- (1.c)
- (1.d)
Onde U e V são as espécies químicas de interesse. Assumimos A e B em excesso para que o sistema não atinja o equilíbrio. Esse sistema químico foi importante para o avanço na área de sistemas complexos porque possibilita o uso de modelos matemáticos de duas dimensões, já que U e V são variáveis dependentes, e admite “limit-cycle oscilations”. [ R. Lefever and G. Nicolis, Chemical instabilities and sustained oscillations, J. Theor. Biol. 30 (1971) 267.].
As equações diferenciais parciais associadas com o sistema Brusselator são dadas por(G. Adomian, The diffusion-Brusselator equation. Comput. Math. Appl. 29, 1–3 (1995)):
onde e são as concentrações a serem investigadas em função de tempo e espaço, e são constantes relativas às concentrações dos reagentes A e B, e e constantes de difusão.
A solução analítica do sistema reação-difusão Brusselator ainda não é conhecida e por isso há o interesse de explorá-la numericamente.
Análise da estabilidade do sistema
Análise de ponto crítico
Considerando o sistema livre de difusão, quando :
Onde e e e são constantes positivas e reais. A matriz jacobiana no ponto crítico é dada por
Os autovalores de são os valores que satisfazem a equação caracterísitca
Os autovalores claramente mostram dependência em e no determinante . Esses autovalores governam a estabilidade do ponto crítico ou determinam a existência de um ciclo limite. As propriedades de estabilidade ou a existência de um ciclo limite estão sumarizadas na tabela abaixo, em relação a figura 1.
[[Arquivo:]]
Utilizando a teoria Hopf, é mostrado que o ponto crítico perde sua estabilidade quando A e B movem da região 2 para região 3, na figura 1, atravessando a curva . Uma bifurcação Hopf ocorre quando ao passo que essa curva é atravessada e um ciclo limite estável existe para A e B nas regiões 1 e 2, mas não para A e B nas regiões 3 e 4.
Conclui-se que a curva governa a estabilidade do sistema.
Análise de ponto fixo
O estado estacionário do sistema pode ser encontrado igualando o coeficiente de difusão, e portanto as derivadas parciais, a zero. Percebe-se que esse sistema converge para os pontos fixos:
Em [twizell] a manipulação da matriz jacobiana nos pontos fixos resulta nos seguintes autovalores
Onde fica claro que os denominadores dos autovalores são sempre positivos quando and . As inequações
- e
São verdadeiras sempre que . Portanto, uma condição suficiente para o ponto fixo atrair a sequência gerada pelo sistema é .
Ainda em [Twizell] o modelo reação-difusão Brusselator foi discretizado e a análise dos pontos fixos concluiu que o sistema converge para e , sendo esse o único estado estacionário do sistema.
Concluiu que também que o sistema apresenta estado oscilatório quando
Estado em que o sistema não converge para nenhum ponto.
Método FTCS
O FTCS (Forward Time Centered Space) é um método de diferença finita que utiliza a derivada à direita ("para frente") no tempo e a derivada segunda centralizada no espaço para discretizar as variáveis. As derivadas no tempo e no espaço bidimensional ficam:
Substituindo nas equações do Brusselator
onde e são as funções que representam a reação sem difusão.
Utilizamos discretização do tipo
Utilizando a notação , assumindo e rearranjando os termos, reescrevemos as equações como
onde e .
Análise de estabilidade do método
Implementação
Código completo no GitHub[1]
O método foi implementado em Python, considerando , variando as constantes e e as condições iniciais do problema.
"""
Esta é uma versão reduzida do código, sem a parte da formação dos gifs e de algumas imagens.
Para a versão oficial utilizada no trabalho, acesse o GitHub forncecido.
"""
import numpy as np
import matplotlib.pyplot as plt
# Constantes
Nx = Ny = 25
a = 1
b = 1.7
# Valores iniciais
u0 = 1
v0 = 2
t = 0
# Constantes do sistema
t_max = 40
dt = 0.1
ds = 1
# Constantes dos reagentes e da estabilidade
Du = 0.1
Dv = 1
ku = Du * dt / (ds ** 2)
kv = Dv * dt / (ds ** 2)
# Pedaço da equação sem derivada parcial
def f(u, v):
return a - (b + 1) * u + u * u * v
# Pedaço da equação sem derivada parcial
def g(u, v):
return b * u - u * u * v
# vetores no tempo n
u_n = np.zeros((Nx, Ny))
v_n = np.zeros((Nx, Ny))
# vetores no tempo n+1
u_n1 = np.zeros((Nx, Ny))
v_n1 = np.zeros((Nx, Ny))
# Nove pontos centrais (u0)
mid = int(Nx / 2) # Centro
mov = int(Nx / 4) # Movendo para cima e para os lados
u_n[mid, mid] = u_n[mid + mov, mid + mov] = u_n[mid + mov, mid] = u_n[mid, mid + mov] = u_n[mid + mov, mid - mov] = u0
u_n[mid - mov, mid - mov] = u_n[mid - mov, mid] = u_n[mid, mid - mov] = u_n[mid - mov, mid + mov] = u0
# Toda a borda (v0)
for i in range(Nx):
v_n[0, i] = v0
v_n[i, 0] = v0
v_n[Nx - 1, i] = v0
v_n[i, Nx - 1] = v0
# Algumas listas são extras, não precisam realmente estar ali, estão apenas para organização.
lista_t = []
lista_u = []
lista_v = []
# Calculando concentrações com FTCS
while t < t_max:
for i in range(Nx):
i_e = (i - 1) % Nx # vizinho a esquerda de 0 é o da ultima posicao
i_d = (i + 1) % Nx # vizinho a direita da ultima posicao é o zero
for j in range(Ny):
j_e = (j - 1) % Ny
j_d = (j + 1) % Ny
u_n1[i, j] = u_n[i, j] + dt * f(u_n[i, j], v_n[i, j]) \
+ ku * (u_n[i_e, j] + u_n[i_d, j] + u_n[i, j_e] + u_n[i, j_d] - 4 * u_n[i, j])
v_n1[i, j] = v_n[i, j] + dt * g(u_n[i, j], v_n[i, j]) \
+ kv * (v_n[i_e, j] + v_n[i_d, j] + v_n[i, j_e] + v_n[i, j_d] - 4 * v_n[i, j])
lista_u.append(u_n1[0][0])
lista_v.append(v_n1[0][0])
# atualizar u_n e v_n
for i in range(Nx):
for j in range(Ny):
u_n[i, j] = u_n1[i, j]
v_n[i, j] = v_n1[i, j]
t += dt
lista_t.append(t)
print(f'{round(t / t_max * 100, 3)}%')
plt.plot(lista_t, lista_u, color='red', label='u(t)')
plt.plot(lista_t, lista_v, color='blue', label='v(t)')
plt.grid(True)
plt.suptitle(f'Reação-Difusão')
plt.title(f'$u_0$ = {u0} | $v_0$ = {v0} | a = {a} | b = {b}')
plt.xlabel('Tempo')
plt.ylabel('Concentrações')
plt.legend()
plt.savefig('Reação-Difusão')
plt.show()
Referências
- ↑ GitHub RicGary