Modelo Brusselator de Reação-Difusão: mudanças entre as edições
| (256 revisões intermediárias por 3 usuários não estão sendo mostradas) | |||
| Linha 1: | Linha 1: | ||
''' Grupo: Carolina Lenzi, Eric Naiber e Vitória Xavier ''' | ''' Grupo: Carolina Lenzi, Eric Naiber e Vitória Xavier ''' | ||
O objetivo deste trabalho é implementar o modelo de reação-difusão Brusselator em duas dimensões, frequentemente utilizado para estudar sistemas complexos químicos e biológicos. O modelo é um sistema não linear de equações diferenciais parciais e foi proposto em 1968 por Ilya Prigogine e seus colaboradores da Universidade Livre de Bruxelas. Desde então tem sido aplicado para analisar reações oscilatórias e autocatalíticas. O método computacional utilizado para implementar o modelo foi o método FTCS (''Forward Time Centered Space''). | |||
O objetivo deste trabalho é implementar o modelo de reação-difusão Brusselator em duas dimensões, frequentemente utilizado para estudar sistemas complexos químicos e biológicos. O modelo é um sistema não linear de equações diferenciais parciais e foi proposto em | |||
==Modelo de Brusselator== | ==Modelo de Brusselator== | ||
O estudo de sistemas químicos e biológicos frequentemente requer o uso de modelos que caracterizam reações de reação-difusão. Um dos modelos mais utilizados é o modelo de Brusselator, que é utilizado para descrever o mecanismo químico de reação-difusão com oscilações não lineares. <ref>J. Tyson, Some further studies of nonlinear oscillations in chemical systems, J. Chem. Phys. 58 (1973) 3919</ref>. Turing observou que quando determinadas reações são associadas a difusão, é possível obter um padrão espacial estável, e isso leva a teoria de morfogênese<ref>Turing A. M., The chemical basis of morphogenesis. 1953. Bull. Math. Biol. 52, 153, discussion 119 (1990).</ref>. Além de processos de reação-difusão, o modelo Brusselator é observado em reações enzimáticas e na física de plasma e de lasers. | |||
O mecanismo de Brusselator proposto por Prigogine<ref>I. Prigogine, R. Lefever, Symmetries breaking instabilities in dissipative systems II. J. Phys. Chem. 48, 1695–1700 (1968)</ref> é dado por: | |||
O mecanismo de Brusselator proposto por Prigogine | |||
:<math>A \to U</math> (1.a) | :<math>A \to U</math> (1.a) | ||
| Linha 18: | Linha 14: | ||
:<math>U \to E</math> (1.d) | :<math>U \to E</math> (1.d) | ||
Onde U e V são as espécies químicas de interesse. Assumimos A e B em excesso para que o sistema não atinja o equilíbrio. Esse sistema químico foi importante para o avanço na área de sistemas complexos porque possibilita o uso de modelos matemáticos de duas dimensões, já que U e V são variáveis dependentes, e admite | Onde U e V são as espécies químicas de interesse. Assumimos A e B em excesso para que o sistema não atinja o estado de equilíbrio. Esse sistema químico foi importante para o avanço na área de sistemas complexos porque possibilita o uso de modelos matemáticos de duas dimensões, já que U e V são variáveis dependentes, e admite oscilações de ciclo limite<ref>R. Lefever and G. Nicolis, Chemical instabilities and sustained oscillations, J. Theor. Biol. 30 (1971)</ref>. | ||
As equações diferenciais parciais associadas com o sistema Brusselator | As equações diferenciais parciais associadas com o sistema Brusselator<ref>G. Adomian, The diffusion-Brusselator equation. Comput. Math. Appl. 29, 1–3 (1995)</ref> são dadas por: | ||
:<math>\frac{\partial u}{\partial t} = a + u^2v - (b+1)u + D_u \left(\frac{\partial ^2 u}{\partial x^2} + \frac{\partial ^2 u}{\partial y^2} \right)</math> | :<math>\frac{\partial u}{\partial t} = a + u^2v - (b+1)u + D_u \left(\frac{\partial ^2 u}{\partial x^2} + \frac{\partial ^2 u}{\partial y^2} \right)</math> | ||
:<math>\frac{\partial v}{\partial t} = bu - u^2v + D_v \left(\frac{\partial ^2 v}{\partial x^2} + \frac{\partial ^2 v}{\partial y^2}\right)</math> | :<math>\frac{\partial v}{\partial t} = bu - u^2v + D_v \left(\frac{\partial ^2 v}{\partial x^2} + \frac{\partial ^2 v}{\partial y^2}\right)</math> | ||
onde <math>u(x, y, t)</math> e <math>v(x, y, t)</math> são as concentrações a serem investigadas em função de tempo e espaço, <math>a</math> e <math>b</math> são constantes relativas às concentrações dos reagentes A e B, e <math>D_u</math> e <math>D_v</math> constantes de difusão. | onde <math>u(x, y, t)</math> e <math>v(x, y, t)</math> são as concentrações a serem investigadas em função de tempo e espaço, <math>a</math> e <math>b</math> são constantes relativas às concentrações dos reagentes A e B, e <math>D_u</math> e <math>D_v</math> são as constantes de difusão de U e V. | ||
A solução analítica do sistema reação-difusão Brusselator ainda não é conhecida e por isso há o interesse de explorá-la numericamente. | A solução analítica do sistema reação-difusão Brusselator ainda não é conhecida e por isso há o interesse de explorá-la numericamente. | ||
| Linha 33: | Linha 29: | ||
===Análise de ponto crítico=== | ===Análise de ponto crítico=== | ||
Considerando o sistema livre de difusão, quando <math> | Considerando o sistema livre de difusão, quando <math>D_u = D_v = 0</math>: | ||
:<math> \frac{du}{dt} = f(u, v) = a + u^2v - (b+1)u | :<math> \frac{du}{dt} = f(u, v) = a + u^2v - (b+1)u </math> | ||
:<math> \frac{dv}{dt} = g(u, v) = bu - u^2v | :<math> \frac{dv}{dt} = g(u, v) = bu - u^2v </math> | ||
Encontramos os pontos críticos do sistema igualando as derivadas à zero. Obtemos que o único ponto crítico é <math>(u^*, v^*) = \left ( a, \frac{b}{a} \right)</math>. | |||
A matriz jacobiana <math>J^*</math> no ponto crítico <math>(u^*, v^*)</math> é dada por | |||
:<math> | :<math> | ||
J^* = \begin{bmatrix} | J^* = \begin{bmatrix} | ||
\frac{\partial f}{\partial u} \Big |_{u^*, v^*} & \frac{\partial f}{\partial v} \Big |_{u^*, v^*} \\ | |||
\frac{\partial g}{\partial u} \Big |_{u^*, v^*} & \frac{\partial g}{\partial v} \Big |_{u^*, v^*} | |||
\end{bmatrix} | |||
= | |||
\begin{bmatrix} | |||
b-1 & a^2 \\ | b-1 & a^2 \\ | ||
-b & -a^2 | -b & -a^2 | ||
| Linha 54: | Linha 55: | ||
:<math> \lambda ^2 + (1 - b + a^2)\lambda + a^2 = 0 </math> | :<math> \lambda ^2 + (1 - b + a^2)\lambda + a^2 = 0 </math> | ||
Os autovalores claramente mostram dependência em <math>1-b+a^2</math> e no determinante <math>\Delta = (1-b+ a^2)^2 - 4a^2</math>. Esses autovalores governam a estabilidade do ponto crítico | Os autovalores claramente mostram dependência em <math>1-b+a^2</math> e no determinante <math>\Delta = (1 - b + a^2)^2 - 4a^2</math>. Esses autovalores governam a estabilidade do ponto crítico e determinam a existência de um ciclo limite. Conforme discutido em Twizell<ref name='t'>Twizell, E. H., Gumel, A. B., & Cao, Q. (1999). Journal of Mathematical Chemistry, 26(4), 297–316 </ref>, quando <math>Re(\lambda) < 0 </math> o sistema é estável e não existe ciclo limite. Já para <math>Re(\lambda) > 0 </math> o sistema é instável e existe ciclo limite. | ||
===Análise de ponto fixo=== | ===Análise de ponto fixo=== | ||
Ainda em Twizell<ref name='t'>Twizell, E. H., Gumel, A. B., & Cao, Q. (1999). Journal of Mathematical Chemistry, 26(4), 297–316 </ref>, foi feita a análise de ponto fixo. O único ponto fixo encontrado é <math>(u^*, v^*) = \left ( a, \frac{b}{a} \right)</math>. Manipulando a matriz Jacobiana nesse ponto e analisando os autovalores, obtém-se que a solução é atraída para o ponto <math>\left ( a, \frac{b}{a} \right)</math> se <math>1-b+a^2 > 0</math>. Caso contrário, a solução não converge. | |||
==Método FTCS== | ==Método FTCS== | ||
| Linha 141: | Linha 110: | ||
onde <math>K_u = \frac{D_u \Delta t}{(\Delta s)^2}</math> e <math>K_v = \frac{D_v \Delta t}{(\Delta s)^2}</math>. | onde <math>K_u = \frac{D_u \Delta t}{(\Delta s)^2}</math> e <math>K_v = \frac{D_v \Delta t}{(\Delta s)^2}</math>. | ||
===Análise de estabilidade do método== | ===Análise de estabilidade=== | ||
A análise de estabilidade do método FTCS pode ser feita com a análise local de von Neumann. Para isso precisamos linearizar as equações do modelo Brusselator e escrever as soluções em modos de Fourier, da seguinte forma: | |||
:<math> | |||
\delta u_{i, j}^{n+1} = \delta u_{i, j}^n + K_u \left( \delta u_{i+1, j}^n + \delta u_{i-1, j}^n + \delta u_{i, j+1}^n + \delta u_{i, j-1}^n - 4 \delta u_{i, j}^n \right) + \Delta t f_u \delta u_{i, j}^n + \Delta t f_v \delta v_{i, j}^n | |||
</math> | |||
:<math> | |||
\delta v_{i, j}^{n+1} = \delta v_{i, j}^n + K_v \left( \delta v_{i+1, j}^n + \delta v_{i-1, j}^n + \delta v_{i, j+1}^n + \delta v_{i, j-1}^n - 4 \delta v_{i, j}^n \right) + \Delta t g_u \delta u_{i, j}^n + \Delta t g_v \delta v_{i, j}^n | |||
</math> | |||
:<math> | |||
{\delta u^n \choose \delta v^n} = \xi ^n e^{i(k_x i \Delta x + k_y j \Delta y)} {\delta u_0 \choose \delta v_0} | |||
</math> | |||
Para que o método seja estável, é preciso que <math>|\xi| < 1</math>. Após inserir as soluções nas equações linearizadas e realizar manipulações algébricas, como feito por Scholz<ref name='s'>Scholz, Christian, Morphology of Experimental and Simulated Turing Patterns, 2009, p. 27-29.</ref>, obtemos | |||
:<math> | |||
\xi_\pm = \frac{1}{2} \left [ \beta \pm \sqrt{\beta ^2 - 4 (\Delta t)^2 a^2 b} \right ] | |||
</math> | |||
onde | |||
:<math>\beta = 2 - 4 \gamma (K_u + K_v) + \Delta t (b - 1 - a^2)</math> | |||
e | |||
:<math>\gamma = sin^2\left( \frac{k_x \Delta s}{2} \right) + sin^2\left( \frac{k_y \Delta s}{2} \right)</math> | |||
Os valores <math>\Delta t = 0.01</math> e <math>\Delta s = 1</math> satisfazem a condição de estabilidade, como mostrado em Scholz<ref name='s'>Scholz, Christian, Morphology of Experimental and Simulated Turing Patterns, 2009, p. 27-29.</ref>. | |||
==Implementação== | ===Implementação=== | ||
A seguir, o trecho do código relativo à implementação do método na linguagem Python. O código completo encontra-se no Github [https://github.com/RicGary/MetCompC/tree/main/Brusselator] | |||
<source lang="python"> | <source lang="python"> | ||
# u_n, v_n -> concentracao dos reagentes no tempo n (matriz Nx x Ny) | |||
# u_n1, v_n1 -> concentracao dos reagentes no tempo n+1 | |||
while t < t_max: | |||
for i in range(Nx): | |||
Nx | i_e = (i - 1) % Nx # vizinho a esquerda de 0 é o da ultima posicao | ||
i_d = (i + 1) % Nx # vizinho a direita da ultima posicao é o zero | |||
for j in range(Ny): | |||
j_e = (j - 1) % Ny | |||
j_d = (j + 1) % Ny | |||
# calcular u e v no tempo n+1 | |||
u_n1[i, j] = u_n[i, j] + dt * f(u_n[i, j], v_n[i, j], b) \ | |||
dt | + ku * (u_n[i_e, j] + u_n[i_d, j] + u_n[i, j_e] + u_n[i, j_d] - 4 * u_n[i, j]) | ||
v_n1[i, j] = v_n[i, j] + dt * g(u_n[i, j], v_n[i, j], b) \ | |||
+ kv * (v_n[i_e, j] + v_n[i_d, j] + v_n[i, j_e] + v_n[i, j_d] - 4 * v_n[i, j]) | |||
# | # atualizar u_n e v_n | ||
for i in range(Nx): | |||
for j in range(Ny): | |||
u_n[i, j] = u_n1[i, j] | |||
v_n[i, j] = v_n1[i, j] | |||
t += dt | |||
</source> | |||
==Resultados== | |||
Esta seção se dedica ao resultados das simulações realizadas, que mostram o caráter oscilatório do Brusselator ao longo do tempo e também como se dá a difusão dos reagentes em um recipiente em duas dimensões. | |||
As constantes utilizadas foram identificadas abaixo para plotar os gráficos, exceto quando indicado o contrário. | |||
{| class="wikitable" style="margin-left: auto; margin-right: 0px;" style="text-align:center;" | |||
|- style="text-align:left;" | |||
! colspan="6" style="text-align:center; font-weight:bold; font-size:medium;" | Tabela de Constantes e Valores | |||
|- style="font-weight:bold;" | |||
| Símbolo | |||
| Nome | |||
| Valor | |||
|- | |||
| <math>N_x</math> | |||
| style="text-align:left;" | Dimensão analisada ao longo do eixo x | |||
| 50 | |||
|- | |||
| <math>N_y</math> | |||
| style="text-align:left;" | Dimensão analisada ao longo do eixo y | |||
| 50 | |||
|- | |||
| <math>a</math> | |||
| style="text-align:left;" | Constante relativa à concentração do reagente A | |||
| 1 | |||
|- | |||
| <math>b</math> | |||
| style="text-align:left;" | Constante relativa à concentração do reagente B | |||
| 1.7 | |||
|- | |||
| <math>D_u</math> | |||
| style="text-align:left;" | Constante de difusão do reagente U | |||
| 0.5 | |||
|- | |||
| <math>D_v</math> | |||
| style="text-align:left;" | Constante de difusão do reagente V | |||
| 1 | |||
|- | |||
| <math>u_0</math> | |||
| style="text-align:left;" | Concentração de u no tempo inicial (t=0) | |||
| 1 | |||
|- | |||
| <math>v_0</math> | |||
| style="text-align:left;" | Concentração de v no tempo inicial (t=0) | |||
| 1 | |||
|- | |||
| <math>dt</math> | |||
| style="text-align:left;" | Passo de tempo entre iterações | |||
| 0.01 | |||
|- | |||
| <math>ds</math> | |||
| style="text-align:left;" | Unidade de avanço dos eixos no espaço | |||
| 1 | |||
|- | |||
|} | |||
As posições das condições iniciais foram escolhidas arbitrariamente, tendo um total de 6 modos que serão explicados na sequência, sendo eles: | |||
# Condição em formato do sinal "+". | |||
# Condição de borda. | |||
# Condição aleatória. | |||
# Condição de nove pontos centrais. | |||
# Condição da borda completa. | |||
===Simulação=== | |||
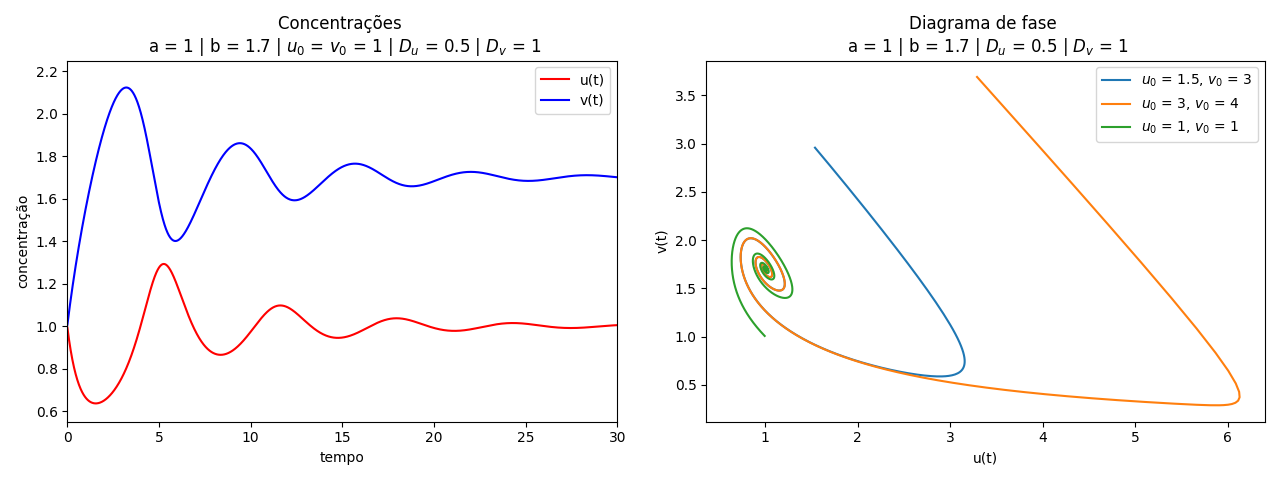
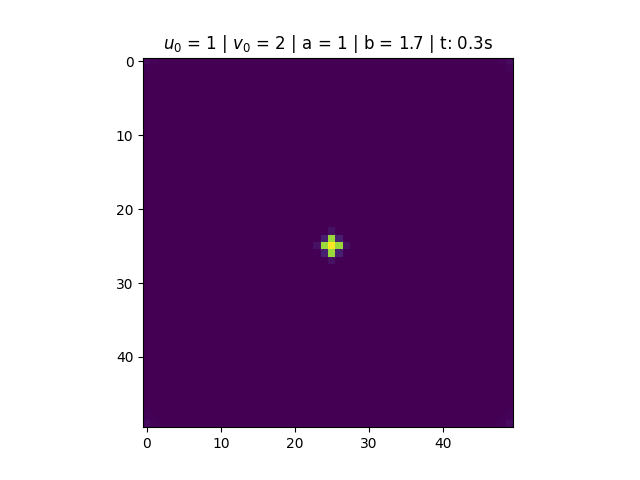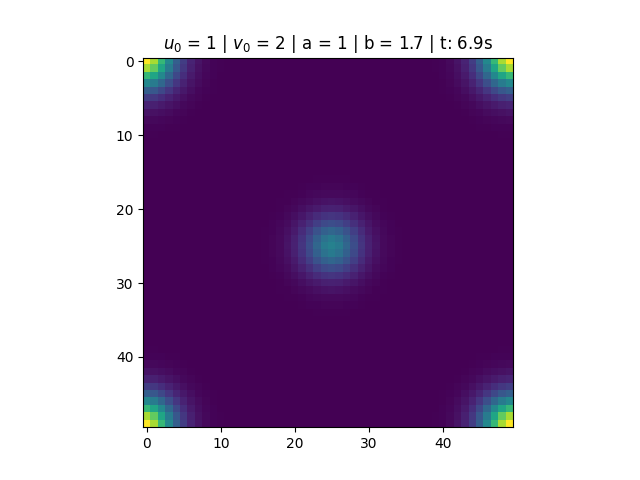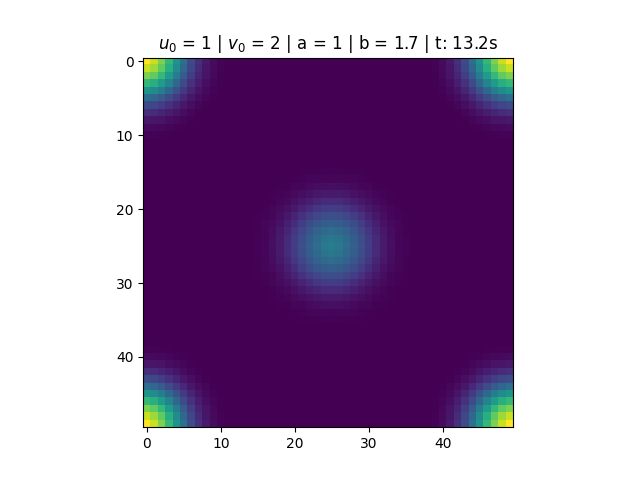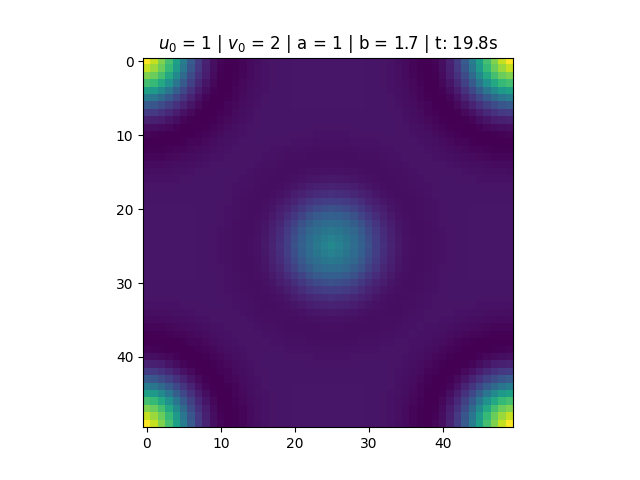
Nas figuras abaixo temos a variação das concentrações dos reagentes ao longo do tempo e os diagramas de fase. Para <math>a = 1</math> e <math>b = 1.7</math> notamos que a solução do sistema converge para <math>u(t) = 1</math> e <math>v(t) = 1.7</math>. Esse resultado está de acordo com o esperado, pois o ponto <math>\left(1, 1.7\right)</math> é um ponto fixo atrator (satisfaz que <math>1-b+a^2 > 0</math>), conforme discutido na seção de análise de estabilidade do Brusselator. Notamos também, pelo diagrama de fase, que mesmo alterando as condições iniciais do problema, a solução converge para o estado estacionário. | |||
[[Arquivo:Sol_bru_b1.png|thumb|center|790px|'''Figura 1''': Solução do Brusselator para a = 1 e b = 1.7.]] | |||
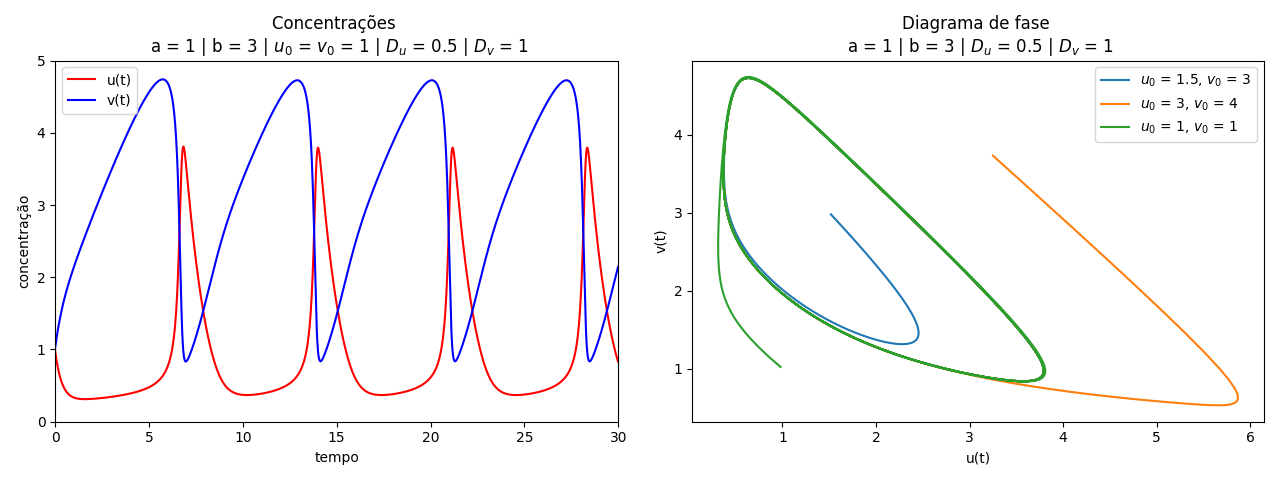
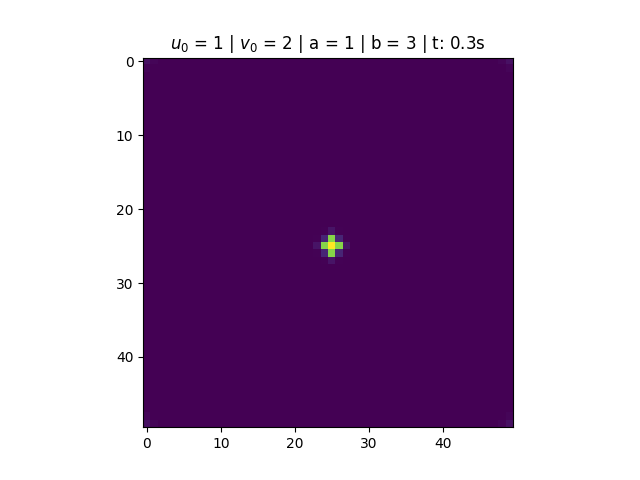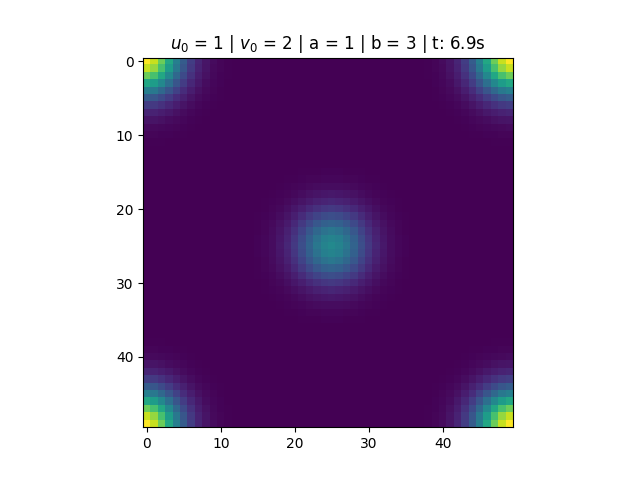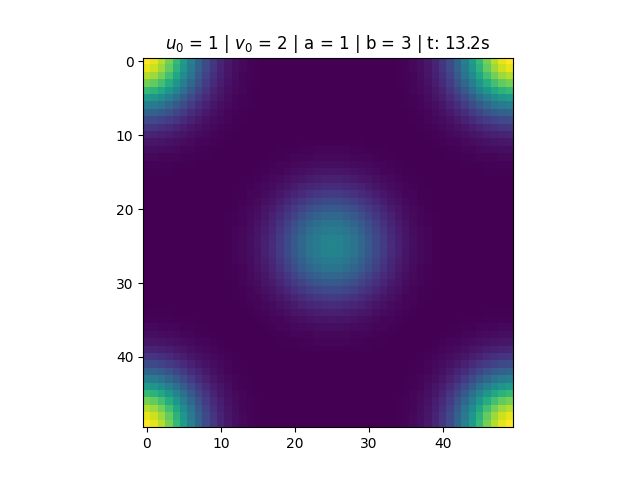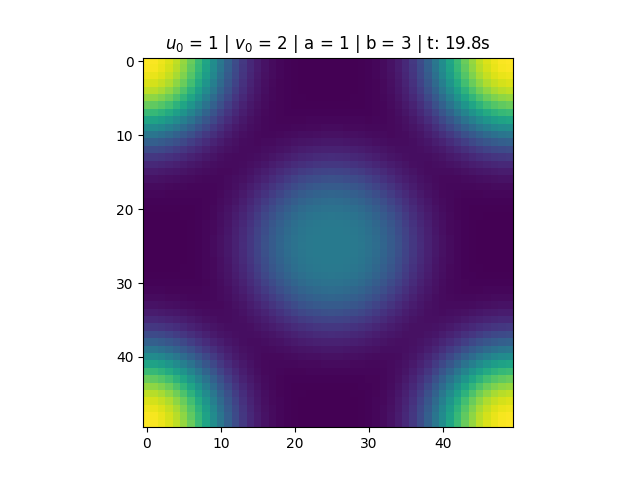
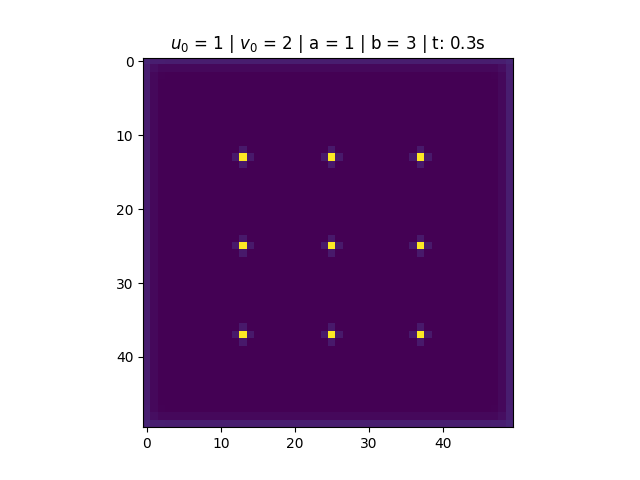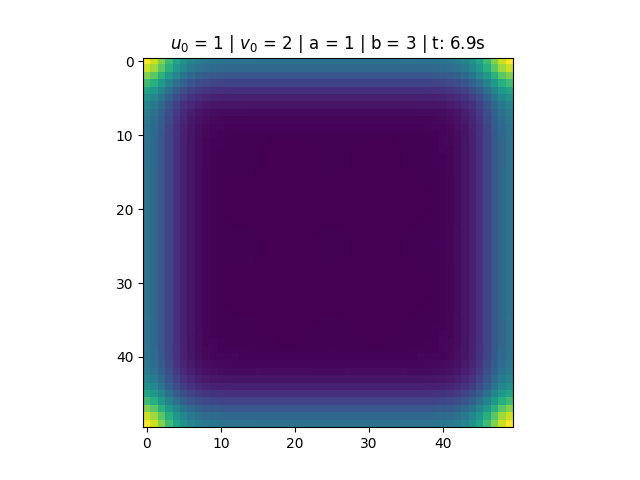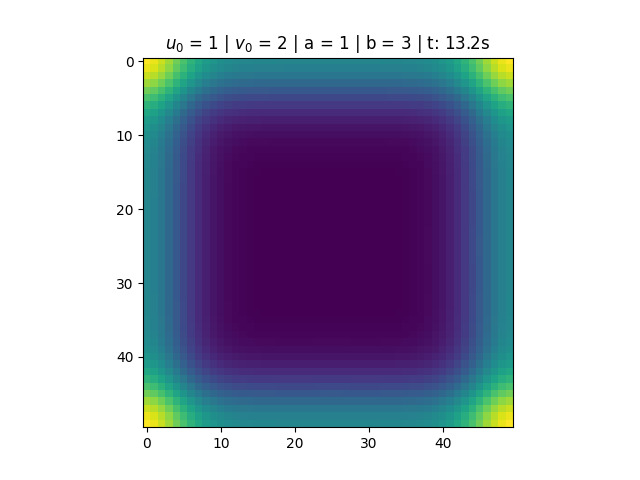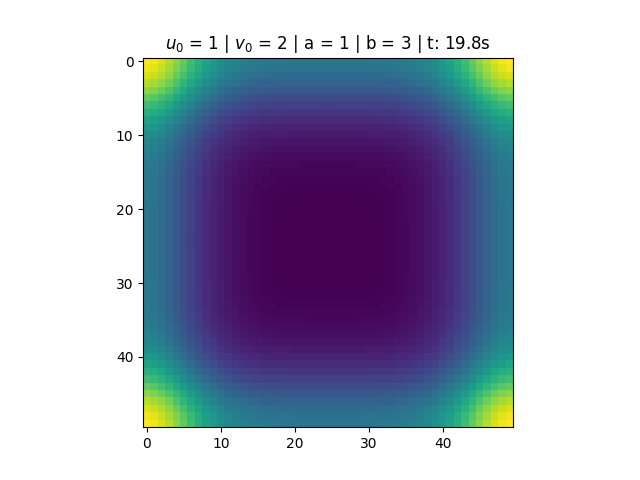
Para <math>a = 1</math> e <math>b = 3</math>, temos que <math>1-b+a^2 < 0</math>, portanto a solução do sistema não converge. O que observamos é um comportamento periódico causado pela existência do ciclo limite. | |||
[[Arquivo:Sol_bru_b3.png|thumb|center|790px|'''Figura 2''': Solução do Brusselator para a = 1 e b = 3.]] | |||
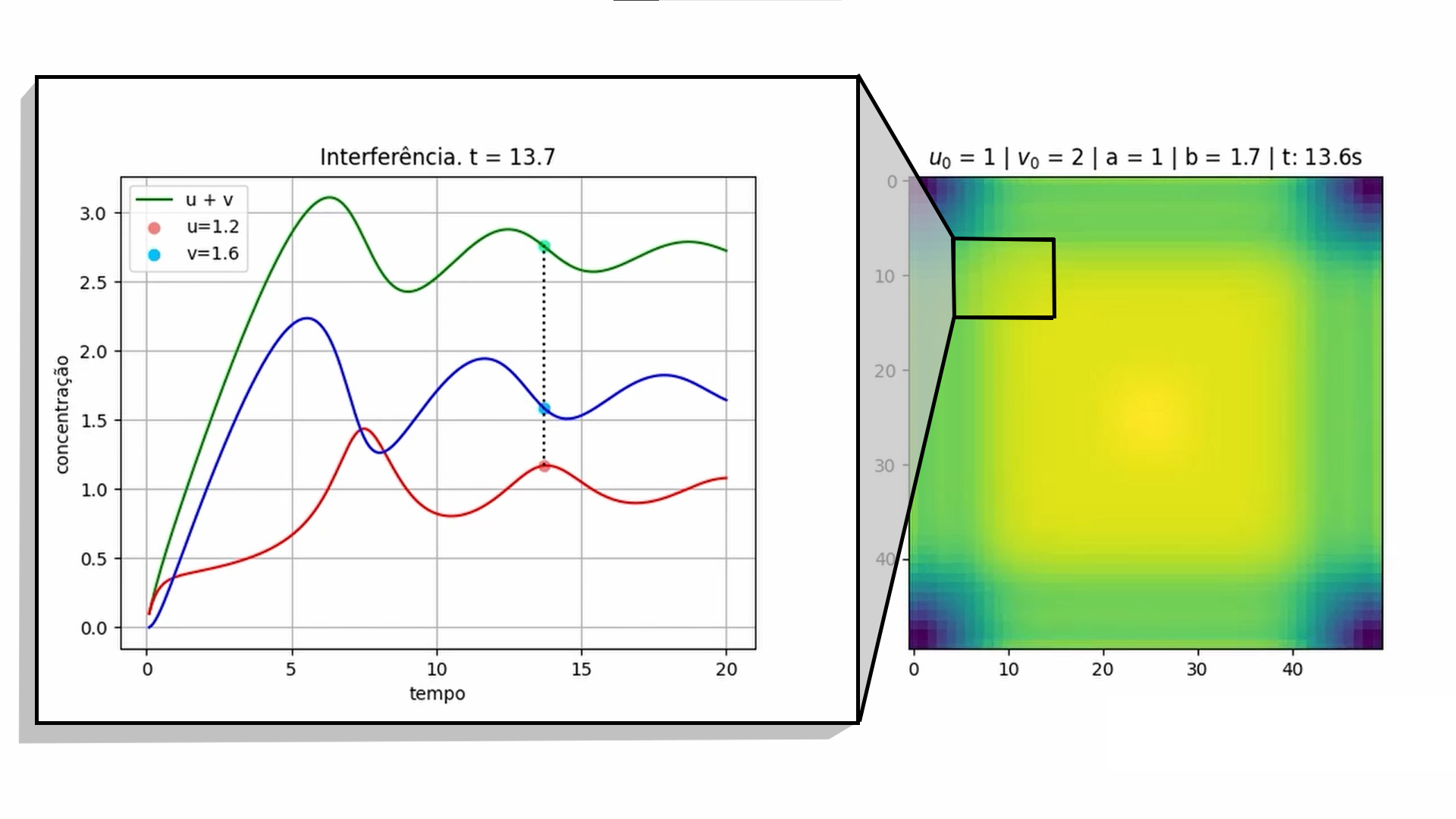
Podemos pensar nas funções de concentração <math>u</math> e <math>v</math> como ondas que sofrem interferência destrutiva, pois estão fora de fase. A animação abaixo mostra este comportamento, observe o reagente <math>U</math>, representado pela cor amarela, no gráfico da direita. Note que quando o ponto do gráfico da esquerda chega ao mínimo local, o valor de <math>U</math> é alto e o de <math>V</math> muito baixo, fazendo com que o gráfico da direita fique muito amarelo. | |||
A | |||
[[Arquivo:AnaliseMid.gif|thumb|center|1300px|'''Figura 3''': Comportamento do reagente.]] | |||
É interessante pensar no gráfico da esquerda como a análise em um ponto específico dentro do recipiente, por exemplo, se <math>Nx=Ny=50</math> estamos analisando o ponto <math>(10,10)</math>. Tendo isso em mente conseguimos analisar os outros 3 cantos da figura, já que, por simetria da posição inicial escolhida, os cantos são iguais. | |||
[[Arquivo:ZoomGraph.jpg|thumb|center|800px|'''Figura 4''': Análise de um ponto.]] | |||
---- | |||
# | ===Condições Iniciais=== | ||
""" | Realizamos as simulações variando espacialmente as condições iniciais, para verificar os diferentes padrões formados pelo sistema. | ||
====Condição em Formato do Sinal "+"==== | |||
O reagente <math>u_0</math> é distribuído no centro formando um sinal de "+". | |||
[[Arquivo:CruzBorda.gif|thumb|right|338px|'''Figura 4''': Comportamento em formato de "+" para o reagente u. Para o reagente v foi utilizado a condição de borda.]] | |||
<source lang="python"> | |||
# u_n [Nx, Ny] | |||
# Centro | |||
u_n[int(Nx / 2), int(Ny / 2)] = u0 | |||
# Lateral do sinal | |||
# X Y | |||
u_n[int(Nx / 2) + 1, int(Ny / 2)] = u0 | |||
u_n[int(Nx / 2) - 1, int(Ny / 2)] = u0 | |||
# Altura do sinal | |||
# X Y | |||
u_n[int(Nx / 2), int(Ny / 2) + 1] = u0 | |||
u_n[int(Nx / 2), int(Ny / 2) - 1] = u0 | |||
</source> | |||
---- | |||
====Condição de borda==== | |||
O reagente <math>v_0</math> foi distribuído nos 4 cantos do recipiente. | |||
[[Arquivo:CruzBordaB3.gif|thumb|right|338px|'''Figura 5''': Analisando u com condição de borda "+". Para o reagente v foi utilizado a condição de borda. Foi utilizado b=3 para mostrar a diferença na simulação.]] | |||
<source lang="python"> | |||
# u_n [Nx, Ny] | |||
# Sup Esq | |||
v_n[0, 0] = v0 | |||
# Sup Dir | |||
v_n[0, Nx - 1] = v0 | |||
# Inf Esq | |||
v_n[Nx - 1, 0] = v0 | |||
# Inf Dir | |||
v_n[Nx - 1, Nx - 1] = v0 | |||
</source> | |||
---- | |||
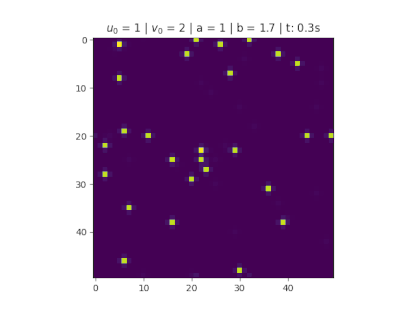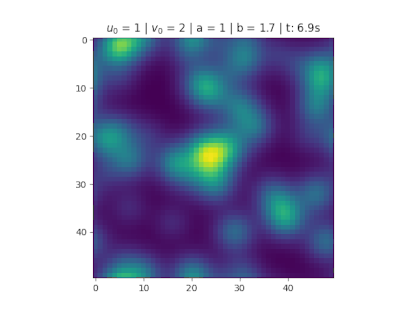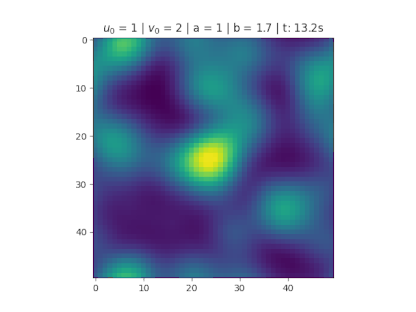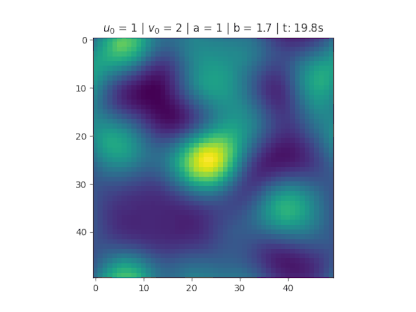
====Condição aleatória==== | |||
Os reagentes são aleatoriamente distribuídos ao longo do recipiente, a quantidade de focos de reagente também é aleatória para <math>u_0</math> e <math>v_0</math>. | |||
[[Arquivo:AleatorioResize.gif|thumb|right|360px|'''Figura 6''': Posição aleatória.]] | |||
<source lang="python"> | |||
from random import * | |||
# u_n [Nx, Ny] | |||
aleatorio = randint(int(Nx / 5) | |||
# Aleatório para u | |||
for _ in range(aleatorio, Nx)): | |||
u_n[randint(0, size), randint(0, size)] = u0 | u_n[randint(0, size), randint(0, size)] = u0 | ||
# | # Aleatório para v | ||
for | for _ in range(aleatorio, Nx)): | ||
v_n[0, | v_n[randint(0, size), randint(0, size)] = v0 | ||
</source> | |||
---- | |||
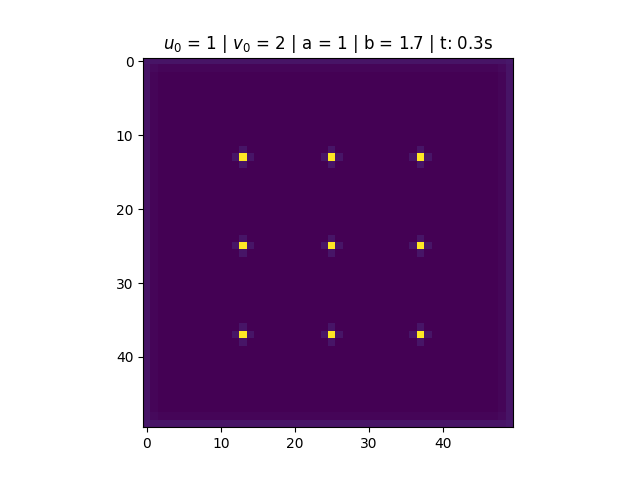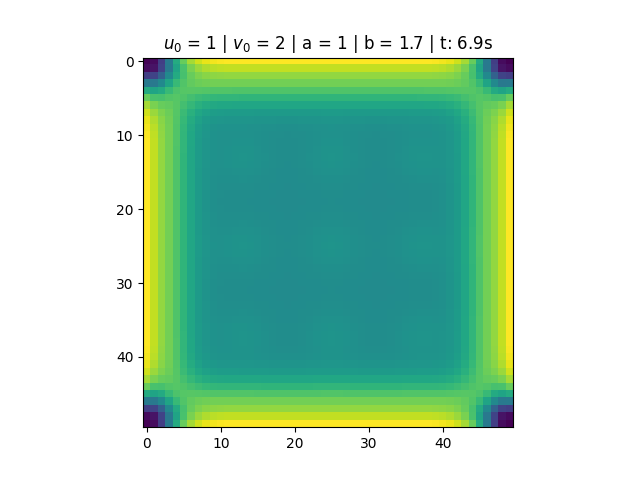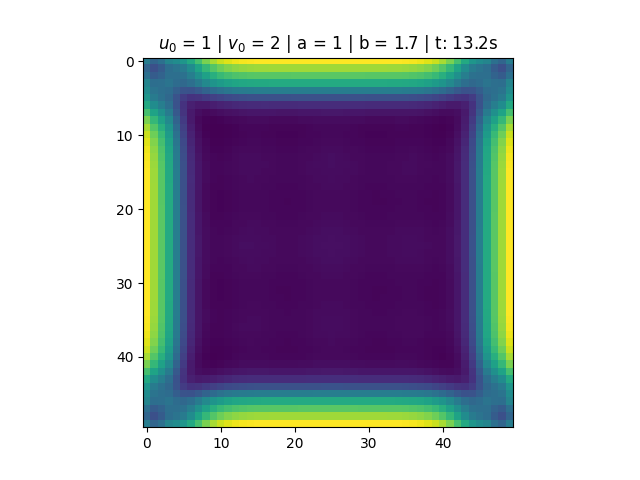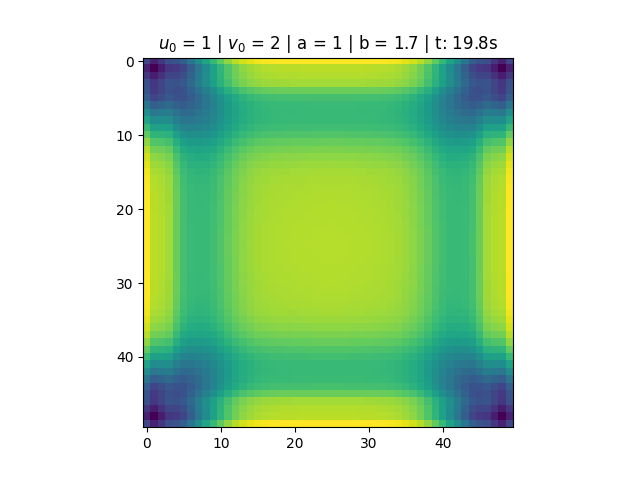
====Condição de nove pontos centrais==== | |||
No centro divide 9 pontos igualmente espaçados de tamanho <math>N_x/4</math>, para <math>u_0</math>. Foi utilizado para <math>u_0</math> na figura 3. | |||
[[Arquivo:9PontosBorda.gif|thumb|right|384px|'''Figura 7''': Utilizando a condição de borda para v.]] | |||
<source lang="python"> | |||
# Nove pontos centrais (u0) | |||
mid = int(Nx / 2); mov = int(Nx / 4) | |||
# Meio & direita | |||
u_n[mid, mid] = u0 | |||
u_n[mid + mov, mid + mov] = u0 | |||
# Meio & esquerda | |||
u_n[mid + mov, mid] = u0 | |||
u_n[mid, mid + mov] = u0 | |||
# Meio, p/baixo & p/lados | |||
u_n[mid + mov, mid - mov] = u0 | |||
u_n[mid - mov, mid - mov] = u0 | |||
# Meio, p/cima & p/lados | |||
u_n[mid - mov, mid] = u0 | |||
u_n[mid, mid - mov] = u0 | |||
u_n[mid - mov, mid + mov] = u0 | |||
</source> | |||
---- | |||
====Condição da borda completa==== | |||
Completa toda a borda do recipiente com reagente <math>v_0</math>. Foi utilizado para <math>v_0</math> na figura 3. | |||
[[Arquivo:9PontosB3.gif|thumb|right|359px|'''Figura 8''': Utilizando a condição de borda para v e b=3.]] | |||
<source lang="python"> | |||
# v_n = [Nx, Ny] | |||
# Toda a borda (v0) | |||
for i in range(Nx): | |||
# | # Completa primeira coluna e primeira linha | ||
v_n[0, i] = v0 | |||
v_n[i, 0] = v0 | |||
# Completa última coluna e última linha | |||
v_n[Nx - 1, i] = v0 | |||
v_n[i, Nx - 1] = v0 | |||
</source> | |||
==Referências== | |||
</ | <references/> | ||
Edição atual tal como às 03h33min de 15 de março de 2022
Grupo: Carolina Lenzi, Eric Naiber e Vitória Xavier
O objetivo deste trabalho é implementar o modelo de reação-difusão Brusselator em duas dimensões, frequentemente utilizado para estudar sistemas complexos químicos e biológicos. O modelo é um sistema não linear de equações diferenciais parciais e foi proposto em 1968 por Ilya Prigogine e seus colaboradores da Universidade Livre de Bruxelas. Desde então tem sido aplicado para analisar reações oscilatórias e autocatalíticas. O método computacional utilizado para implementar o modelo foi o método FTCS (Forward Time Centered Space).
Modelo de Brusselator
O estudo de sistemas químicos e biológicos frequentemente requer o uso de modelos que caracterizam reações de reação-difusão. Um dos modelos mais utilizados é o modelo de Brusselator, que é utilizado para descrever o mecanismo químico de reação-difusão com oscilações não lineares. [1]. Turing observou que quando determinadas reações são associadas a difusão, é possível obter um padrão espacial estável, e isso leva a teoria de morfogênese[2]. Além de processos de reação-difusão, o modelo Brusselator é observado em reações enzimáticas e na física de plasma e de lasers.
O mecanismo de Brusselator proposto por Prigogine[3] é dado por:
- (1.a)
- (1. b)
- (1.c)
- (1.d)
Onde U e V são as espécies químicas de interesse. Assumimos A e B em excesso para que o sistema não atinja o estado de equilíbrio. Esse sistema químico foi importante para o avanço na área de sistemas complexos porque possibilita o uso de modelos matemáticos de duas dimensões, já que U e V são variáveis dependentes, e admite oscilações de ciclo limite[4].
As equações diferenciais parciais associadas com o sistema Brusselator[5] são dadas por:
onde e são as concentrações a serem investigadas em função de tempo e espaço, e são constantes relativas às concentrações dos reagentes A e B, e e são as constantes de difusão de U e V.
A solução analítica do sistema reação-difusão Brusselator ainda não é conhecida e por isso há o interesse de explorá-la numericamente.
Análise da estabilidade do sistema
Análise de ponto crítico
Considerando o sistema livre de difusão, quando :
Encontramos os pontos críticos do sistema igualando as derivadas à zero. Obtemos que o único ponto crítico é . A matriz jacobiana no ponto crítico é dada por
Os autovalores de são os valores que satisfazem a equação caracterísitca
Os autovalores claramente mostram dependência em e no determinante . Esses autovalores governam a estabilidade do ponto crítico e determinam a existência de um ciclo limite. Conforme discutido em Twizell[6], quando o sistema é estável e não existe ciclo limite. Já para o sistema é instável e existe ciclo limite.
Análise de ponto fixo
Ainda em Twizell[6], foi feita a análise de ponto fixo. O único ponto fixo encontrado é . Manipulando a matriz Jacobiana nesse ponto e analisando os autovalores, obtém-se que a solução é atraída para o ponto se . Caso contrário, a solução não converge.
Método FTCS
O FTCS (Forward Time Centered Space) é um método de diferença finita que utiliza a derivada à direita ("para frente") no tempo e a derivada segunda centralizada no espaço para discretizar as variáveis. As derivadas no tempo e no espaço bidimensional ficam:
Substituindo nas equações do Brusselator
onde e são as funções que representam a reação sem difusão.
Utilizamos discretização do tipo
Utilizando a notação , assumindo e rearranjando os termos, reescrevemos as equações como
onde e .
Análise de estabilidade
A análise de estabilidade do método FTCS pode ser feita com a análise local de von Neumann. Para isso precisamos linearizar as equações do modelo Brusselator e escrever as soluções em modos de Fourier, da seguinte forma:
Para que o método seja estável, é preciso que . Após inserir as soluções nas equações linearizadas e realizar manipulações algébricas, como feito por Scholz[7], obtemos
onde
e
Os valores e satisfazem a condição de estabilidade, como mostrado em Scholz[7].
Implementação
A seguir, o trecho do código relativo à implementação do método na linguagem Python. O código completo encontra-se no Github [1]
# u_n, v_n -> concentracao dos reagentes no tempo n (matriz Nx x Ny)
# u_n1, v_n1 -> concentracao dos reagentes no tempo n+1
while t < t_max:
for i in range(Nx):
i_e = (i - 1) % Nx # vizinho a esquerda de 0 é o da ultima posicao
i_d = (i + 1) % Nx # vizinho a direita da ultima posicao é o zero
for j in range(Ny):
j_e = (j - 1) % Ny
j_d = (j + 1) % Ny
# calcular u e v no tempo n+1
u_n1[i, j] = u_n[i, j] + dt * f(u_n[i, j], v_n[i, j], b) \
+ ku * (u_n[i_e, j] + u_n[i_d, j] + u_n[i, j_e] + u_n[i, j_d] - 4 * u_n[i, j])
v_n1[i, j] = v_n[i, j] + dt * g(u_n[i, j], v_n[i, j], b) \
+ kv * (v_n[i_e, j] + v_n[i_d, j] + v_n[i, j_e] + v_n[i, j_d] - 4 * v_n[i, j])
# atualizar u_n e v_n
for i in range(Nx):
for j in range(Ny):
u_n[i, j] = u_n1[i, j]
v_n[i, j] = v_n1[i, j]
t += dt
Resultados
Esta seção se dedica ao resultados das simulações realizadas, que mostram o caráter oscilatório do Brusselator ao longo do tempo e também como se dá a difusão dos reagentes em um recipiente em duas dimensões. As constantes utilizadas foram identificadas abaixo para plotar os gráficos, exceto quando indicado o contrário.
| Tabela de Constantes e Valores | |||||
|---|---|---|---|---|---|
| Símbolo | Nome | Valor | |||
| Dimensão analisada ao longo do eixo x | 50 | ||||
| Dimensão analisada ao longo do eixo y | 50 | ||||
| Constante relativa à concentração do reagente A | 1 | ||||
| Constante relativa à concentração do reagente B | 1.7 | ||||
| Constante de difusão do reagente U | 0.5 | ||||
| Constante de difusão do reagente V | 1 | ||||
| Concentração de u no tempo inicial (t=0) | 1 | ||||
| Concentração de v no tempo inicial (t=0) | 1 | ||||
| Passo de tempo entre iterações | 0.01 | ||||
| Unidade de avanço dos eixos no espaço | 1 | ||||
As posições das condições iniciais foram escolhidas arbitrariamente, tendo um total de 6 modos que serão explicados na sequência, sendo eles:
- Condição em formato do sinal "+".
- Condição de borda.
- Condição aleatória.
- Condição de nove pontos centrais.
- Condição da borda completa.
Simulação
Nas figuras abaixo temos a variação das concentrações dos reagentes ao longo do tempo e os diagramas de fase. Para e notamos que a solução do sistema converge para e . Esse resultado está de acordo com o esperado, pois o ponto é um ponto fixo atrator (satisfaz que ), conforme discutido na seção de análise de estabilidade do Brusselator. Notamos também, pelo diagrama de fase, que mesmo alterando as condições iniciais do problema, a solução converge para o estado estacionário.
Para e , temos que , portanto a solução do sistema não converge. O que observamos é um comportamento periódico causado pela existência do ciclo limite.
Podemos pensar nas funções de concentração e como ondas que sofrem interferência destrutiva, pois estão fora de fase. A animação abaixo mostra este comportamento, observe o reagente , representado pela cor amarela, no gráfico da direita. Note que quando o ponto do gráfico da esquerda chega ao mínimo local, o valor de é alto e o de muito baixo, fazendo com que o gráfico da direita fique muito amarelo.
É interessante pensar no gráfico da esquerda como a análise em um ponto específico dentro do recipiente, por exemplo, se estamos analisando o ponto . Tendo isso em mente conseguimos analisar os outros 3 cantos da figura, já que, por simetria da posição inicial escolhida, os cantos são iguais.
Condições Iniciais
Realizamos as simulações variando espacialmente as condições iniciais, para verificar os diferentes padrões formados pelo sistema.
Condição em Formato do Sinal "+"
O reagente é distribuído no centro formando um sinal de "+".
# u_n [Nx, Ny]
# Centro
u_n[int(Nx / 2), int(Ny / 2)] = u0
# Lateral do sinal
# X Y
u_n[int(Nx / 2) + 1, int(Ny / 2)] = u0
u_n[int(Nx / 2) - 1, int(Ny / 2)] = u0
# Altura do sinal
# X Y
u_n[int(Nx / 2), int(Ny / 2) + 1] = u0
u_n[int(Nx / 2), int(Ny / 2) - 1] = u0
Condição de borda
O reagente foi distribuído nos 4 cantos do recipiente.
# u_n [Nx, Ny]
# Sup Esq
v_n[0, 0] = v0
# Sup Dir
v_n[0, Nx - 1] = v0
# Inf Esq
v_n[Nx - 1, 0] = v0
# Inf Dir
v_n[Nx - 1, Nx - 1] = v0
Condição aleatória
Os reagentes são aleatoriamente distribuídos ao longo do recipiente, a quantidade de focos de reagente também é aleatória para e .
from random import *
# u_n [Nx, Ny]
aleatorio = randint(int(Nx / 5)
# Aleatório para u
for _ in range(aleatorio, Nx)):
u_n[randint(0, size), randint(0, size)] = u0
# Aleatório para v
for _ in range(aleatorio, Nx)):
v_n[randint(0, size), randint(0, size)] = v0
Condição de nove pontos centrais
No centro divide 9 pontos igualmente espaçados de tamanho , para . Foi utilizado para na figura 3.
# Nove pontos centrais (u0)
mid = int(Nx / 2); mov = int(Nx / 4)
# Meio & direita
u_n[mid, mid] = u0
u_n[mid + mov, mid + mov] = u0
# Meio & esquerda
u_n[mid + mov, mid] = u0
u_n[mid, mid + mov] = u0
# Meio, p/baixo & p/lados
u_n[mid + mov, mid - mov] = u0
u_n[mid - mov, mid - mov] = u0
# Meio, p/cima & p/lados
u_n[mid - mov, mid] = u0
u_n[mid, mid - mov] = u0
u_n[mid - mov, mid + mov] = u0
Condição da borda completa
Completa toda a borda do recipiente com reagente . Foi utilizado para na figura 3.
# v_n = [Nx, Ny]
# Toda a borda (v0)
for i in range(Nx):
# Completa primeira coluna e primeira linha
v_n[0, i] = v0
v_n[i, 0] = v0
# Completa última coluna e última linha
v_n[Nx - 1, i] = v0
v_n[i, Nx - 1] = v0
Referências
- ↑ J. Tyson, Some further studies of nonlinear oscillations in chemical systems, J. Chem. Phys. 58 (1973) 3919
- ↑ Turing A. M., The chemical basis of morphogenesis. 1953. Bull. Math. Biol. 52, 153, discussion 119 (1990).
- ↑ I. Prigogine, R. Lefever, Symmetries breaking instabilities in dissipative systems II. J. Phys. Chem. 48, 1695–1700 (1968)
- ↑ R. Lefever and G. Nicolis, Chemical instabilities and sustained oscillations, J. Theor. Biol. 30 (1971)
- ↑ G. Adomian, The diffusion-Brusselator equation. Comput. Math. Appl. 29, 1–3 (1995)
- ↑ 6,0 6,1 Twizell, E. H., Gumel, A. B., & Cao, Q. (1999). Journal of Mathematical Chemistry, 26(4), 297–316
- ↑ 7,0 7,1 Scholz, Christian, Morphology of Experimental and Simulated Turing Patterns, 2009, p. 27-29.















































![{\displaystyle \xi _{\pm }={\frac {1}{2}}\left[\beta \pm {\sqrt {\beta ^{2}-4(\Delta t)^{2}a^{2}b}}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/c31aebfe3a229eeb8f5053e710e7a32cec8b6fd1)