Modelo Brusselator de Reação-Difusão: mudanças entre as edições
Sem resumo de edição |
|||
| Linha 147: | Linha 147: | ||
Durante as simulações em 2D foram utilizados no código: | Durante as simulações em 2D foram utilizados no código: | ||
{| class="wikitable" style="text-align:center;" | {| class="wikitable" style="text-align:center;" | ||
| Linha 166: | Linha 165: | ||
|- | |- | ||
| <math>a</math> | | <math>a</math> | ||
| style="text-align:left;" | Constante relativa à concentração | | style="text-align:left;" | Constante relativa à concentração do reagente A. | ||
| 1 | | 1 | ||
|- | |- | ||
| <math>b</math> | | <math>b</math> | ||
| style="text-align:left;" | Constante relativa à concentração | | style="text-align:left;" | Constante relativa à concentração do reagente B. | ||
| 1.7 | | 1.7 | ||
|- | |||
| <math>D_u</math> | |||
| style="text-align:left;" | Constante de difusão do reagente U. | |||
| 0.1 | |||
|- | |||
| <math>D_v</math> | |||
| style="text-align:left;" | Constante de difusão do reagente V. | |||
| 1 | |||
|- | |- | ||
| <math>u_0</math> | | <math>u_0</math> | ||
| style="text-align:left;" | Concentração no tempo inicial (t=0). | | style="text-align:left;" | Concentração de u no tempo inicial (t=0). | ||
| 1 | | 1 | ||
|- | |- | ||
| <math>v_0</math> | | <math>v_0</math> | ||
| style="text-align:left;" | Concentração no tempo inicial (t=0). | | style="text-align:left;" | Concentração de v no tempo inicial (t=0). | ||
| 2 | | 2 | ||
|- | |- | ||
| <math>dt</math> | | <math>dt</math> | ||
| style="text-align:left;" | Passo entre iterações. | | style="text-align:left;" | Passo de tempo entre iterações. | ||
| 0.1 | | 0.1 | ||
|- | |- | ||
| Linha 189: | Linha 196: | ||
| 1 | | 1 | ||
|- | |- | ||
|} | |} | ||
Constantes que dependem diretamente de outros parâmetros não recebem um valor fixo, como é o caso de <math>k_u</math> e <math>k_v</math> que são constantes de estabilidade. Utilizamos estes valores pois foi utilizado no artigos<ref name='simulated-turing-pattern'>Morphology of Experimental and | Constantes que dependem diretamente de outros parâmetros não recebem um valor fixo, como é o caso de <math>k_u</math> e <math>k_v</math> que são constantes de estabilidade. Utilizamos estes valores pois foi utilizado no artigos<ref name='simulated-turing-pattern'>Morphology of Experimental and | ||
| Linha 259: | Linha 228: | ||
<source lang="python"> | <source lang="python"> | ||
import numpy as np | import numpy as np | ||
# Constantes | # Constantes | ||
| Linha 271: | Linha 234: | ||
a = 1 | a = 1 | ||
b = 1.7 | b = 1.7 | ||
Du = 0.1 | |||
Dv = 1 | |||
t_max = 40 | t_max = 40 | ||
dt = 0.1 | dt = 0.1 | ||
ds = 1 | ds = 1 | ||
ku = Du * dt / (ds ** 2) | ku = Du * dt / (ds ** 2) | ||
kv = Dv * dt / (ds ** 2) | kv = Dv * dt / (ds ** 2) | ||
# Valores iniciais | |||
u0 = 1 | |||
v0 = 2 | |||
t = 0 | |||
def f(u, v): | def f(u, v): | ||
return a - (b + 1) * u + u * u * v | return a - (b + 1) * u + u * u * v | ||
def g(u, v): | def g(u, v): | ||
return b * u - u * u * v | return b * u - u * u * v | ||
| Linha 306: | Linha 266: | ||
u_n1 = np.zeros((Nx, Ny)) | u_n1 = np.zeros((Nx, Ny)) | ||
v_n1 = np.zeros((Nx, Ny)) | v_n1 = np.zeros((Nx, Ny)) | ||
# Condições iniciais | |||
# Nove pontos centrais (u0) | # Nove pontos centrais (u0) | ||
| Linha 320: | Linha 283: | ||
v_n[i, Nx - 1] = v0 | v_n[i, Nx - 1] = v0 | ||
# | # FTCS | ||
while t < t_max: | while t < t_max: | ||
for i in range(Nx): | for i in range(Nx): | ||
| Linha 339: | Linha 298: | ||
v_n1[i, j] = v_n[i, j] + dt * g(u_n[i, j], v_n[i, j]) \ | v_n1[i, j] = v_n[i, j] + dt * g(u_n[i, j], v_n[i, j]) \ | ||
+ kv * (v_n[i_e, j] + v_n[i_d, j] + v_n[i, j_e] + v_n[i, j_d] - 4 * v_n[i, j]) | + kv * (v_n[i_e, j] + v_n[i_d, j] + v_n[i, j_e] + v_n[i, j_d] - 4 * v_n[i, j]) | ||
# atualizar u_n e v_n | # atualizar u_n e v_n | ||
| Linha 350: | Linha 306: | ||
t += dt | t += dt | ||
</source> | </source> | ||
Edição das 15h06min de 11 de março de 2022
Grupo: Carolina Lenzi, Eric Naiber e Vitória Xavier
O objetivo deste trabalho é implementar o modelo de reação-difusão Brusselator em duas dimensões, frequentemente utilizado para estudar sistemas complexos químicos e biológicos. O modelo é um sistema não linear de equações diferenciais parciais e foi proposto em 1970 por Ilya Prigogine e seus colaboradores da Universidade Livre de Bruxelas. Desde então tem sido aplicado para analisar reações oscilatórias e autocatalíticas. O método computacional utilizado para implementar o modelo foi o método FTCS (Forward Time Centered Space).
Modelo de Brusselator
O estudo de sistemas químicos e biológicos frequentemente requer o uso de modelos que caracterizam reações de reação-difusão. Um dos modelos mais utilizados é o modelo de Brusselator, que é utilizado para descrever o mecanismo químico de reação-difusão com oscilações não lineares. [J. Tyson, Some further studies of nonlinear oscillations in chemical systems, J. Chem. Phys. 58 (1973) 3919.] Turing [ref?] observou que quando determinadas reações são associadas a difusão, é possível obter um padrão espacial estável, e isso leva a teoria de morfogênese. Além de processos de reação-difusão, o modelo Brusselator é observado em reações enzimáticas e na física de plasma e de lasers.
O mecanismo de Brusselator proposto por Prigogine (1970) é dado por [I. Prigogine, R. Lefever, Symmetries breaking instabilities in dissipative systems II. J. Phys. Chem. 48, 1695–1700 (1968)]:
- (1.a)
- (1. b)
- (1.c)
- (1.d)
Onde U e V são as espécies químicas de interesse. Assumimos A e B em excesso para que o sistema não atinja o equilíbrio. Esse sistema químico foi importante para o avanço na área de sistemas complexos porque possibilita o uso de modelos matemáticos de duas dimensões, já que U e V são variáveis dependentes, e admite “limit-cycle oscilations”. [ R. Lefever and G. Nicolis, Chemical instabilities and sustained oscillations, J. Theor. Biol. 30 (1971) 267.].
As equações diferenciais parciais associadas com o sistema Brusselator são dadas por(G. Adomian, The diffusion-Brusselator equation. Comput. Math. Appl. 29, 1–3 (1995)):
onde e são as concentrações a serem investigadas em função de tempo e espaço, e são constantes relativas às concentrações dos reagentes A e B, e e constantes de difusão.
A solução analítica do sistema reação-difusão Brusselator ainda não é conhecida e por isso há o interesse de explorá-la numericamente.
Análise da estabilidade do sistema
Análise de ponto crítico
Considerando o sistema livre de difusão, quando :
Onde e e e são constantes positivas e reais. A matriz jacobiana no ponto crítico é dada por
Os autovalores de são os valores que satisfazem a equação caracterísitca
Os autovalores claramente mostram dependência em e no determinante . Esses autovalores governam a estabilidade do ponto crítico ou determinam a existência de um ciclo limite. As propriedades de estabilidade ou a existência de um ciclo limite estão sumarizadas na tabela abaixo, em relação a figura 1.
[[Arquivo:]]
Utilizando a teoria Hopf, é mostrado que o ponto crítico perde sua estabilidade quando A e B movem da região 2 para região 3, na figura 1, atravessando a curva . Uma bifurcação Hopf ocorre quando ao passo que essa curva é atravessada e um ciclo limite estável existe para A e B nas regiões 1 e 2, mas não para A e B nas regiões 3 e 4.
Conclui-se que a curva governa a estabilidade do sistema.
Análise de ponto fixo
O estado estacionário do sistema pode ser encontrado igualando o coeficiente de difusão, e portanto as derivadas parciais, a zero. Percebe-se que esse sistema converge para os pontos fixos:
Em [twizell] a manipulação da matriz jacobiana nos pontos fixos resulta nos seguintes autovalores
Onde fica claro que os denominadores dos autovalores são sempre positivos quando and . As inequações
- e
São verdadeiras sempre que . Portanto, uma condição suficiente para o ponto fixo atrair a sequência gerada pelo sistema é .
Ainda em [Twizell] o modelo reação-difusão Brusselator foi discretizado e a análise dos pontos fixos concluiu que o sistema converge para e , sendo esse o único estado estacionário do sistema.
Concluiu que também que o sistema apresenta estado oscilatório quando
Estado em que o sistema não converge para nenhum ponto.
Método FTCS
O FTCS (Forward Time Centered Space) é um método de diferença finita que utiliza a derivada à direita ("para frente") no tempo e a derivada segunda centralizada no espaço para discretizar as variáveis. As derivadas no tempo e no espaço bidimensional ficam:
Substituindo nas equações do Brusselator
onde e são as funções que representam a reação sem difusão.
Utilizamos discretização do tipo
Utilizando a notação , assumindo e rearranjando os termos, reescrevemos as equações como
onde e .
Análise de estabilidade do método
Resultados
Esta seção se dedica ao resultados da simulação, utilizando valores de constantes de outros artigos como o [Nome do artigo e referência]. As constantes utilizadas foram identificadas na próxima subseção, com exceção nos gráficos em que indicamos outros valores. As simulações mostram o caráter oscilatório do Brusselator ao longo do tempo e também como se da a reação e difusão em um recipiente em duas dimensões. O código utilizado (incompleto) estará logo após a seção de resultados.
Definindo Constantes
Durante as simulações em 2D foram utilizados no código:
| Tabela de Constantes e Valores | |||||
|---|---|---|---|---|---|
| Símbolo | Nome | Valor | |||
| Dimensão analisada ao longo do eixo x. | 50 | ||||
| Dimensão analisada ao longo do eixo y. | 50 | ||||
| Constante relativa à concentração do reagente A. | 1 | ||||
| Constante relativa à concentração do reagente B. | 1.7 | ||||
| Constante de difusão do reagente U. | 0.1 | ||||
| Constante de difusão do reagente V. | 1 | ||||
| Concentração de u no tempo inicial (t=0). | 1 | ||||
| Concentração de v no tempo inicial (t=0). | 2 | ||||
| Passo de tempo entre iterações. | 0.1 | ||||
| Unidade de avanço dos eixos no espaço. | 1 | ||||
Constantes que dependem diretamente de outros parâmetros não recebem um valor fixo, como é o caso de e que são constantes de estabilidade. Utilizamos estes valores pois foi utilizado no artigos[1]. Já as posições das condições iniciais foram escolhidas arbitrariamente, tendo um total de 6 modos (dentro do código completo[1]), sendo eles:
- Condição em formato do sinal "+"; o reagente é distribuído no centro, formando um sinal de "+", para .
- Condição de borda; onde os há reagente nos 4 cantos do recipiente para .
- Condição aleatória; coloca uma quantia aleatória de pontos em qualquer posição, para e .
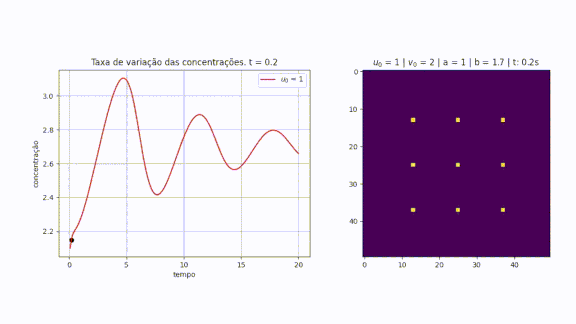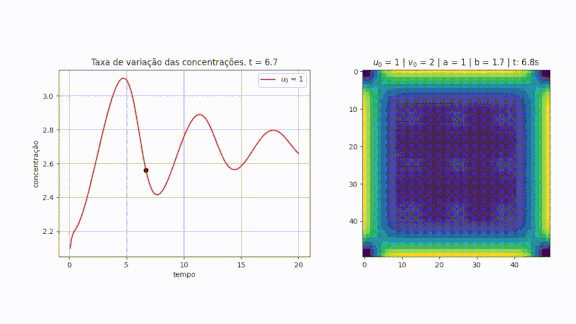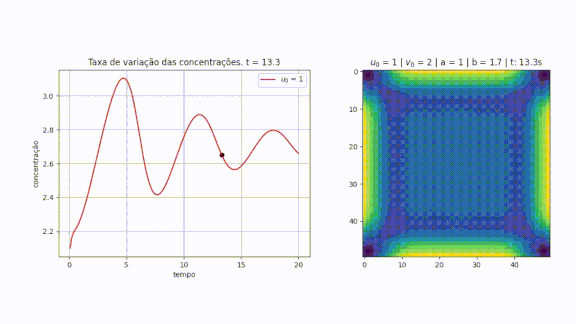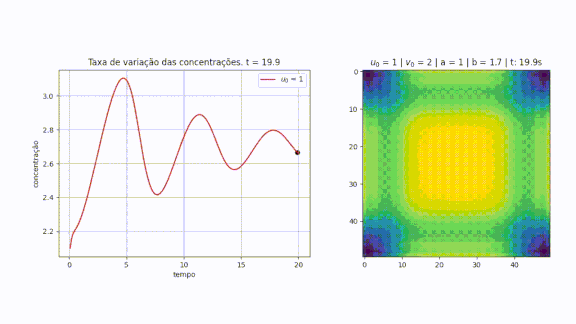
- Condição de nove pontos centrais; no centro divide 9 pontos com espaçamento de , para .
- Condição da borda completa; completa toda a borda do recipiente com reagente .
Simulação
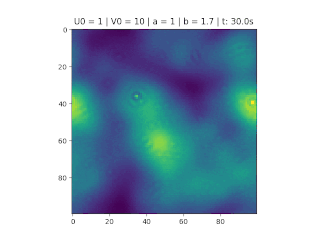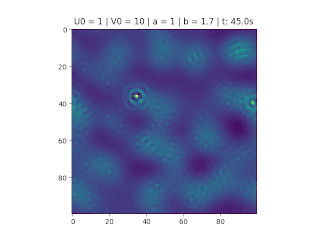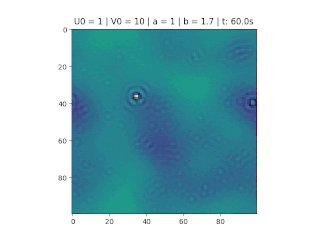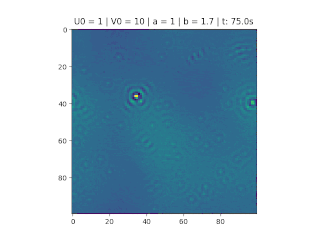
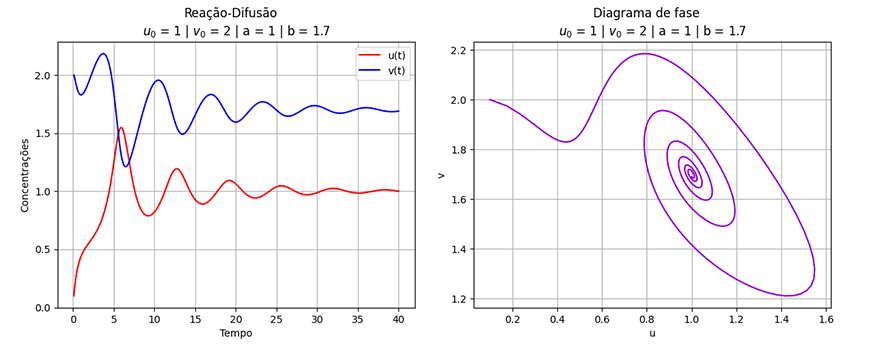
No gráfico abaixo podemos notar o caráter ondulatório da variação da concentração ao longo do tempo, as linhas (azul e vermelho) significam a taxa de concentração no recipiente.
Note que quando diminui, aumenta rapidamente, mantendo-se um ciclo oscilatório em que a amplitude da onda de e vão diminuindo ao longo do tempo, como mostrado nas figuras.
Pensando no gráfico da reação-difusão como ondas, é correto afirmar que ambas juntas formam um padrão de interferência destrutivo. A animação abaixo mostra exatamente este comportamento, observando o reagente no gráfico da direita. Note que quando o ponto do gráfico da esquerda chega ao mínimo local, o valor de é alto e o de muito baixo, fazendo com que o gráfico da direita fique muito amarelo (amarelo significa uma alta concentração de sendo formada).
Condições Iniciais
Para observar melhor os resultados, foram feitas diversas condições iniciais que foram separadas em tópicos citados em Definindo Constantes.
Implementação
O método foi implementado em Python, considerando , variando as constantes e e as condições iniciais do problema.
- Código completo no GitHub[2]
import numpy as np
# Constantes
Nx = Ny = 25
a = 1
b = 1.7
Du = 0.1
Dv = 1
t_max = 40
dt = 0.1
ds = 1
ku = Du * dt / (ds ** 2)
kv = Dv * dt / (ds ** 2)
# Valores iniciais
u0 = 1
v0 = 2
t = 0
def f(u, v):
return a - (b + 1) * u + u * u * v
def g(u, v):
return b * u - u * u * v
# vetores no tempo n
u_n = np.zeros((Nx, Ny))
v_n = np.zeros((Nx, Ny))
# vetores no tempo n+1
u_n1 = np.zeros((Nx, Ny))
v_n1 = np.zeros((Nx, Ny))
# Condições iniciais
# Nove pontos centrais (u0)
mid = int(Nx / 2) # Centro
mov = int(Nx / 4) # Movendo para cima e para os lados
u_n[mid, mid] = u_n[mid + mov, mid + mov] = u_n[mid + mov, mid] = u_n[mid, mid + mov] = u_n[mid + mov, mid - mov] = u0
u_n[mid - mov, mid - mov] = u_n[mid - mov, mid] = u_n[mid, mid - mov] = u_n[mid - mov, mid + mov] = u0
# Toda a borda (v0)
for i in range(Nx):
v_n[0, i] = v0
v_n[i, 0] = v0
v_n[Nx - 1, i] = v0
v_n[i, Nx - 1] = v0
# FTCS
while t < t_max:
for i in range(Nx):
i_e = (i - 1) % Nx # vizinho a esquerda de 0 é o da ultima posicao
i_d = (i + 1) % Nx # vizinho a direita da ultima posicao é o zero
for j in range(Ny):
j_e = (j - 1) % Ny
j_d = (j + 1) % Ny
u_n1[i, j] = u_n[i, j] + dt * f(u_n[i, j], v_n[i, j]) \
+ ku * (u_n[i_e, j] + u_n[i_d, j] + u_n[i, j_e] + u_n[i, j_d] - 4 * u_n[i, j])
v_n1[i, j] = v_n[i, j] + dt * g(u_n[i, j], v_n[i, j]) \
+ kv * (v_n[i_e, j] + v_n[i_d, j] + v_n[i, j_e] + v_n[i, j_d] - 4 * v_n[i, j])
# atualizar u_n e v_n
for i in range(Nx):
for j in range(Ny):
u_n[i, j] = u_n1[i, j]
v_n[i, j] = v_n1[i, j]
t += dt
Referências
- ↑ Morphology of Experimental and Simulated Turing Patterns, Christian Scholz, August 2009, Pgs 27-29