|
|
| Linha 133: |
Linha 133: |
| ==Código== | | ==Código== |
|
| |
|
| <source lang=sh>
| | Adicionar programas usados |
| from scipy import *
| |
| import sys
| |
| import numpy as np
| |
| | |
| #Rede aleatória para Ising 2D
| |
| def RandomL(N):
| |
| latt=zeros((N,N),dtype=int)
| |
| for i in range(N):
| |
| for j in range(N):
| |
| latt[i,j]=sign(2*rand()-1)
| |
| return latt
| |
|
| |
| #Energia da rede
| |
| def CEnergy(latt):
| |
| Ene=0
| |
| for i in range(N):
| |
| for j in range(N):
| |
| S=latt[i,j]
| |
| WF=latt[(i+1)%N,j]+latt[i,(j+1)%N]+latt[(i-1)%N,j]+latt[i,(j-1)%N]
| |
| Ene+=-WF*S
| |
| return int(Ene/2.)
| |
|
| |
| #Quantidades termodinâmicas usando a densidade de estados
| |
| def Thermod(T,lngE,Energies,E0):
| |
| Z=0
| |
| Ev=0
| |
| E2v=0
| |
| for i,E in enumerate(Energies):
| |
| #w=exp(lngE[i]-lngE[0]-(E+E0)/T)
| |
| w=exp(lngE[i]-E/T)
| |
| Z+=w
| |
| Ev+=w*E
| |
| E2v+=w*E**2
| |
| #print(i, E, lngE[i], w, Z, Ev)
| |
| Ev*=1./Z
| |
| cv=(E2v/Z-Ev**2)/T**2
| |
| F = -T*np.log(Z)
| |
| S = (Ev - F)/T
| |
| return (Ev/N2,cv/N2,F/N2,S/N2)
| |
|
| |
| #Algoritmo de Wang-Landau
| |
| def WangLandau(Nitt,N,N2,indE,E0,flatness):
| |
| latt=RandomL(N)
| |
| Ene=CEnergy(latt)
| |
| lngE=zeros(len(Energies),dtype=float)
| |
| Hist=zeros(len(Energies),dtype=float)
| |
| lnf=1.0
| |
| itt = 0
| |
| while exp(lnf) > fmin:
| |
| itt = itt + 1
| |
| ii=int(rand()*N2)
| |
| (i,j)=(ii%N,ii/N)
| |
| i=int(rand()*N)
| |
| j=int(rand()*N)
| |
| S=latt[i,j]
| |
| WF=latt[(i+1)%N,j]+latt[i,(j+1)%N]+latt[(i-1)%N,j]+latt[i,(j-1)%N]
| |
| Enew=Ene+2*S*WF
| |
| P=exp(lngE[indE[Ene+E0]]-lngE[indE[Enew+E0]])
| |
| #P=exp(lngE[indE[Ene]]-lngE[indE[Enew]])
| |
| if P>rand():
| |
| latt[i,j]=-S
| |
| Ene=Enew
| |
| Hist[indE[Ene+E0]]+=1.
| |
| #Hist[indE[Ene+E0]]+=1.
| |
| lngE[indE[Ene+E0]]+=lnf
| |
| #lngE[indE[Ene]]+=lnf
| |
| if itt%100==0:
| |
| aH=sum(Hist)/(N2+0.0)
| |
| mH=min(Hist)
| |
| if mH>aH*flatness:
| |
| Hist=zeros(len(Hist))
| |
| lnf/=2.
| |
| print("iteracao =", itt, 'f=', exp(lnf))
| |
| return lngE,Hist
| |
|
| |
| from scipy import *
| |
| import sys
| |
| from pylab import *
| |
| from matplotlib import pyplot as plt
| |
| import numpy as np
| |
| | |
| Nitt=10000000 #remover essa parte, não é mais usado
| |
| fmin = 1.00000001
| |
| print(fmin)
| |
| N=16
| |
| flatness=0.8
| |
| N2=N*N
| |
| | |
| # energias possiveis
| |
| Energies = (4*arange(N2+1)-2*N2).tolist()
| |
| print(Energies)
| |
| Energies.pop(1)
| |
| Energies.pop(-2)
| |
| E0=Energies[-1]
| |
| print(Energies)
| |
| indE=-ones(E0*2+1,dtype=int)
| |
| | |
| for i,E in enumerate(Energies):
| |
| indE[E+E0]=i
| |
| | |
|
| |
| lngE=np.load('lngE.npy')
| |
| Hist=np.load('Hist.npy')
| |
| | |
| Hist *= len(Hist)/sum(Hist)
| |
| | |
| len(lngE),len(Hist)
| |
| | |
| from pylab import *
| |
| EE=array(Energies);EE=EE/(N*N)
| |
| plot(EE,lngE,'o',markersize=1.5,label='Simulação')
| |
| #plot(EE,Hist,'-s',markersize=2.0,label='Histogram')
| |
| xlabel(r'$E/N$')
| |
| ylabel(r'$ln[g(E)]$')
| |
| legend(loc='best')
| |
| show()
| |
| | |
| f=open('bealeL16.dat','r');f=f.readlines()
| |
| | |
| beale=[]
| |
| for i in range(len(f)):
| |
| beale.append(float(f[i]))
| |
| | |
| plot(Energies,beale,label='Exata')
| |
| xlabel('Energia')
| |
| ylabel('ln[g(E)]')
| |
| legend(loc='best')
| |
| show()
| |
| | |
| plot(EE,beale,label='Exata')
| |
| plot(EE,lngE,'o',markersize=1.5,label='Simulação')
| |
| xlabel(r'$E/N$')
| |
| ylabel(r'$ln[g(E)]$')
| |
| legend(loc='best')
| |
| show()
| |
| | |
| #Erro relativo
| |
| | |
| erro=[]
| |
| for i in range(len(lngE)):
| |
| erro.append((abs(lngE[i]-beale[i]))/beale[i])
| |
| | |
| plot(Energies,erro)
| |
| xlabel('Energia')
| |
| ylabel(r'$\epsilon [ln(g)]$')
| |
| #legend(loc='best')
| |
| yscale('log')
| |
| show()
| |
| | |
| from mpl_toolkits.axes_grid.inset_locator import (inset_axes,InsetPosition,mark_inset)
| |
| | |
| fig,ax1=subplots()
| |
| ax1.plot(EE,lngE,'o',markersize=1.5,label='Simulação')
| |
| ax1.plot(EE,beale,label='Exata')
| |
| ax1.set_xlabel('E/N')
| |
| ax1.set_ylabel(r'$ln[g(E)]$')
| |
| ax1.set_xlim(-2.,4.)
| |
| ax1.set_ylim(0.,200.)
| |
| ax1.legend(loc='best')
| |
| ax2=axes([0,0,1,1])
| |
| ip=InsetPosition(ax1,[0.685,0.5,0.3,0.3])
| |
| ax2.set_axes_locator(ip)
| |
| ax2.plot(EE,erro)
| |
| ax2.set_yscale('log')
| |
| ax2.set_xlabel('E/N')
| |
| ax2.set_ylabel(r'$\epsilon [ln(g)]$')
| |
| savefig('fig1.png')
| |
| show()
| |
| | |
| print(beale[0:5])
| |
| | |
| print(lngE[0:5])
| |
| | |
| lmb23 = -1**300
| |
| lmb23e = -1**300
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo23 = lngE[i] - Energies[i]/2.3
| |
| if termo23 > lmb23:
| |
| lmb23 = termo23
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo23e = beale[i] - Energies[i]/2.3
| |
| if termo23e > lmb23e:
| |
| lmb23e = termo23e
| |
| i = i+1
| |
| | |
| print(lmb23,lmb23e)
| |
| | |
| lmb22 = -1**300
| |
| lmb22e = -1**300
| |
| lmb24 = -1**300
| |
| lmb24e = -1**300
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo22 = lngE[i] - Energies[i]/2.2
| |
| if termo22 > lmb22:
| |
| lmb22 = termo22
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo22e = beale[i] - Energies[i]/2.2
| |
| if termo22e > lmb22e:
| |
| lmb22e = termo22e
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo24 = lngE[i] - Energies[i]/2.4
| |
| if termo24 > lmb24:
| |
| lmb24 = termo24
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo24e = beale[i] - Energies[i]/2.4
| |
| if termo24e > lmb24e:
| |
| lmb24e = termo24e
| |
| i = i+1
| |
| | |
| print(lmb22,lmb22e,lmb24,lmb24e)
| |
| | |
| E=array(Energies);E1=E/N2
| |
| P=exp(lngE-E/2.3 - lmb23)
| |
| Pexact=exp(beale-E/2.3 - lmb23e)
| |
| | |
| | |
| plot(E1,P,label=r'$k_B T_c = 2.3$')
| |
| plot(E1,Pexact,label=r'$k_B T_c = 2.3$ exata')
| |
| #plot(Energies,P2,label=r'$k_B T_c = 2.2$')
| |
| #plot(Energies,P3,label=r'$k_B T_c = 2.4$')
| |
| xlabel('E/N')
| |
| ylabel(r'$g(E)e^{-E/k_B T_c}$')
| |
| legend(loc='best')
| |
| show()
| |
| | |
| P2=exp(lngE-E/2.2-lmb22)
| |
| P2exact=exp(beale-E/2.2-lmb22e)
| |
| P3=exp(lngE-E/2.4-lmb24)
| |
| P3exact=exp(beale-E/2.4-lmb24e)
| |
| | |
| fig,ax1=subplots()
| |
| ax1.plot(E1,P,label=r'$k_B T_c = 2.3$')
| |
| #ax1.plot(E1,Pexact,label=r'$k_B T_c = 2.3$ exata')
| |
| ax1.set_xlabel('E/N')
| |
| ax1.set_ylabel(r'$g(E)e^{-E/k_B T_c}$')
| |
| #ax1.set_ylim(0.,200.)
| |
| ax1.legend(loc='best')
| |
| ax2=axes([0,0,1,1])
| |
| ip=InsetPosition(ax1,[0.5,0.3,0.5,0.5])
| |
| ax2.set_axes_locator(ip)
| |
| ax2.plot(E1,P2,label=r'$k_B T = 2.2$')
| |
| #ax2.plot(E1,P2exact,label=r'$k_B T = 2.2$ exata')
| |
| ax2.plot(E1,P3,label=r'$k_B T = 2.4$')
| |
| #ax2.plot(E1,P3exact,label=r'$k_B T = 2.4$ exata')
| |
| #ax2.set_yscale('log')
| |
| ax2.set_xlabel('E/N')
| |
| ax2.set_ylabel(r'$g(E)e^{-E/k_B T}$')
| |
| ax2.legend(loc='best')
| |
| savefig('fig2.png')
| |
| show()
| |
| | |
| E=array(Energies);E1=E/N2
| |
| P=exp(lngE-E/2.3 - lmb23)
| |
| Pexact=exp(beale-E/2.3 - lmb23e)
| |
| | |
| | |
| plot(E1,P,label=r'$k_B T_c = 2.3$')
| |
| plot(E1,Pexact,label=r'$k_B T_c = 2.3$ exata')
| |
| #plot(Energies,P2,label=r'$k_B T_c = 2.2$')
| |
| #plot(Energies,P3,label=r'$k_B T_c = 2.4$')
| |
| xlabel('E/N')
| |
| ylabel(r'$g(E)e^{-E/k_B T_c}$')
| |
| legend(loc='best')
| |
| show()
| |
| | |
| lmb22 = -1**300
| |
| lmb22e = -1**300
| |
| lmb24 = -1**300
| |
| lmb24e = -1**300
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo22 = lngE[i] - Energies[i]/2.2
| |
| if termo22 > lmb22:
| |
| lmb22 = termo22
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo22e = beale[i] - Energies[i]/2.2
| |
| if termo22e > lmb22e:
| |
| lmb22e = termo22e
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo24 = lngE[i] - Energies[i]/2.4
| |
| if termo24 > lmb24:
| |
| lmb24 = termo24
| |
| i = i+1
| |
| | |
| i=0
| |
| while i<N2-1:
| |
| termo24e = beale[i] - Energies[i]/2.4
| |
| if termo24e > lmb24e:
| |
| lmb24e = termo24e
| |
| i = i+1
| |
| | |
| print(lmb22,lmb22e,lmb24,lmb24e)
| |
| | |
| P2=exp(lngE-E/2.2-lmb22)
| |
| P2exact=exp(beale-E/2.2-lmb22e)
| |
| P3=exp(lngE-E/2.4-lmb24)
| |
| P3exact=exp(beale-E/2.4-lmb24e)
| |
| | |
| fig,ax1=subplots()
| |
| ax1.plot(E1,P,label=r'$k_B T_c = 2.3$')
| |
| #ax1.plot(E1,Pexact,label=r'$k_B T_c = 2.3$ exata')
| |
| ax1.set_xlabel('E/N')
| |
| ax1.set_ylabel(r'$g(E)e^{-E/k_B T_c}$')
| |
| #ax1.set_ylim(0.,200.)
| |
| ax1.legend(loc='best')
| |
| ax2=axes([0,0,1,1])
| |
| ip=InsetPosition(ax1,[0.5,0.3,0.5,0.5])
| |
| ax2.set_axes_locator(ip)
| |
| ax2.plot(E1,P2,label=r'$k_B T = 2.2$')
| |
| #ax2.plot(E1,P2exact,label=r'$k_B T = 2.2$ exata')
| |
| ax2.plot(E1,P3,label=r'$k_B T = 2.4$')
| |
| #ax2.plot(E1,P3exact,label=r'$k_B T = 2.4$ exata')
| |
| #ax2.set_yscale('log')
| |
| ax2.set_xlabel('E/N')
| |
| ax2.set_ylabel(r'$g(E)e^{-E/k_B T}$')
| |
| ax2.legend(loc='best')
| |
| savefig('fig2.png')
| |
| show()
| |
| | |
| Te = linspace(0,8,255)
| |
| Thm=[];Thm_exact=[]
| |
| | |
| for T in Te:
| |
| Thm.append(Thermod(T, lngE, Energies, E0))
| |
| Thm_exact.append(Thermod(T, beale, Energies, E0))
| |
| | |
| Thm = array(Thm)
| |
| Thm_exact=array(Thm_exact)
| |
| | |
| #Energia interna
| |
| plt.plot(Te,Thm_exact[:,0],label='exata')
| |
| plt.plot(Te,Thm[:,0],label='simulação')
| |
| plt.xlabel(r'$k_B T$')
| |
| plt.ylabel(r'$U(T)/N$')
| |
| plt.legend(loc='best')
| |
| plt.show()
| |
| | |
| #Calor específico
| |
| plt.plot(Te,Thm_exact[:,1],label='exata')
| |
| plt.plot(Te,Thm[:,1],label='simulação')
| |
| plt.xlabel(r'$k_B T$')
| |
| plt.ylabel(r'$C(T)/N$')
| |
| plt.legend(loc='best')
| |
| plt.show()
| |
| | |
| #Energia livre de Helmholtz
| |
| plt.plot(Te,Thm_exact[:,2],label='exata')
| |
| plt.plot(Te,Thm[:,2],label='simulação')
| |
| plt.xlabel(r'$k_B T$')
| |
| plt.ylabel(r'$F(T)/N$')
| |
| plt.legend(loc='best')
| |
| plt.show()
| |
| | |
| #Entropia
| |
| plt.plot(Te,Thm_exact[:,3],label='exata')
| |
| plt.plot(Te,Thm[:,3],label='simulação')
| |
| plt.xlabel(r'$k_B T$')
| |
| plt.ylabel(r'$S(T)/N$')
| |
| plt.legend(loc='best')
| |
| plt.show()
| |
| | |
| erroUt=(abs(Thm[:,0]-Thm_exact[:,0]))/abs(Thm_exact[:,0])
| |
| erroCt=(abs(Thm[:,1]-Thm_exact[:,1]))/Thm_exact[:,1]
| |
| erroFt=(abs(Thm[:,2]-Thm_exact[:,2]))/abs(Thm_exact[:,2])
| |
| erroSt=(abs(Thm[:,3]-Thm_exact[:,3]))/Thm_exact[:,3]
| |
| | |
| #Erro relativo para energia interna
| |
| plt.plot(Te,erroUt)
| |
| xlabel(r'$k_B T$')
| |
| ylabel(r'$\epsilon [U(T)]$')
| |
| yscale('log')
| |
| plt.show()
| |
| | |
| #Erro relativo para calor específico
| |
| plt.plot(Te,erroCt)
| |
| xlabel(r'$k_B T$')
| |
| ylabel(r'$\epsilon [C(T)]$')
| |
| yscale('log')
| |
| plt.show()
| |
| | |
| #Erro relativo para energia livre de Helmholtz
| |
| plt.plot(Te,erroFt)
| |
| xlabel(r'$k_B T$')
| |
| ylabel(r'$\epsilon [F(T)]$')
| |
| yscale('log')
| |
| plt.show()
| |
| | |
| #Erro relativo para entropia
| |
| plt.plot(Te,erroSt)
| |
| xlabel(r'$k_B T$')
| |
| ylabel(r'$\epsilon [S(T)]$')
| |
| yscale('log')
| |
| plt.show()
| |
| | |
| figure(figsize=(10,8))
| |
| | |
| ax1=subplot(2,2,1)
| |
| ax1.plot(Te,Thm_exact[:,0],label='exata')
| |
| ax1.plot(Te,Thm[:,0],label='simulação')
| |
| ax1.set_xlabel(r'$k_B T$')
| |
| ax1.set_ylabel(r'$U(T)/N$')
| |
| ax1.legend(loc='best')
| |
| ax12=axes([1,0,0,0])
| |
| ip=InsetPosition(ax1,[0.685,0.5,0.3,0.3])
| |
| ax12.set_axes_locator(ip)
| |
| ax12.plot(Te,erroUt)
| |
| ax12.set_yscale('log')
| |
| ax12.set_xlabel(r'$k_B T$')
| |
| ax12.set_ylabel(r'$\epsilon [U(T)]$')
| |
| | |
| ax2=subplot(2,2,2)
| |
| ax2.plot(Te,Thm_exact[:,1],label='exata')
| |
| ax2.plot(Te,Thm[:,1],label='simulação')
| |
| ax2.set_xlabel(r'$k_B T$')
| |
| ax2.set_ylabel(r'$C(T)/N$')
| |
| ax2.legend(loc='best')
| |
| ax22=axes([0,1,0,0])
| |
| ip=InsetPosition(ax2,[0.685,0.5,0.3,0.3])
| |
| ax22.set_axes_locator(ip)
| |
| ax22.plot(Te,erroCt)
| |
| ax22.set_yscale('log')
| |
| ax22.set_xlabel(r'$k_B T$')
| |
| ax22.set_ylabel(r'$\epsilon [C(T)]$')
| |
| | |
| ax3=subplot(2,2,3)
| |
| ax3.plot(Te,Thm_exact[:,2],label='exata')
| |
| ax3.plot(Te,Thm[:,2],label='simulação')
| |
| ax3.set_xlabel(r'$k_B T$')
| |
| ax3.set_ylabel(r'$F(T)/N$')
| |
| ax3.legend(loc='best')
| |
| ax32=axes([0,0,1,0])
| |
| ip=InsetPosition(ax3,[0.25,0.2,0.3,0.3])
| |
| ax32.set_axes_locator(ip)
| |
| ax32.plot(Te,erroFt)
| |
| ax32.set_yscale('log')
| |
| ax32.set_xlabel(r'$k_B T$')
| |
| ax32.set_ylabel(r'$\epsilon [F(T)]$')
| |
| | |
| ax4=subplot(2,2,4)
| |
| ax4.plot(Te,Thm_exact[:,3],label='exata')
| |
| ax4.plot(Te,Thm[:,3],label='simulação')
| |
| ax4.set_xlabel(r'$k_B T$')
| |
| ax4.set_ylabel(r'$S(T)/N$')
| |
| ax4.legend(loc='best')
| |
| ax42=axes([0,0,0,1])
| |
| ip=InsetPosition(ax4,[0.685,0.5,0.3,0.3])
| |
| ax42.set_axes_locator(ip)
| |
| ax42.plot(Te,erroSt)
| |
| ax42.set_yscale('log')
| |
| ax42.set_xlabel(r'$k_B T$')
| |
| ax42.set_ylabel(r'$\epsilon [S(T)]$')
| |
| savefig('fig3.png')
| |
| show()
| |
| </source>
| |
|
| |
|
| ==Referências== | | ==Referências== |
|
| |
|
| <references> | | <references> |
Nomes: Rafael Abel da Silveira e William Machado Pantaleão
Introdução
Simulações computacionais, como o método de Monte Carlo, são vastamente utilizadas para estudar transições de fase e fenômenos críticos. O método padrão para simulações de Monte Carlo é o algoritmo de Metropolis, entretanto, algoritmos novos e mais eficientes são usados em simulações modernas, como o algoritmo de Wang-Landau. Ao contrário dos métodos convencionais de Monte Carlo, que geram diretamente uma distribuição canônica  a uma dada temperatura
a uma dada temperatura  , a abordagem de Wang-Landau estima a densidade de estados
, a abordagem de Wang-Landau estima a densidade de estados  diretamente por meio de um passeio aleatório, que produz um histograma plano no espaço de energia
diretamente por meio de um passeio aleatório, que produz um histograma plano no espaço de energia  . [1]
. [1]
Mesmo para modelos que podem ser resolvidos analiticamente, a densidade de estados  não pode ser determinada para sistemas maiores [2]. Com o algoritmo de Wang-Landau, é possível obter a
não pode ser determinada para sistemas maiores [2]. Com o algoritmo de Wang-Landau, é possível obter a  a partir de um passeio aleatório. A estimativa para
a partir de um passeio aleatório. A estimativa para  é melhorada a cada etapa do passeio aleatório, usando um fator de modificação
é melhorada a cada etapa do passeio aleatório, usando um fator de modificação  cuidadosamente controlado, para produzir um resultado que converge para o valor real rapidamente.
cuidadosamente controlado, para produzir um resultado que converge para o valor real rapidamente.
Amostragem de Wang-Landau
No início da simulação,  é desconhecido e fazemos uma estimativa inicial para ele. A abordagem mais simples é definir
é desconhecido e fazemos uma estimativa inicial para ele. A abordagem mais simples é definir  para todas as energias possíveis
para todas as energias possíveis  . A configuração de spin inicial para toda a rede pode ser escolhida arbitrariamente. Então, uma caminhada aleatória no espaço de energia é iniciada pela formação de estados de teste, cada um dos quais é produzido escolhendo aleatoriamente um spin e alterando seu estado.
. A configuração de spin inicial para toda a rede pode ser escolhida arbitrariamente. Então, uma caminhada aleatória no espaço de energia é iniciada pela formação de estados de teste, cada um dos quais é produzido escolhendo aleatoriamente um spin e alterando seu estado.
Cada vez que uma energia  é visitada, o histograma
é visitada, o histograma  é incrementado em 1. A estimativa de
é incrementado em 1. A estimativa de  é então modificada por um fator multiplicativo
é então modificada por um fator multiplicativo  , e o valor atualizado realiza um passeio aleatório adicional no espaço de
, e o valor atualizado realiza um passeio aleatório adicional no espaço de  .
.
Se  e
e  são as energias antes e depois de um valor de spin ser alterado, a probabilidade de transição da energia
são as energias antes e depois de um valor de spin ser alterado, a probabilidade de transição da energia  para
para  é dada por
é dada por

A razão das probabilidades de transição de  para
para  e de
e de  a
a  podem ser calculados como
podem ser calculados como

Logo, o algoritmo de passeio aleatório satisfaz o equilíbrio detalhado:

onde  é a probabilidade na energia
é a probabilidade na energia  e
e  é a probabilidade de transição de
é a probabilidade de transição de  para
para  .
.
Se o estado de energia  é aceito, a densidade de estados
é aceito, a densidade de estados  é multiplicada pelo fator de modificação
é multiplicada pelo fator de modificação  de maneira que
de maneira que  e a entrada no histograma para
e a entrada no histograma para  é atualizada de forma
é atualizada de forma  . Se o estado de energia não é aceito, a densidade de estados
. Se o estado de energia não é aceito, a densidade de estados  é multiplicada pelo fator de modificação,
é multiplicada pelo fator de modificação,  e
e  é atualizada de forma
é atualizada de forma  .
.
Flatness
O procedimento de passeio aleatório é seguido até o histograma  estar reto (do inglês, "flat"), e para determinar isso, a cada
estar reto (do inglês, "flat"), e para determinar isso, a cada  iterações verificamos se todos valores possíveis de
iterações verificamos se todos valores possíveis de  estão a uma distância, no máximo,
estão a uma distância, no máximo,  de
de  . A variável
. A variável  é denominada "flatness". Quando o histograma está reto, todos estados de energia foram visitados aproximadamente igualmente.
é denominada "flatness". Quando o histograma está reto, todos estados de energia foram visitados aproximadamente igualmente.
O número de passos, </math> n </math> que devemos realizar antes de checar deve ser maior que  onde
onde  indica o tamanho da rede, para que o algoritmo tenha a oportunidade de visitar cada estado pelo menos uma vez.
indica o tamanho da rede, para que o algoritmo tenha a oportunidade de visitar cada estado pelo menos uma vez.
Para sistemas simples, podemos utilizar um valor tão alto quanto 95%, entretanto, para este trabalho foi escolhido o valor de 80%.
Fator de modificação
Em geral, como  se torna muito grande, trabalhamos com o logaritmo natural dessas quantidades,
se torna muito grande, trabalhamos com o logaritmo natural dessas quantidades,  . Portanto, cada atualização da densidade de estados é dada por
. Portanto, cada atualização da densidade de estados é dada por  . O valor comumente utilizado para o fator de modificação é
. O valor comumente utilizado para o fator de modificação é  .
.
Quando o histograma é considerado reto, pelas condições descritas acima, reduzimos o valor de  de forma que o novo valor será
de forma que o novo valor será  , resetamos o histograma
, resetamos o histograma  e recomeçamos o passeio aleatório.
e recomeçamos o passeio aleatório.
A simulação é parada para um valor de  predeterminado. No caso, usamos
predeterminado. No caso, usamos  .
.
Aplicação ao Modelo de Ising 2D
Modelo de Ising
O modelo de Ising é uma rede 2D, de tamanho  que consiste de uma variável discreta em cada sítio que pode ser usada para representar o momento de dipolo magnético de um átomo[3] Cada sítio pode ter o valor de spin
que consiste de uma variável discreta em cada sítio que pode ser usada para representar o momento de dipolo magnético de um átomo[3] Cada sítio pode ter o valor de spin  ou
ou  .
.
Para este trabalho, o hamiltoniano de interação pode ser calculado por  onde
onde  indica pares distintos de vizinhos-mais-próximos.
indica pares distintos de vizinhos-mais-próximos.
Com a densidade de estados, podemos calcular as seguintes quantidades termodinâmicas:
Energia interna:

Calor específico:

Energia livre de Helmoltz:

Entropia:

Finalmente, podemos também calcular a distribuição canônica usando:

Algoritmo
Resumindo, o passo a passo do algoritmo pode ser escrito como:
1. Defino  para todos
para todos  e o fator de modificação inicial
e o fator de modificação inicial  ;
;
2. Aleatoriamente, escolho um spin e troco o seu valor. Aceito a transição com probabilidade  ;
;
3. Modifico a densidade de estados  e atualizo o histograma
e atualizo o histograma  ;
;
4. Continuo até o histograma estar reto, então diminuo o valor de  pela metade e reseto o histograma
pela metade e reseto o histograma  ;
;
5. Repito os passos 2-4 até  .
.
6. Obtendo a  , posso calcular as quantidades termodinâmicas descritas anteriormente.
, posso calcular as quantidades termodinâmicas descritas anteriormente.
Resultados
Densidade de estados
A estimativa da densidade de estados para  usando a amostragem de Wang-Landau é mostrada na Fig. 1, junto com os resultados exatos de Beale [2].
usando a amostragem de Wang-Landau é mostrada na Fig. 1, junto com os resultados exatos de Beale [2].
Os fatores de modificação inicial e final para os passeios aleatórios foram Falhou ao verificar gramática (erro de sintaxe): {\displaystyle ln(f_0) = 1 e ln(f_{final}) = 10^{−8} }
.
O histograma  foi considerado plano quando todas as entradas não eram inferiores a 80% da média
foi considerado plano quando todas as entradas não eram inferiores a 80% da média  .
.
A densidade absoluta de estados na Fig. 1 é obtida pela condição de que o número de estados fundamentais seja 2 para o modelo de Ising 2D.
Com a escala logarítmica usada na Fig. 1, os dados simulados e a solução exata se sobrepõem perfeitamente. Na inserção da Fig. 1, vemos que o erro relativo é, de fato, muito pequeno.
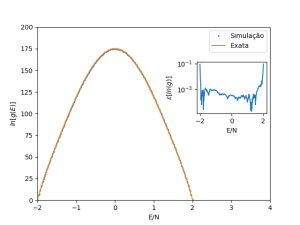
Figura 1: Logaritmo da densidade de estados

do modelo de Ising 2D com

.
Distribuição canônica
Podemos calcular a distribuição canônica usando a equação Falhou ao verificar gramática (erro de sintaxe): {\displaystyle P(E, T) = g(E)e^{−E/k_BT}}
a qualquer temperatura, sem a necessidade de realizar várias simulações.
Na Fig. 2, mostramos a distribuição canônica resultante na temperatura crítica  , que exibe um único pico.
, que exibe um único pico.
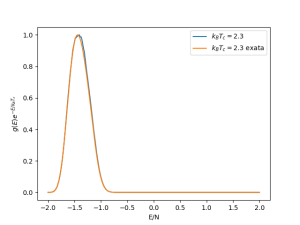
Figura 2: Distribuição canônica

na temperatura de transição do modelo de Ising 2D com

e

.
As distribuições em temperaturas acima e abaixo de  também apresentam pico único, conforme ilustrado no detalhe da Fig. 3.
também apresentam pico único, conforme ilustrado no detalhe da Fig. 3.
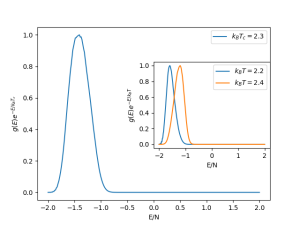
Figura 3: Distribuição canônica

nas temperaturas

e

do modelo de Ising 2D com

.
Quantidades termodinâmicas
Os resultados simulados, calculados diretamente com as equações descritas no capítulo anterior, e as soluções exatas se sobrepõem quase perfeitamente em uma ampla região de temperatura de  .
.
Testes mais rigorosos de precisão são fornecidos a partir dos erros relativos para as respectivas quantidades termodinâmicas. Os erros relativos são muito pequenos para toda a região de temperatura de  .
.
Como o sistema tem uma transição de fase de segunda ordem, a primeira derivada da energia livre é uma função contínua da temperatura. Não há saltos na energia interna ou na entropia, mesmo no limite, pois o
tamanho do sistema vai para o infinito.
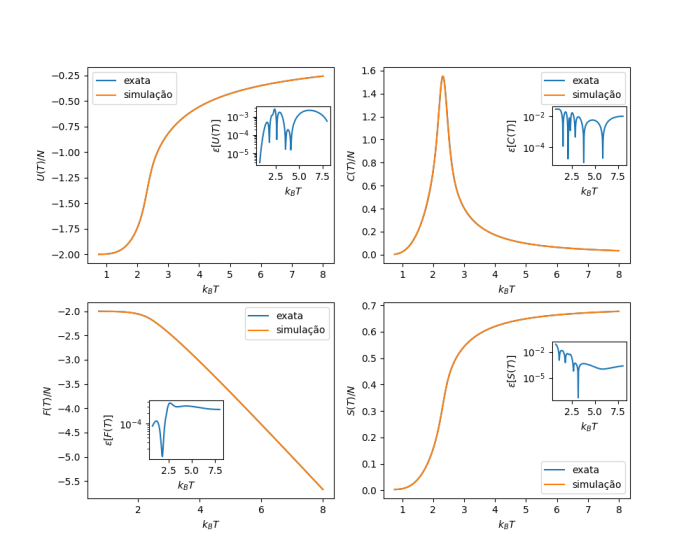
Figura 4: Quantidades termodinâmicas do modelo de Ising 2D com

calculado a partir da densidade de estados

. Na figura, estão mostrados: (a) energia interna, (b) calor específico, (c) energia livre de Helmholtz e (d) entropia. Os gráficos
inset são os erros relativos.
Código
Adicionar programas usados
Referências
<references>
- ↑ D. P. Landau, Shan-ho Tsai, M. Exler, A new approach to Monte Carlo simulations in statistical physics: Wang-Landau sampling, American Journal of Physics 72, 1294 (2004). https://doi.org/10.1119/1.1707017
- ↑ 2,0 2,1 P. D. Beale, Exact Distribution of Energies in the Two-Dimensional Ising Model, Phys. Rev. Lett. 76,78 (1996). https://doi.org/10.1103/PhysRevLett.76.78
- ↑ A. Rosa, C. Pires, L. Doria, Ising 2D, Wiki da Física Computacional da UFRGS. https://fiscomp.if.ufrgs.br/index.php/Ising_2D#Modelo_de_Ising



























































