Código em Python para simulações de osciladores lineares e FPUT quadrático
Bibliotecas usadas:
import numpy as np
import random
import matplotlib.pyplot as plt
import os
import imageio
Funções:
#flag == 1 -> Duas massas acopladas por mola central (livres nos lados) -> Não está sendo usado neste trabalho
#flag == 2 -> Oscilador acoplado com duas massas e três molas (linear) -> Não precisaria mais existir... o flag 3 já # resolve o caso N=2 também
#flag == 3 -> Oscilador acoplado com N>=2 massas e N+1 molas (linear)
#flag == 4 -> FPUT com termo quadrático
# L é o tamanho unidimensional de todo o sistema, de parede a parede;
# N é o número de partículas
# m é o número do modo que se quer inicializar: m=1,...,N (pode ser maior q N, mas vai repetir o primeiro modo)
def condicao_inicial_1d(L, N, m, flag):
#Espaçamento das posições de equilíbrio das partículas
step = L/(N+1)
posx = np.zeros((N,1), dtype="float")
posxeq = np.zeros((N,1), dtype="float")
for i in range(0, N):
posxeq[i] = (i+1)*step
l = posxeq[1] - posxeq[0]
#Duas massas acopladas por uma mola, ligeiramente comprimidas. Deixei por razões sentimentais, foi o primeiro que
#fiz.
if(flag==1):
posx[0] = posxeq[0]+0.5
posx[1] = posxeq[1]-0.5
#Duas massas, três molas, sistema acoplado:
elif(flag==2):
for i in range(0, N):
posx[i] = modo_de_osc(posxeq[i], i, m, 0.5, N)
#N>=2 massas, N+1 molas, sistema acoplado, condição randômica de deslocamento do equilíbrio comentada:
elif(flag==3):
for i in range(N):
#posx[i] = posxeq[i] + random.uniform(-l/5, +l/5) -> Se quiser condição inicial randômica, descomentar essa
#linha e comentar a linha seguinte
posx[i] = modo_de_osc(posxeq[i], i, m, 0.5, N)
#FPUT:
elif(flag==4):
for i in range(0, N):
posx[i] = modo_de_osc(posxeq[i], i, m, 0.5, N)
return posx, posxeq
# Para inicializar o sistema em um determinado modo de oscilação no tempo t=0
# l é a posição da partícula na cadeia (de 0 a N-1), m é o número do modo que se quer: 1, 2, 3,...,N
# am é a amplitude da oscilação (tecnicamente, a amplitude do modo vai ser am*np.sin(...))
def modo_de_osc(poseq, l, m, am, N):
return poseq + am*np.sin(np.pi*(l+1)*m/(N+1))
#Só para corda do FPUT, não precisa de flag
def condicao_inicial_1d_vertical(L, N, m):
pos = np.zeros((N,1), dtype="float")
poseq = np.zeros((N,1), dtype="float")
for i in range(0, N):
pos[i] = modo_de_osc(poseq[i], i, m, 0.5, N)
return pos
def condicao_inicial_2d(N):
N_sqrt = int(np.sqrt(N)) # N tem q ser um quadrado perfeito!
L = N_sqrt + 1
l = step = 1
posx = np.zeros((N,1), dtype="float")
posxeq = np.zeros((N,1), dtype="float")
posy = np.zeros((N,1), dtype="float")
posyeq = np.zeros((N,1), dtype="float")
for i in range(N):
posxeq[i] = (i%N_sqrt)*step + 1
posyeq[i] = int(i/N_sqrt)
posx[i] = posxeq[i] + random.uniform(-l/5, +l/5)
posy[i] = posyeq[i] + random.uniform(-l/5, +l/5)
#Oscilador 2D também poderia ter inicialização por modo... mas não implementei.
return L, posx, posy, posxeq, posyeq
def acel_quad(pos, poseq, k, m, alpha, flag):
size = len(pos)
l = poseq[1] - poseq[0]
a = np.zeros((size,1), dtype="float")
if(flag==1):
a[0] = -k/m*(pos[0]-pos[1]+l) #l é a distância entre as massas quando a mola está relaxada
a[1] = k/m*(pos[0]-pos[1]+l)
elif(flag==2):
a[0] = k/m*((pos[1]-poseq[1])-2*(pos[0]-poseq[0]))
a[1] = k/m*((pos[0]-poseq[0])-2*(pos[1]-poseq[1]))
elif(flag==3 or flag==4): #Se flag==3, cuidar para fazer alpha=0!
x = pos-poseq
for i in range(size):
#Cuidar as fronteiras
if(i==0):
a[i] = k/m * (x[i+1] - 2*x[i] - alpha*(x[i]**2-(x[i+1]-x[i])**2))
elif(i==(size-1)):
a[i] = k/m * (x[i-1] - 2*x[i] - alpha*((x[i]-x[i-1])**2-x[i]**2))
else:
a[i] = k/m * (x[i+1] + x[i-1] - 2*x[i]) * (1.0 + alpha * (x[i+1] - x[i-1]));
return a
def velocity_verlet(pos, poseq, vel, acel, k, m, alpha, flag):
size = len(pos)
new_acel = np.zeros((size,1), dtype="float")
new_pos = np.zeros((size,1), dtype="float")
new_vel = np.zeros((size,1), dtype="float")
new_pos = pos + vel*dt + 0.5*acel*dt**2
new_acel = acel_quad(new_pos, poseq, k, m, alpha, flag)
new_vel = vel + 0.5*(acel + new_acel)*dt
return new_pos, new_vel, new_acel
def plota_figura_1d(L, posX, posY, t, flag):
figname = 'f{:.1f}.jpeg'.format(t)
plt.figure(figsize=(5,3))
plt.xlim(0, L)
plt.ylim(-1, 1)
#plt.yticks([])
plt.axis("off")
#plt.grid()
if(flag==1):
plt.title('Duas massas acopladas por uma mola - Tempo {:.1f}'.format(t))
plt.scatter(posX, posY, s=300, c="black")
elif(flag==2 or flag == 3):
plt.title('Oscilador Acoplado Linear - Tempo {:.1f}'.format(t))
plt.scatter(posX, posY, s=300, c="black")
elif(flag==4):
plt.title('FPUT quadrático - Tempo {:.1f}'.format(t))
plt.scatter(posX, posY, s=5, c="black")
plt.axvline(x=0)
plt.axvline(x=L)
if(flag != 4):
size = len(posX)
aj = 0.4 #Ajuste para plotar as linhas horizontais, varia com o tamanho do scatter, aqui, s=300
for i in range(size):
if(i==0):
plt.axhline(y=0, xmin=0, xmax=(posX[i]-aj)/L, color='r', linestyle=':')
plt.axhline(y=0, xmin=(posX[i]+aj)/L, xmax=(posX[i+1]-aj)/L, color='k', linestyle=':')
elif(i==(size-1)):
plt.axhline(y=0, xmin=(posX[i]+aj)/L, xmax=1, color='r', linestyle=':')
else:
plt.axhline(y=0, xmin=(posX[i]+aj)/L, xmax=(posX[i+1]-aj)/L, color='k', linestyle=':')
plt.savefig(figname, dpi=100)
plt.close()
return figname
def plota_figura_2d(L, posX, posY, t, flag):
figname = 'f{:.1f}.jpeg'.format(t)
plt.figure(figsize=(5,3))
plt.xlim(0, L)
plt.ylim(-1, L)
#plt.yticks([])
plt.axis("off")
#plt.grid()
plt.title('Oscilador Acoplado Linear 2D - Tempo {:.1f}'.format(t))
plt.scatter(posX, posY, s=300, c="black")
plt.savefig(figname, dpi=100)
plt.close()
return figname
def energia_do_modo(x, x_old, N, m, dt):
#Energia do modo m
omega = 2*np.sin(m*np.pi/(2*(N+1)))
omega2 = omega**2
tmp = np.arange(1, N+1, 1)
tmp[-1:] = 0
pos = np.reshape(x, (1,N))
pos_old = np.reshape(x_old, (1,N))
produto_escalar = np.sqrt(2.0 / (N+1)) * np.dot(pos, np.sin( tmp * m * np.pi / (N+1) ))
produto_escalar_ant = np.sqrt(2.0 / (N+1)) * np.dot(pos_old, np.sin( ( tmp * m * np.pi / (N+1) ) ))
return 0.5 * (produto_escalar - produto_escalar_ant)**2 / dt**2 + (0.5 * omega2 * produto_escalar**2)
Imprimir os cinco primeiros modos de oscilação para N=32 partículas (Figura 12 da Wiki).
N=32
modo = np.zeros((5,N), dtype="float")
nodes = [i for i in range(1,33)]
for i in range(0,5):
for j in range(0, N):
modo[i,j] = modo[i,j] + np.sin((j+1)*np.pi*(i+1)/(N+1))
plt.plot(nodes, modo[0,:], "k.", label="Modo 1")
plt.plot(nodes, modo[1,:], "r-.", label="Modo 2")
plt.plot(nodes, modo[2,:], "b-", label="Modo 3")
plt.plot(nodes, modo[3,:], "g--", label="Modo 4")
plt.plot(nodes, modo[4,:], "y:", label="Modo 5")
plt.xlabel("Número da partícula")
plt.ylabel("Deslocamento em relação à posição de equilíbrio")
plt.legend()
plt.show()
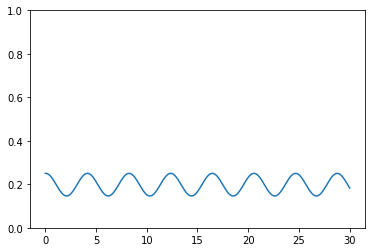
Exemplo de como podem ser feitos os gráficos dos modos e como comparar com as frequências esperadas (gráficos 2 a 11 da Wiki). Também estou calculando a energia total do sistema. O exemplo é do modo 1, do caso N=3. Note que a energia não é exatamente conservada, é uma dificuldade do método velocity Verlet. Para N=2 a conservação é bem obedecida, uma reta próxima a 0.2. Seria possível melhorar diminuindo o passo de integração, mas preferi deixar assim para ilustrar.
alpha = 0.0
k = 1.0
massa = 1.0
L = 12
N = 3
t=0.0
dt = 0.1
tmax = 30
posX = np.zeros((N,1), dtype="float")
posXeq = np.zeros((N,1), dtype="float")
posY = np.zeros((N,1), dtype="float")
velX = np.zeros((N,1), dtype="float")
acelX = np.zeros((N,1), dtype="float")
posX, posXeq = condicao_inicial_1d(L, N, 1, 3)
x1 = []
x2 = []
x3 = []
dotx1 = []
dotx2 = []
dotx3 = []
energia = []
x1.append(posX[0]-posXeq[0])
x2.append(posX[1]-posXeq[1])
x3.append(posX[2]-posXeq[2])
dotx1.append(velX[0])
dotx2.append(velX[1])
dotx3.append(velX[2])
energia.append(0.5*((posX[0]-posXeq[0])**2+(posX[1]-posXeq[1])**2+(posX[2]-posXeq[2])**2))
while(t < tmax):
posX, velX, acelX = velocity_verlet(posX, posXeq, velX, acelX, k, massa, alpha, 3)
x1.append(posX[0]-posXeq[0])
x2.append(posX[1]-posXeq[1])
x3.append(posX[2]-posXeq[2])
dotx1.append(velX[0])
dotx2.append(velX[1])
dotx3.append(velX[2])
energia.append(0.5*((posX[0]-posXeq[0])**2+(posX[1]-posXeq[1])**2+(posX[2]-posXeq[2])**2) + 0.5*(velX[0]**2+velX[1]**2+velX[2]**2))
t += dt
time = np.linspace(0, tmax, len(energia))
plt.plot(time, energia)
plt.ylim(0, 1)
plt.show()
time = np.linspace(0, 30, len(x1))
plt.plot(time, 0.35*np.cos(np.sqrt(2-np.sqrt(2))*time), "g-", label=r"$cos(\sqrt{2-\sqrt{2}}t)$")
plt.plot(time, x1, "k.", label=r"$x_{1}$")
#plt.plot(time, x2, "r-", label=r"$x_{2}$")
#plt.plot(time, x3, "b.", label=r"$x_{2}$")
plt.xlim(0,30)
plt.xlabel("t")
plt.ylabel("Deslocamento em relação à posição de equilíbrio")
plt.title("Oscilador Linear (Modo 1) - N=3")
plt.legend(loc="lower right")
plt.show()
FPUT unidimensional, deslocamento horizontal:
Setar alpha diferente de zero e flag=4 (último parâmetro das funções condicao_inicial(), plota_figura_1d() e velocity_verlet()).
Salva animação no arquivo fput.gif. Bem grande o gif gerado.
As animações não têm linhas representando as molas e o tamanho das partículas é bem pequeno. Fiz isso de propósito pra visualizar melhor o caso com N=32 partículas, do artigo da RBF.
Lembrar de alterar as variáveis globais de acordo. Diferentemente do caso bidimensional, o valor de L tem q ser fornecido e deve ser obviamente diferente de zero.
alpha = 0.25
k = 1.0
massa = 1.0
L = 33
N = 32
images=[]
t=0.0
dt = 0.1
tmax = 30
posX = np.zeros((N,1), dtype="float")
posXeq = np.zeros((N,1), dtype="float")
#Por enquanto não estou usando posições em Y
posY = np.zeros((N,1), dtype="float")
velX = np.zeros((N,1), dtype="float")
acelX = np.zeros((N,1), dtype="float")
posX, posXeq = condicao_inicial_1d(L, N, 1, 4)
while(t < tmax):
figname = plota_figura_1d(L, posX, posY, t, 4)
images.append(imageio.imread(figname))
os.remove(figname)
posX, velX, acelX = velocity_verlet(posX, posXeq, velX, acelX, k, massa, alpha, 4)
t += dt
imageio.mimsave('fput.gif', images, fps=30)
FPUT unidimensional, deslocamento vertical (como no programa em C):
Setar alpha diferente de zero e flag=4 (último parâmetro das funções condicao_inicial(), plota_figura_1d() e velocity_verlet()).
alpha = 0.25
k = 1.0
massa = 1.0
L = 33
N = 32
images=[]
t=0.0
dt = 0.1
tmax = 100
posX = np.zeros((N,1), dtype="float")
posY = np.zeros((N,1), dtype="float")
posYeq = np.zeros((N,1), dtype="float")
velY = np.zeros((N,1), dtype="float")
acelY = np.zeros((N,1), dtype="float")
step = L/(N+1)
for i in range(0, N):
posX[i] = (i+1)*step
posY = condicao_inicial_1d_vertical(L, N, 1)
while(t < tmax):
figname = plota_figura_1d(L, posX, posY, t, 4)
images.append(imageio.imread(figname))
os.remove(figname)
posY, velY, acelY = velocity_verlet(posY, posYeq, velY, acelY, k, massa, alpha, 4)
t += dt
imageio.mimsave('fput.gif', images, fps=30)
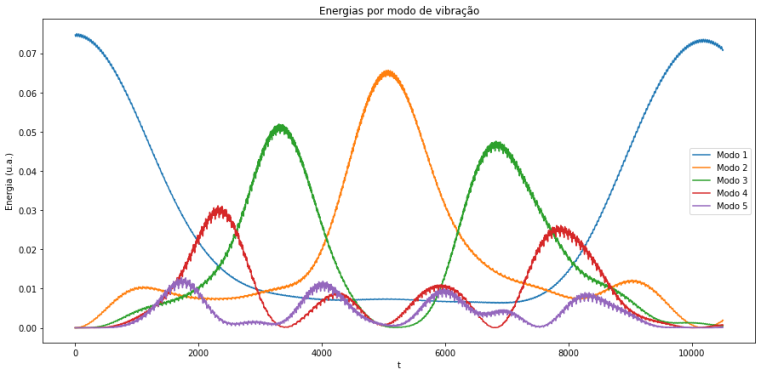
Cálculo e gráficos das energias por modo do FPUT quadrático:
alpha = 0.25
k = 1.0
massa = 1.0
L = 33
N = 32
images=[]
t=0.0
dt = 0.1
tmax = 14000
energia1 = []
energia2 = []
energia3 = []
energia4 = []
energia5 = []
posX = np.zeros((N,1), dtype="float")
posY = np.zeros((N,1), dtype="float")
posYeq = np.zeros((N,1), dtype="float")
velY = np.zeros((N,1), dtype="float")
acelY = np.zeros((N,1), dtype="float")
step = L/(N+1)
for i in range(0, N):
posX[i] = (i+1)*step
posY = condicao_inicial_1d_vertical(L, N, 1)
while(t < tmax):
pos_old = posY
posY, velY, acelY = velocity_verlet(posY, posYeq, velY, acelY, k, massa, alpha, 4)
energia1.append(energia_do_modo(posY, pos_old, N, 1, dt))
energia2.append(energia_do_modo(posY, pos_old, N, 2, dt))
energia3.append(energia_do_modo(posY, pos_old, N, 3, dt))
energia4.append(energia_do_modo(posY, pos_old, N, 4, dt))
energia5.append(energia_do_modo(posY, pos_old, N, 5, dt))
t += dt
time = np.linspace(0, tmax, int(tmax/dt))
plt.figure(figsize=(15,7))
plt.title('Energias por modo de vibração')
plt.xlabel("t")
plt.ylabel("Energia (u.a.)")
plt.plot(time, energia1,label="Modo 1")
plt.plot(time, energia2,label="Modo 2")
plt.plot(time, energia3,label="Modo 3")
plt.plot(time, energia4,label="Modo 4")
plt.plot(time, energia5,label="Modo 5")
plt.legend()
Osciladores acoplados lineares unidimensionais:
Salva animação no arquivo OA1D-N#.gif, com # igual ao número de partículas da simulação. Bem grande o gif gerado. Fica ainda maior quanto mais partículas tiver a simulação. Lembrar de alterar as variáveis globais de acordo. Diferentemente do caso bidimensional, o valor de L tem q ser fornecido e deve ser obviamente diferente de zero.
alpha = 0.0
k = 1.0
massa = 1.0
L = 12
N = 4
images=[]
t=0.0
dt = 0.1
tmax = 50
posX = np.zeros((N,1), dtype="float")
posXeq = np.zeros((N,1), dtype="float")
posY = np.zeros((N,1), dtype="float")
velX = np.zeros((N,1), dtype="float")
acelX = np.zeros((N,1), dtype="float")
posX, posXeq = condicao_inicial_1d(L, N, 1, 3)
while(t < tmax):
figname = plota_figura_1d(L, posX, posY, t, 3)
images.append(imageio.imread(figname))
os.remove(figname)
posX, velX, acelX = velocity_verlet(posX, posXeq, velX, acelX, k, massa, alpha, 3)
t += dt
imageio.mimsave('OA1D-N{:.0f}.gif'.format(N), images, fps=60)
Osciladores acoplados em uma rede bidimensional L x L (Aqui o L é calculado de maneira a ter um grid quadrado com espaçamento 1 entre as partículas, só precisa ser fornecida a quantidade de partículas N). N TEM QUE SER UM QUADRADO PERFEITO. LEMBRAR DE ALTERAR AS VARIÁVEIS GLOBAIS, FAZER L=0.0 E N = 4, 9, 16, 25,..., ETC.
alpha = 0.0
k = 1.0
massa = 6.0
L = 0.0
N = 49
images=[]
t=0.0
dt = 0.1
tmax = 30
posX = np.zeros((N,1), dtype="float")
posXeq = np.zeros((N,1), dtype="float")
posY = np.zeros((N,1), dtype="float")
posYeq = np.zeros((N,1), dtype="float")
velX = np.zeros((N,1), dtype="float")
velY = np.zeros((N,1), dtype="float")
acelX = np.zeros((N,1), dtype="float")
acelY = np.zeros((N,1), dtype="float")
L, posX, posY, posXeq, posYeq = condicao_inicial_2d(N)
while(t < tmax):
figname = plota_figura_2d(L, posX, posY, t, 3)
images.append(imageio.imread(figname))
os.remove(figname)
posX, velX, acelX = velocity_verlet(posX, posXeq, velX, acelX, k, massa, alpha, 3)
posY, velY, acelY = velocity_verlet(posY, posYeq, velY, acelY, k, massa, alpha, 3)
t += dt
imageio.mimsave('OA2D-N{:.0f}.gif'.format(N), images, fps=60)