Potencial de Lennard-Jones: mudanças entre as edições
| (Uma revisão intermediária pelo mesmo usuário não está sendo mostrada) | |||
| Linha 128: | Linha 128: | ||
|} | |} | ||
Para checar se a energia segue a distribuição de poisson, olhamos os histogramas. | Para checar se a energia segue a distribuição de poisson, olhamos os histogramas. Todos parecem seguir a distribuição de poisson correta, exceto no caso da densidade 0.9 e temperatura 0.5, onde ocorreu algum erro. | ||
== Posição final das Partículas == | == Posição final das Partículas == | ||
Edição atual tal como às 03h20min de 20 de outubro de 2022
Nome: André Bracht Burmeister
Fluidos estão em todos os lugares e, portanto, estudá-los é muito importante. Como são objetos de estudo extremamente complicados, se fazem necessários modelos e simplificações. Uma simplificação muito usada é a análise do sistema em duas dimensões apenas, excluindo a terceira dimensão espacial.
O Modelo
Um fluido bidimensional pode ser modelado como partículas em um plano com densidade (partículas por ). O plano é um quadrado de lado . Para escolher a posição inicial de uma partícula, são escolhidas coordenadas e aleatórias entre e .
O Método de Monte Carlo
Para evoluir o sistema, usaremos caminhadas aletórias, escolhendo as partículas com o método de Monte Carlo[[1]]. O método de Monte Carlo consiste em escolher uma partícula aleatoriamente entre as . Então escolhemos aleatoriamente um deslocamento proposto para a partícula , onde e estão entre e . A partícula só é realmente deslocada em de acordo com o Algoritmo de Metropolis-Hasting.
Algoritmo de Metropolis-Hasting
O Algoritmo de Metropolis-Hasting consiste em escolher se o movimento será ou não aceito usando a diferença de energia potencial entre o estado após o movimento e anterior ao movimento . A probabilidade de aceitar o movimento é dada por:
Onde , é a temperatura e é a constante de Boltzmann.
O Potencial de Lennard Jones
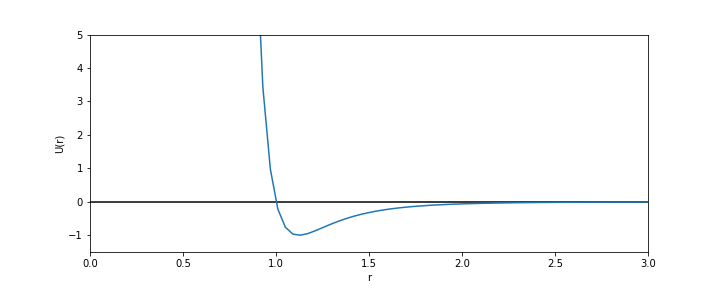
Para que o algoritmo funcione, precisamos então encontrar uma maneira de calcular a energia potencial de cada partícula. O potencial que uma partícula causa na outra, pode ser aproximado a uma distribuição em termos da distância entre elas:
Esse potencial é conhecido como o potencial de Lennard-Jones, primeiro apresentado por ele em seu artigo [1].
Unidades Reduzidas
Ao realizar a simulação computacionalmente, usar unidades do sistema internacional faria com que os números ficassem muito grandes ou muito pequenos, fazendo com que perdêssemos em precisão. Portanto, faz sentido que usemos unidades reduzidas em termos de , e a constante de Boltzmann . As unidades utilizadas foram:
| Grandeza | Comprimento | Temperatura | Energia | Densidade |
|---|---|---|---|---|
| Unidade |
Usando as novas unidades o potencial de Lennard-Jones fica:
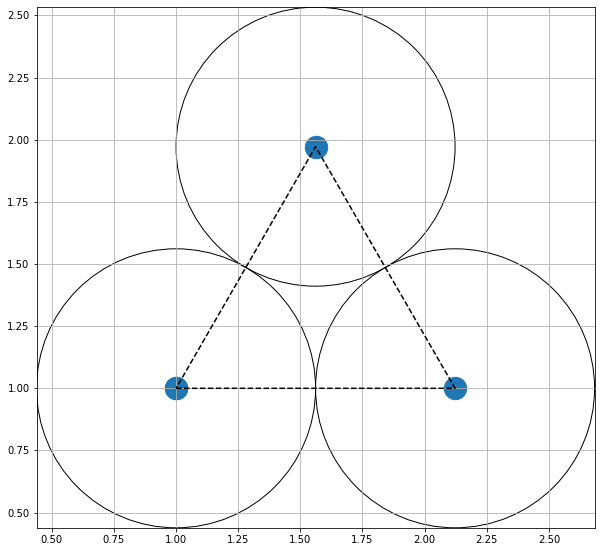
O formato do potencial faz com que para distâncias grandes, a interação entre partículas seja muito pequena e, para distâncias muito pequenas, haja uma força de repulsão que tende ao infinito. Além disso, a região de potencial mínimo se encontra a do centro da partícula. Ou seja, as partículas tendem a se organizar em uma configuração similar a da figura à direita, onde o raio dos círculos é
| Potencial de Lennard-Jones com unidades reduzidas |
|---|
Escolha de
Foi escolhido um deslocamento máximo inicial de , pois isso faz com que o deslocamento não seja maior que metade da distância ao mínimo do potencial. Mesmo assim, a depender da densidade e temperatura, pode ser que o deslocamento escolhido seja muito grande ou muito pequeno.
Se deslocamento máximo é muito grande, muitos movimentos acabam caindo próximos de outras partículas onde o potencial é muito alto, fazendo com que poucos movimentos sejam aceitos. Por outro lado, se o deslocamento é pequeno demais, muitos movimentos são aceitos (pequenos ajustes que fazem a energia ficar levemente menor), mas as partículas não exploram suficientemente as configurações possíveis. Portando, o valor de deve variar, dependendo da taxa de aceitação.
Na simulação, foi usado o seguinte algoritmo: Se a taxa de aceitação () sobe acima de 70%, é multiplicado por (para que a aceitação suba). Se a taxa de aceitação fica abaixo de 30%, é multiplicado por (para que a aceitação desça).
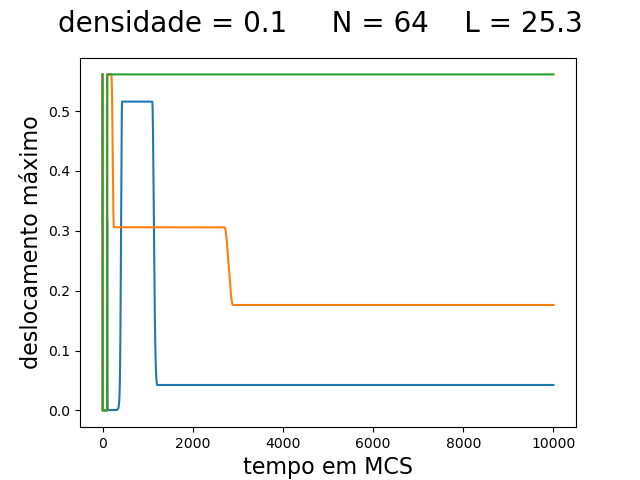
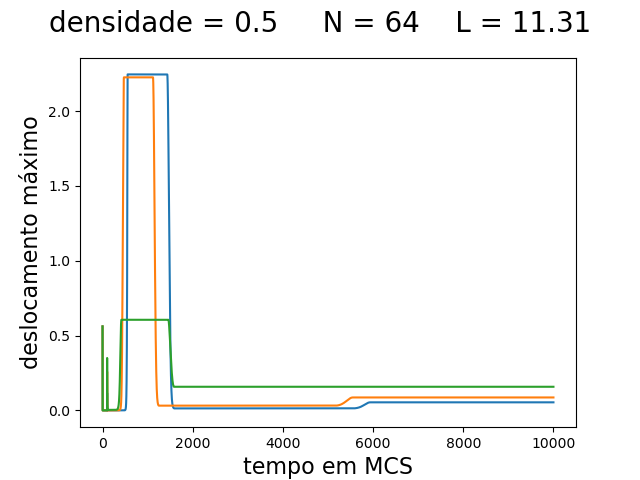
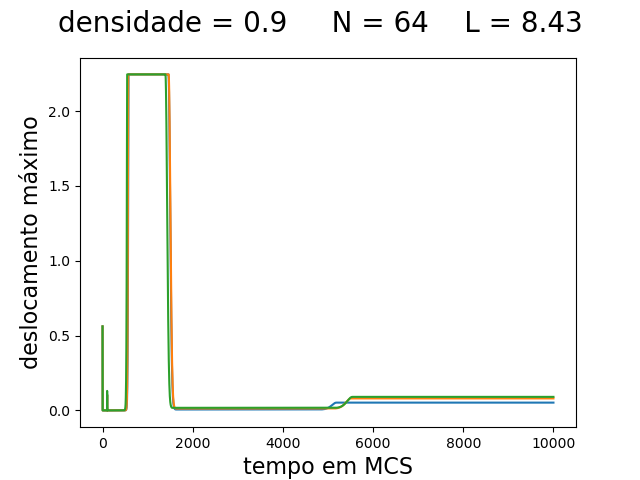
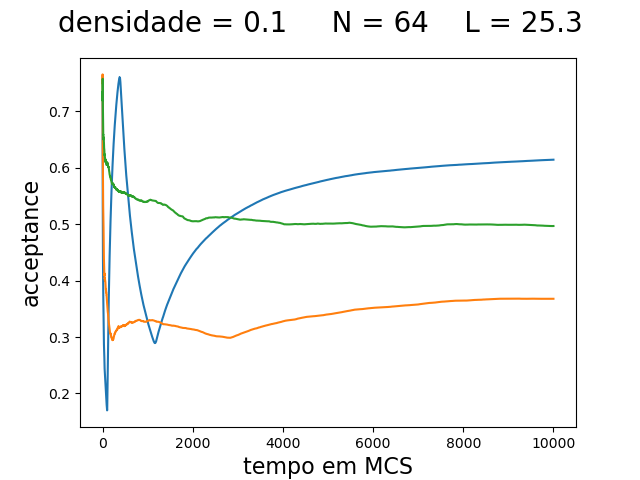
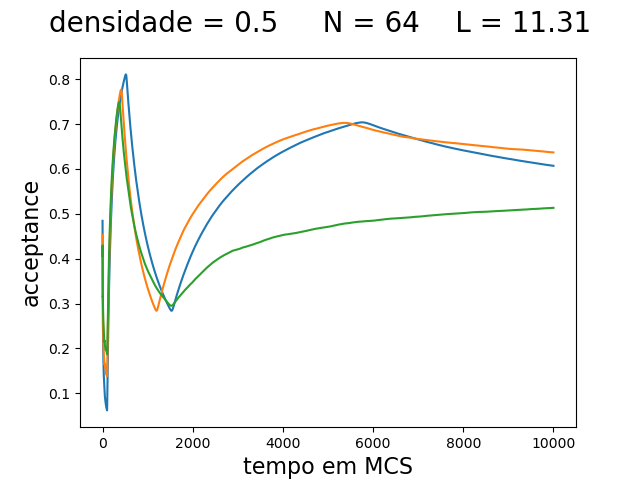
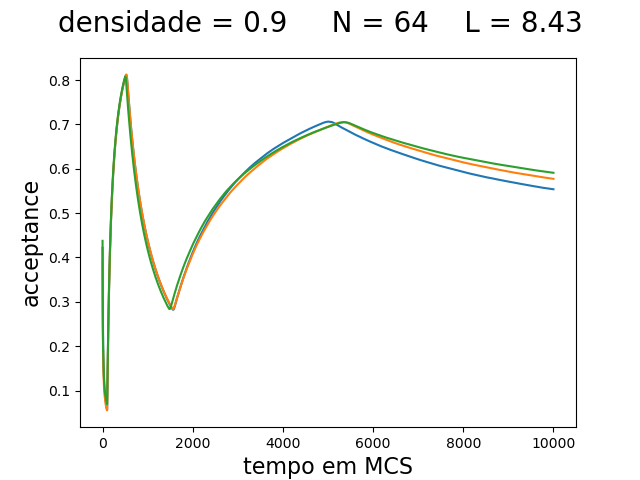
O deslocamento máximo e a aceitação ao longo da simulação são mostrados na imagem abaixo:
| Deslocamento máximo e aceitação ao longo da simulação | ||
|---|---|---|
| Os gráficos da primeira linha, mostram da esquerda para a direita: o deslocamento máximo () para densidade 0.1, 0.5 e 0.9 respectivamente. Os gráficos da segunda linha, mostram da esquerda para a direita: a taxa de aceitação () para densidade 0.1, 0.5 e 0.9 respectivamente. As linhas em azul, laranja e verde são as medidas feitas nas simulações com temperatura de 0.1, 0.3 e 0.5 respectivamente. | ||
Resultados
A simulação foi feita com 64 partículas, para temperaturas de 0.1, 0.3 e 0.5, densidades de 0.1, 0.5 e 0.9 e com 10 mil MCS. Onde 1 MCS (monte carlo step) é o número de passos correspondente ao número de partículas (nesse caso, 1 MCS = 64 passos).
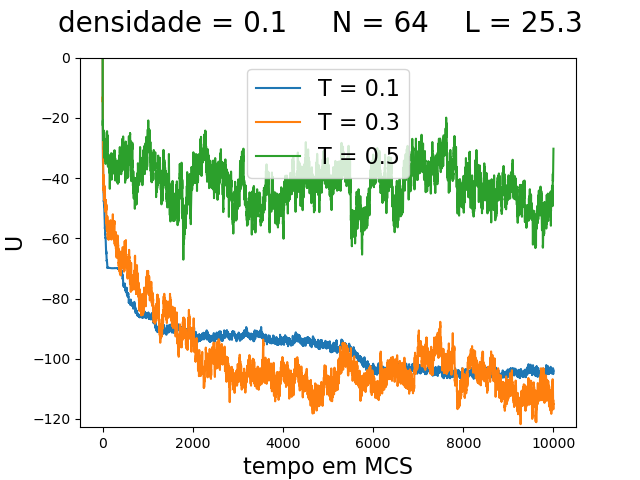
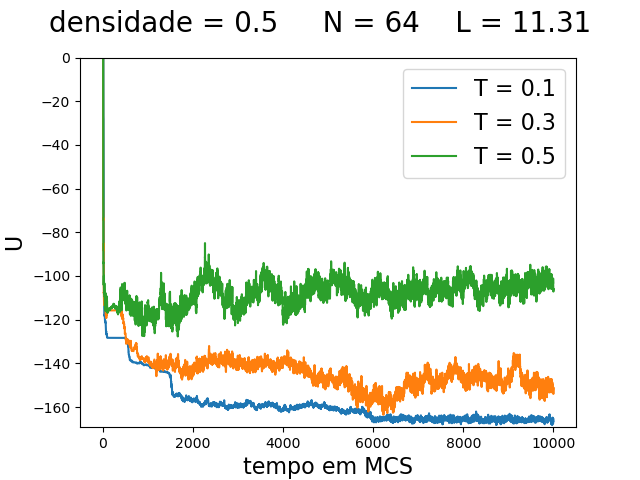
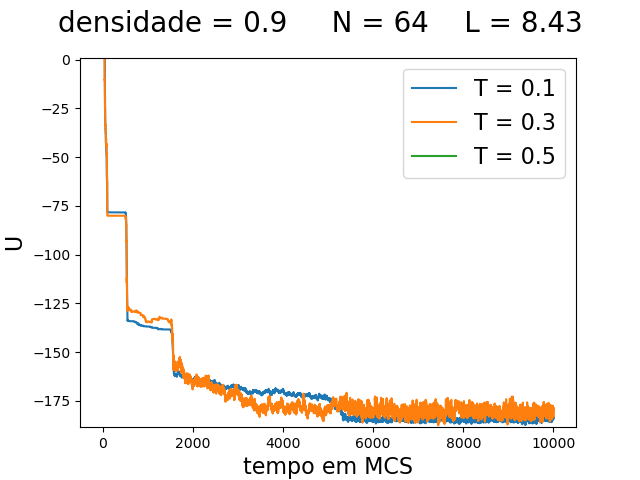
Energia
| Medidas de energia a cada para diferentes densidades e temperaturas | ||
|---|---|---|
| Os gráficos mostram da esquerda para a direita: a energia potencial total do sistema () para densidade 0.1, 0.5 e 0.9 respectivamente. As linhas em azul, laranja e verde são as medidas feitas nas simulações com temperatura de 0.1, 0.3 e 0.5 respectivamente. O gráfico a direita, com densidade 0.9 não possui dados para temperatura 0.5, pois ococrreu um erro na simulação. | ||
Olhando para os gráficos de energia podemos notar uma grande diferença entre temperaturas e densidades. Claramente quanto maior a temperatura, maior a energia, o que é esperado. Além disso, quando maior a temperatura, aparentemente há uma maior variabilidade na energia, o que também é esperado, pois a temperatura maior, faz com que mais movimentos desfavoráveis () sejam aceitos.
Olhando para a densidade, fica claro, que quanto maior a densidade, menor a energia. Isso provavelmente acontece, pois com maior densidade as partículas ficam confinadas à configuração típica de menor energia [[2]]. Já com menores densidades, as partículas se agrupam em grupos separados, que dificilmente se juntam. A separaçao desses grupos faz com que o potencial de um grupo no outro não seja tão negativo, aumentando a energia potencial total do sistema.
Outra consideração importante é que, com densidades 0.1 e 0.3, há uma separação maior entre a energia com temperatura 0.3 e 0.5, do que há entre a energia com temperatura 0.1 e 0.3. Não há dados para a simulação com temperatura 0.5 e densidade 0.9, pois houve um erro na simulação.
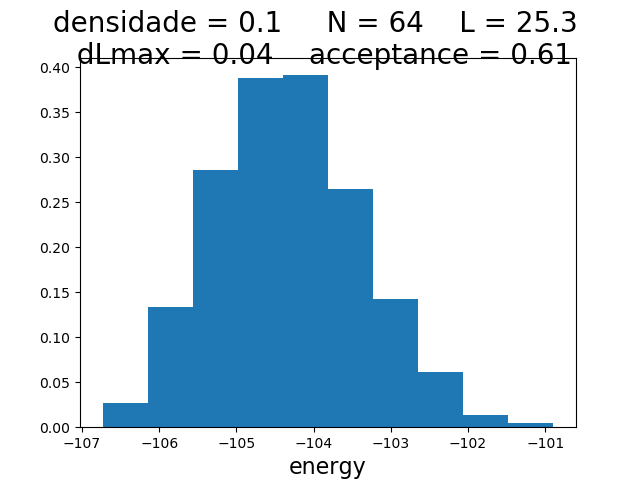
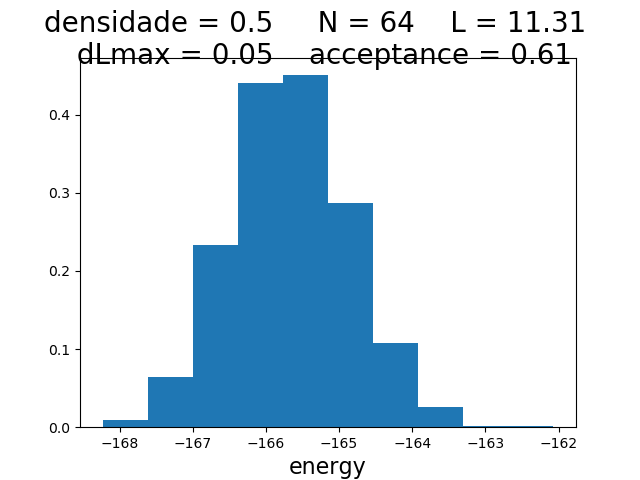
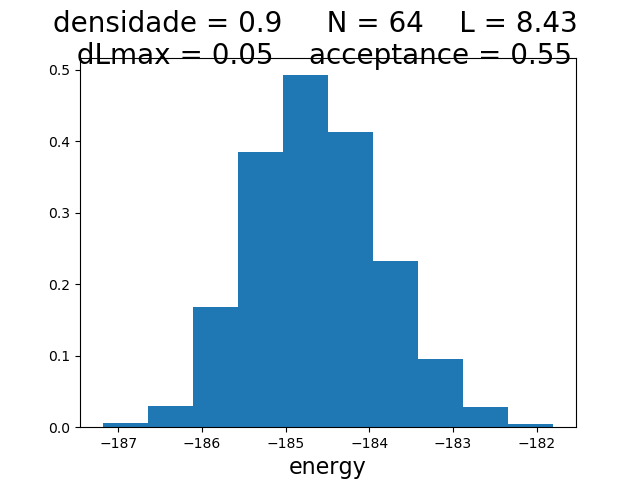
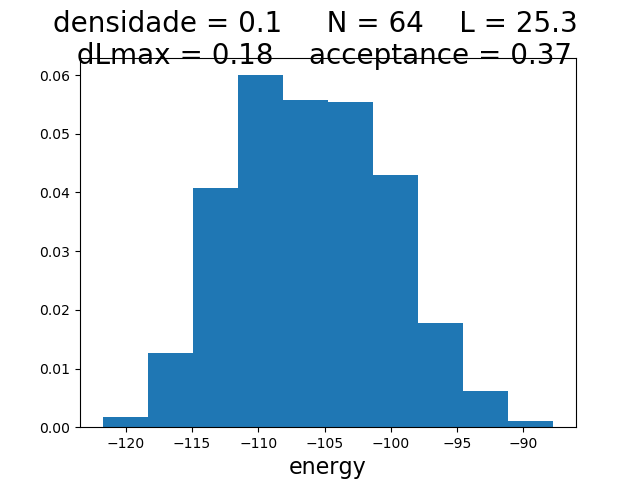
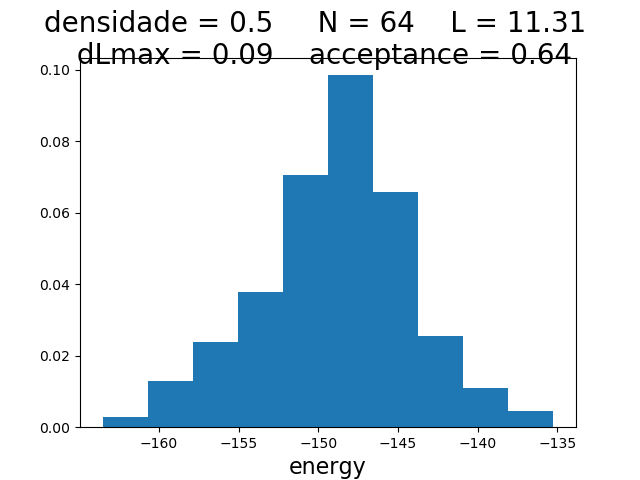
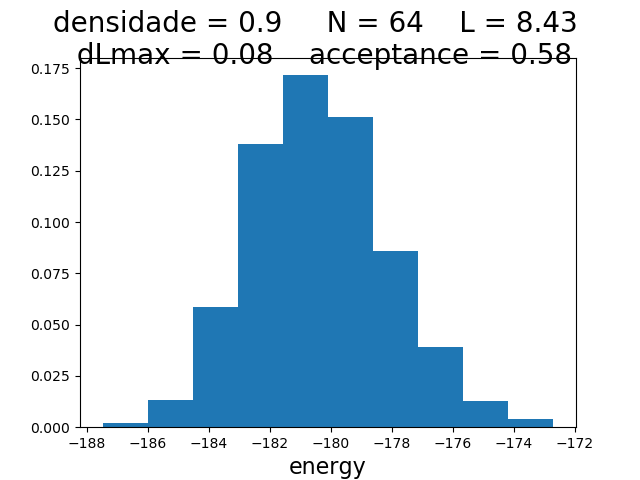
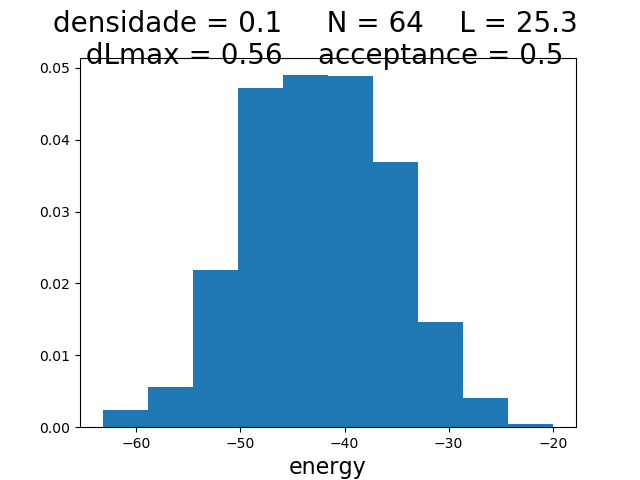
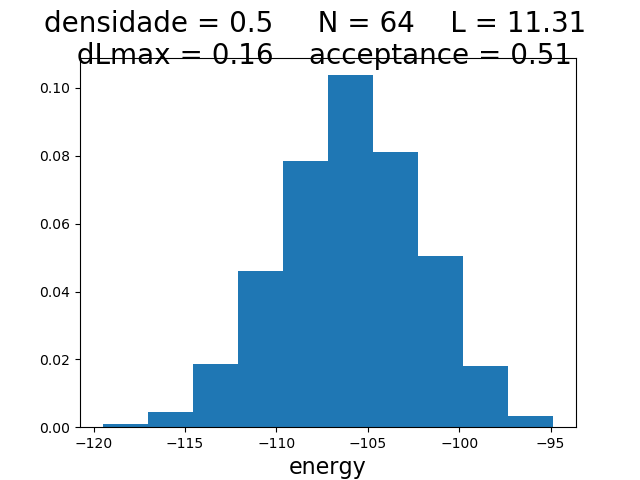
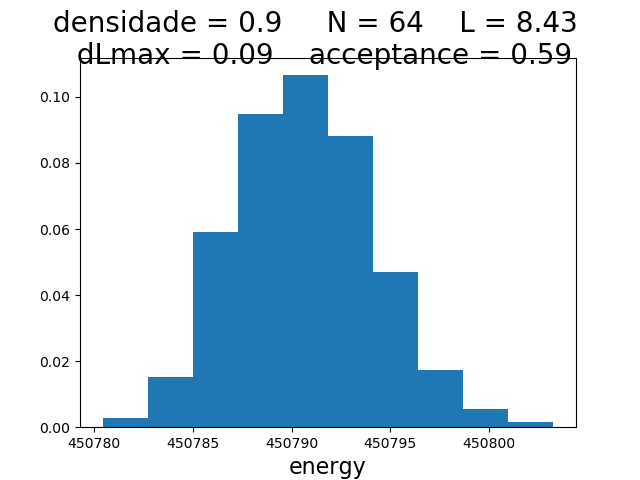
Histogramas
| Histogramas de energia a partir do MCS 6000 | ||
|---|---|---|
| Os gráficos da coluna mais a esquerda, central e mais a direita mostram respectivamente os hstogramas de energia para densidade 0.1, 0.5 e 0.9 respectivamente. Os histogramas mais acima têm temperatura 0.1, os da segunda linha têm temperatura 0.3 e os da terceira têm temperatura 0.5. | ||
Para checar se a energia segue a distribuição de poisson, olhamos os histogramas. Todos parecem seguir a distribuição de poisson correta, exceto no caso da densidade 0.9 e temperatura 0.5, onde ocorreu algum erro.
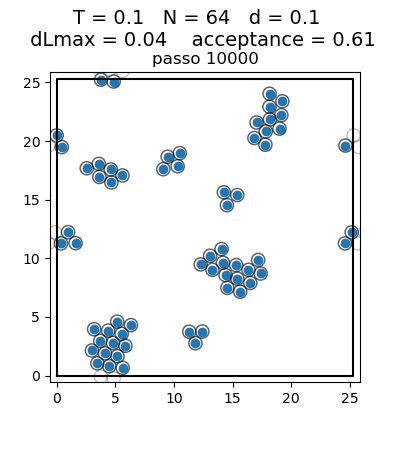
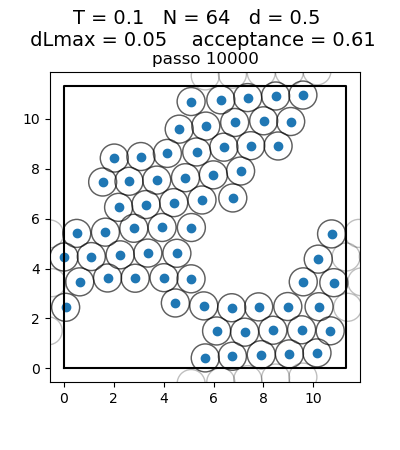
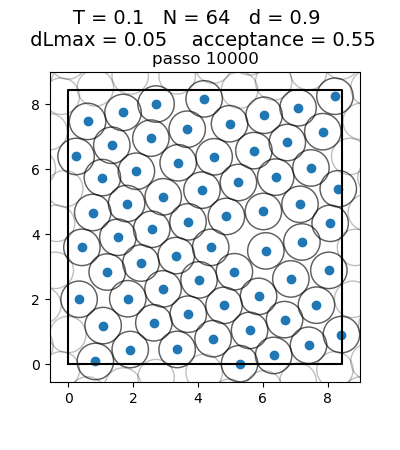
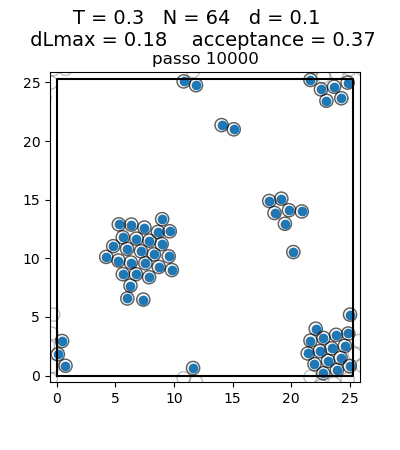
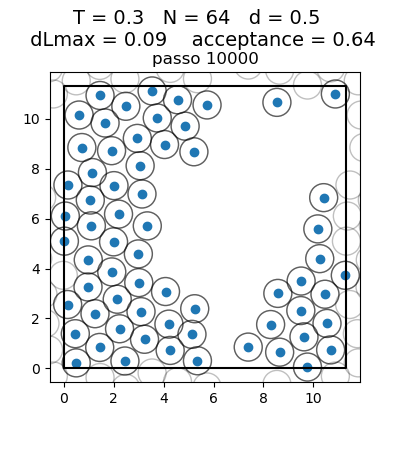
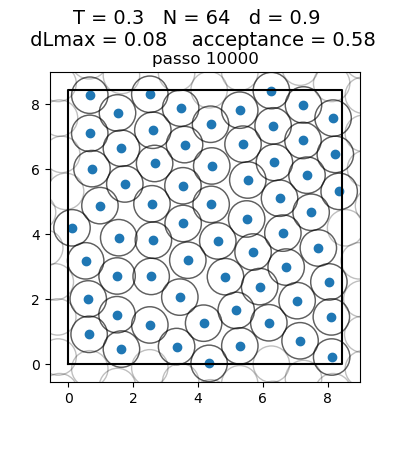
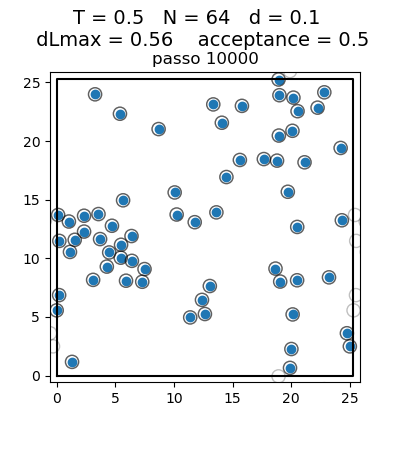
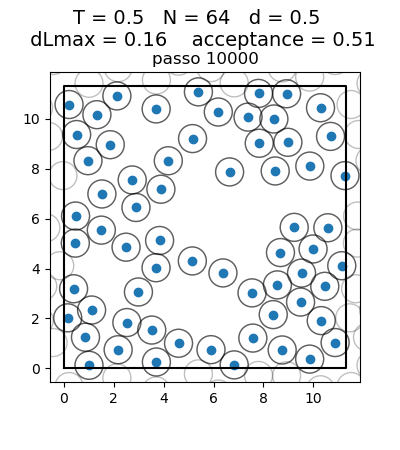
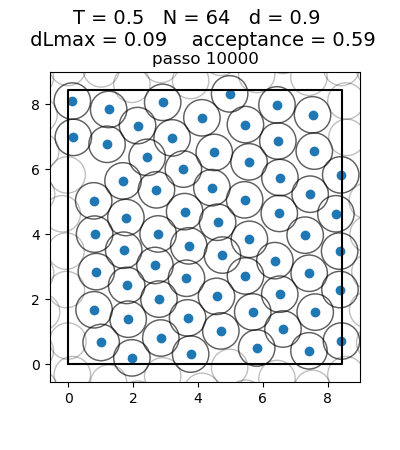
Posição final das Partículas
| Posição das partículas ao fim da simulação com diferentes densidades e temperaturas | ||
|---|---|---|
| Os gráficos da coluna mais a esquerda, central e mais a direita mostram respectivamente a posição final das partículas para densidade 0.1, 0.5 e 0.9 respectivamente. Os gráficos mais acima têm temperatura 0.1, os da segunda linha têm temperatura 0.3 e os da terceira têm temperatura 0.5. | ||
Transição de Fase
Olhando para a posição das partículas, podemos ver claramente uma transição de fase. Com temperaturas 0.1 e 0.3, para todas as densidades as partículas se encontram juntas, na configuração de menor energia, ou seja, numa fase líquida. Já para a temperatura 0.5 e densidades baixas (0.1 e 0.5) as partículas se encontram mais afastadas umas das outras, ou seja, numa fase gasosa. Com densidade 0.9, isso não ocorre, pois as partículas são forçadas a continuarem juntas, por não terem espaço para se afastar. Esse caso seria o de maior pressão. A transição de fase faz sentido com a diferença maior entre a temperatura 0.3 e 0.5 que aparece no gráfico da energia.
Código
Simulação
# -*- coding: utf-8 -*-
"""lennard_jones.ipynb
Automatically generated by Colaboratory.
Original file is located at
https://colab.research.google.com/drive/1jSPjBz0RXXWLEAzf3zcHgdlQpzjyO_ni
# Potencial de Lennard-Jones
# Monte Carlo no contínuo
#### Delete variables and import packages
"""
"""import sys
this = sys.modules[__name__]
for n in dir():
if n[0]!='_': delattr(this, n)"""
import numpy as np
from matplotlib import pyplot as plt
from matplotlib.animation import FuncAnimation
import random
from scipy.optimize import bisect as find_root
import time
"""## Simulação:
### Definições Numéricas:
"""
densidade = 0.1 #(2 ** (2/3)) * (3 ** (-1/2)) # partícula(s) por sigma^2 (partículas por area)
densidade_round = round(densidade, 2)
n_particles = 64
L = np.sqrt(n_particles / densidade) # unidade de sigma
print(L)
MCS = 10000
eps = 1
T = .1
k = 1
beta = 1/(k*T)
raio = 2**(1/6)
"""### Funcões:"""
def lennard_jones(r1, r2, L): # r1 e r2 em unidades de sigma (na vdd seria r1/sig e r2/sig)
dr = np.absolute(np.subtract(r2, r1))
for i in np.arange(2):
if dr[i] > 0.5*L:
dr[i] = L - dr[i]
r2 = (dr[0] ** 2) + (dr[1] ** 2)
sr2 = 1/r2
return 4 * (((sr2) ** 6) - ((sr2) ** 3))
def initial_energy(pos_particles, n_particles, L):
U0 = 0
for xxx in range(n_particles - 1):
for yyy in range(xxx+1, n_particles):
U0 += lennard_jones(pos_particles[:, xxx], pos_particles[:, yyy], L=L)
return U0
def energy_var(p, pos_particles, n_particles, L, new_pos=np.zeros(2)):
U1 = 0
U2 = 0
for i in range(n_particles - 1):
j = (i + p + 1) % n_particles # j vai ser todas as partículas
# exceto a partícula p (porque p interage com todas, menos com ela mesma)
U1 += lennard_jones(pos_particles[:, j], pos_particles[:, p], L=L)
U2 += lennard_jones(pos_particles[:, j], new_pos, L=L)
return U2 - U1
def main_loop(beta, n_particles, MCS, L, raio = 2 ** (1/6), time_check=2000):
pos_particles = L * np.random.random([2, n_particles])
# print(pos_particles)
# Random positions:
pos_MCS = np.zeros([MCS+1, 2, n_particles])
pos_particles = pos_particles
pos_MCS[0] = pos_particles
# Energy:
energy_arr = np.zeros(MCS+1)
energy = initial_energy(pos_particles, n_particles, L=L)
energy_arr[0] = energy
# Acceptance:
accepted = 0
acceptance_arr = np.zeros(MCS)
# dLmax:
dLmax = 2 ** (-5/6)
dLmax_arr = np.zeros(MCS + 1)
dLmax_arr[0] = dLmax
# Main Loop:
t0 = time.time()
for m in range(MCS):
for _ in range(n_particles):
# print()
p = random.randint(0, n_particles - 1)
# print(f'p = {p}')
# change position:
displacement = dLmax * ((2 * np.random.random([2])) - 1)
# print(f'displacement = {displacement}')
new_pos = pos_particles[:, p] + displacement
# print(f'new_pos = {new_pos}')
for i in range(2):
if new_pos[i] < 0:
new_pos[i] += L
elif new_pos[i] > L:
new_pos[i] -= L
# print(f'new_pos = {new_pos}')
dU = energy_var(p, pos_particles, n_particles, L, new_pos)
if dU <= 0:
pos_particles[0][p] = new_pos[0]
pos_particles[1][p] = new_pos[1]
energy += dU
accepted += 1
else:
if random.random() < np.exp(-beta * dU):
pos_particles[0][p] = new_pos[0]
pos_particles[1][p] = new_pos[1]
energy += dU
accepted += 1
pos_MCS[m+1] = pos_particles
energy_arr[m+1] = energy
acceptance = accepted / (n_particles*(m + 1))
acceptance_arr[m] = acceptance
if m > 100:
if acceptance < 0.3:
dLmax = dLmax * (acceptance / 0.3)
elif acceptance > 0.7:
dLmax = np.min([dLmax * (acceptance / 0.7), 2*raio])
dLmax_arr[m+1] = dLmax
if (m!=0) and (m % time_check == 0):
t1 = time.time()
dt = round((t1 - t0) / 60, 1)
min_to_go = round(dt * (MCS - m)/m, 1)
print(f'{m} steps done in t = {dt}min, {MCS - m} steps (~{min_to_go}min)to go')
return pos_MCS, energy_arr, acceptance_arr, dLmax_arr
def save_data(n_particles=n_particles, MCS=MCS, T=T, densidade=densidade, fixed_dLmax = False, dLmax=2 ** (-5/6)):
beta = 1 / (k*T)
L = np.sqrt(n_particles / densidade) # unidade de sigma
print(f'L={L}')
pos, energy, acceptance, dLmax_arr = main_loop(beta=beta, n_particles=n_particles,
MCS=MCS, L=L)
densidade_round = round(densidade, 3)
# save positions:
pos_reshaped = pos.reshape(pos.shape[0], -1)
np.savetxt(f"positions_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}.txt",
pos_reshaped)
# save energy:
np.savetxt(f"energy_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}.txt",
energy)
# save acceptance:
np.savetxt(f"acceptance_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}.txt",
acceptance)
# save acceptance:
np.savetxt(f"dLmax_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}.txt",
dLmax_arr)
return pos, energy, acceptance, dLmax_arr
def open_pos(file, n_particles):
loaded_pos = np.loadtxt(file)
load_original_pos = loaded_pos.reshape(loaded_pos.shape[0], loaded_pos.shape[1] // n_particles, n_particles)
return load_original_pos
"""#### Rodar para vários T e rô:"""
n_particles = 64
T_arr = np.array([0.1, 0.3, 0.5])
dens_arr = np.array([0.1, 0.5, 0.9])
MCS = 10000
for T in T_arr:
print()
print(f'T = {T}')
for densidade in dens_arr:
print(f'd = {densidade}')
pos, energy, acceptance, dLmax_arr = save_data(n_particles=n_particles, MCS=MCS, T=T, densidade=densidade)
#print(f'dLmax={dLmax} \n beta={beta} \n n_particles={n_particles} \n MCS={MCS} \n\n pos[0]: \n {pos[0]}')
print(f'acceptance = {acceptance[-1]}')
print(f'dLmax = {dLmax_arr[-1]}')
Gráficos
import numpy as np
from matplotlib import pyplot as plt
from matplotlib.animation import FuncAnimation
import random
from scipy.optimize import bisect as find_root
raio = 2 ** (1/6)
path = '/N32/'
save_path = '/N32/figures/'
dLmax = round(0.5*raio, 3)
def open_pos(file, n_particles=32):
loaded_pos = np.loadtxt(file)
load_original_pos = loaded_pos.reshape(loaded_pos.shape[0], loaded_pos.shape[1] // n_particles, n_particles)
return load_original_pos
"""
## Figuras
### Intial and final positions:
"""
def scatter_pos(pos, indices, T, L, dLmax, acceptance, circles=True, n_particles=64):
graphs = len(indices)
fig, axes = plt.subplots(1, graphs, figsize=(4 * graphs, 4.5))
fig.suptitle(
f'T = {T} N = {n_particles} d = {densidade_round} \n dLmax = {round(dLmax, 2)} acceptance = {acceptance}',
fontsize=14)
for j in range(graphs):
k = indices[j]
if graphs == 1:
ax = axes
else:
ax = axes[j]
if circles:
# draw the circles where they are
for i in range(n_particles):
c = plt.Circle([pos[k][0][i], pos[k][1][i]], raio / 2, fill=False, edgecolor='black', alpha=0.5)
ax.add_patch(c)
# draw the circle's projections:
for a in range(-1, 2):
for b in range(-1, 2):
for i in range(n_particles):
c = plt.Circle([pos[k][0][i] + a * L, pos[k][1][i] + b * L], raio / 2, fill=False,
edgecolor='black', alpha=0.25)
ax.add_patch(c)
points = ax.scatter(pos[k][0], pos[k][1])
margem = raio / 2
ax.plot([0, L, L, 0, 0], [0, 0, L, L, 0], color='black')
ax.axis([-margem, L + margem, -margem, L + margem], 'equal')
ax.grid(visible=False)
ax.set_aspect("equal")
ax.set_title(f'passo {k}')
if graphs == 1:
axes = ax
else:
axes[j] = ax
return fig, axes, points
n_particles = 64
T_arr = np.array([0.1, 0.3, 0.5])
dens_arr = np.array([0.1, 0.5, 0.9])
MCS = 10000
for T in T_arr:
print()
print(f'T = {T}')
for densidade in dens_arr:
print(f'd = {densidade}')
densidade_round = round(densidade, 3)
file_end = f"_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}"
file_name = f"{path}positions{file_end}.txt"
pos = open_pos(file_name, n_particles=n_particles)
acceptance = round(np.loadtxt(f'{path}acceptance{file_end}.txt')[-1], 2)
dLmax = round(np.loadtxt(f'{path}dLmax{file_end}.txt')[-1], 2)
L = np.sqrt(n_particles / densidade) # unidade de sigma
plot = scatter_pos(pos=pos, indices=[len(pos) - 1], T=T, L=L, dLmax=dLmax, acceptance=acceptance, circles=True)
plt.savefig(f"{save_path}first_last_positions{file_end}.png")
# plt.show()
plt.close()
"""### Energy:"""
def y_limits(energy, limites):
energy_hist_range = energy[int(round(0.2*len(energy), 0)):]
max_energy_range = max(energy_hist_range)
if max_energy_range > 100:
max_energy_range = 0
return [min([min(energy_hist_range)-1, limites[0]]), max([0, max_energy_range + 1, limites[1]])]
"""#### Energy/ acceptance / dLmax graphs:"""
for densidade in dens_arr:
print()
print(f'd = {densidade}')
densidade_round = round(densidade, 3)
fig_acc, ax_acc = plt.subplots(1, 1)
fig_dLmax, ax_dLmax = plt.subplots(1, 1)
fig, ax = plt.subplots(1, 1)
L = np.sqrt(n_particles / densidade) # unidade de sigma
titulo = f'densidade = {densidade_round} N = {n_particles} L = {round(L, 2)}'
fig.suptitle(titulo, fontsize=20)
fig_acc.suptitle(titulo, fontsize=20)
fig_dLmax.suptitle(titulo, fontsize=20)
limites = [0, 0]
for T in T_arr:
print(f'T = {T}')
file_end = f"_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}"
energy = np.loadtxt(f"{path}energy{file_end}.txt")
acc = np.loadtxt(f"{path}acceptance{file_end}.txt")
ax_acc.plot(np.arange(len(acc)), acc, label=f'T = {T}')
dLmax = np.loadtxt(f"{path}dLmax{file_end}.txt")
ax_dLmax.plot(np.arange(len(dLmax)), dLmax, label=f'T = {T}')
# Histogram:
fig2, ax2 = plt.subplots(1, 1)
fig2.suptitle(f'{titulo} \n dLmax = {round(dLmax[-1], 2)} acceptance = {round(acc[-1], 2)}', fontsize=20)
ax2.hist(energy[6000:], density=True)
ax2.set_xlabel('energy', fontsize=16)
# plt.show()
fig2.savefig(f"{save_path}energy_histogram{file_end}.png")
plt.close(fig2)
# Plot:
energy_range = energy
ax.plot(np.arange(len(energy_range)), energy_range, label=f'T = {T}')
limites = y_limits(energy, limites)
ax.set_ylim(limites)
ax.set_ylabel('U', fontsize=16)
ax.set_xlabel('tempo em MCS', fontsize=16)
ax.legend(fontsize=16)
ax_acc.set_ylabel('acceptance', fontsize=16)
ax_acc.set_xlabel('tempo em MCS', fontsize=16)
fig_acc.savefig(f"{save_path}acceptance_graph{file_end}.png")
plt.close(fig_acc)
ax_dLmax.set_ylabel('deslocamento máximo', fontsize=16)
ax_dLmax.set_xlabel('tempo em MCS', fontsize=16)
fig_dLmax.savefig(f"{save_path}dLmax_graph{file_end}.png")
plt.close(fig_dLmax)
# plt.show()
file_end2 = f"N{n_particles}_d{densidade_round}_MCS{MCS}"
fig.savefig(f"{save_path}energy_graph{file_end}.png")
plt.close(fig)
"""### Potencial Lennard Jones:"""
def lennard_jones1d(r):
sr6 = 1/(r ** 6)
return 4 * (((sr6) ** 2) - (sr6))
xx = np.linspace(0, 4, 100)
yy = lennard_jones1d(xx)
fig, ax = plt.subplots(1, 1, figsize=(10, 4))
ax.plot([-10, 10], [0, 0], color='black')
ax.plot(xx, yy)
ax.set_ylabel('U(r)')
ax.set_xlabel('r')
ax.set_ylim([-1.5, 5])
ax.set_xlim([0, 3])
# plt.show()
fig.savefig('lennard_jones_potential.png')
plt.close()
- ↑ J E Lennard-Jones 1931 Proc. Phys. Soc. 43 461





















![{\displaystyle U'(r')=4\epsilon \left[\left({\frac {\sigma }{r'}}\right)^{12}-\left({\frac {\sigma }{r'}}\right)^{6}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/7ef84cdd8901e3feec058279a5ff0f497a5fbf51)





![{\displaystyle U(r)=4\left[\left({\frac {1}{r}}\right)^{12}-\left({\frac {1}{r}}\right)^{6}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b6cc1c7619b98414369331ae46a5d8eab347f25c)