Otimizações: mudanças entre as edições
Sem resumo de edição |
Sem resumo de edição |
||
| Linha 122: | Linha 122: | ||
Deve-se inicialmente construir uma matriz indicando as células vizinhas de cada célula. | Deve-se inicialmente construir uma matriz indicando as células vizinhas de cada célula. | ||
< | <code><pre>//NC=numero linear de celulas - celulas totais=NC*NC | ||
//NC=numero linear de celulas - celulas totais=NC*NC | //cviz[i][j] = j-esima celula vizinha da i-esima celula | ||
//cviz[i][j] = j-esima celula vizinha da i-esima celula | void init_viz(int **cviz) | ||
void init_viz(int **cviz) | { | ||
{ | int i, j, ind, ip, im, jp, jm; | ||
for(i=0; i<NC; i++) | |||
for(j=0; j<NC; j++) | |||
{ | |||
ip=(i+1)%NC; im=(i+NC-1)%NC; //condicoes de contorno periodicas em i | |||
jp=(j+1)%NC; jm=(j+NC-1)%NC; //condicoes de contorno periodicas em j | |||
ind=i*NC+j; | |||
cviz[ind][0]=i*NC+jm; | |||
cviz[ind][1] =i*NC+jp; | |||
cviz[ind][2]=im*NC+jm; | |||
cviz[ind][3]=im*NC+jp; | |||
cviz[ind][4]=im*NC+j; //define vizinhos | |||
cviz[ind][5]=ip*NC+jm; | |||
cviz[ind][6]=ip*NC+jp; | |||
cviz[ind][7]=ip*NC+j; | |||
cviz[ind][8]=ind; | |||
} | |||
} | }</pre></code> | ||
</ | |||
Então, para cada passo de tempo, deve-se determinar quais partículas estão em quais células. | Então, para cada passo de tempo, deve-se determinar quais partículas estão em quais células. | ||
< | <code><pre> | ||
//ncell[i]=numero de particulas na celula i | //ncell[i]=numero de particulas na celula i | ||
//cell[i][j]=indice da j-esima particula na i-esima celula | //cell[i][j]=indice da j-esima particula na i-esima celula | ||
| Linha 174: | Linha 172: | ||
ncell[ind]++; | ncell[ind]++; | ||
} | } | ||
} | }</pre> | ||
< | </code> | ||
Calcula-se então as forças entre cada partícula e suas vizinhas. | Calcula-se então as forças entre cada partícula e suas vizinhas. | ||
< | <code><pre> | ||
void comp_forces(double *ffx, double *ffy, double *xx, double *yy, int **cell, int *ncell, int **cviz) | void comp_forces(double *ffx, double *ffy, double *xx, double *yy, int **cell, int *ncell, int **cviz) | ||
{ | { | ||
| Linha 217: | Linha 215: | ||
} | } | ||
} | } | ||
} | }</pre> | ||
< | </code> | ||
Em geral, o algoritmo fica: | Em geral, o algoritmo fica: | ||
< | <code><pre> | ||
init_viz(...) | init_viz(...) | ||
loopTemporal | loopTemporal | ||
| Linha 228: | Linha 226: | ||
integra(...) | integra(...) | ||
endLoopTemporal | endLoopTemporal | ||
< | </pre> | ||
</code> | |||
=== Simulação - Resultados === | === Simulação - Resultados === | ||
Edição das 16h32min de 8 de junho de 2016
Cerca de 95% do tempo de uma simulação é gasto com o cálculo das forças e a respectiva integração. Baseado nisso, criam-se métodos de otimização dos programas de simulação que podem acelerar de forma gigantesca (dependendo do número de partículas em interação) este processo. Claro que estes métodos por sua vez não são perfeitos, eles dependem de algumas características do sistema, no caso em questão, as otimizações chamadas Lista de Verlet e Células baseiam-se na simetria do potencial utilizado (Lennard-Jones) e no fato de ele provocar uma interação de curto alcance (em relação ao tamanho total do sistema), e são restritas a estas simetrias.
Em um programa sem otimização alguma, o calculo executado é a interação de cada partícula com as N-1 outras partículas, o que faz com que o tempo escale na ordem: ttot ~ N(N-1) ~ N2. Tomando um bom algoritmo de programação e levando em consideração a terceira lei de Newton, em que a Fi,j = -Fj,i, pode-se fazer metade das interações executadas antes, onde acaba-se com ttot ~ N(N-1)/2.
Lista de Verlet
Método
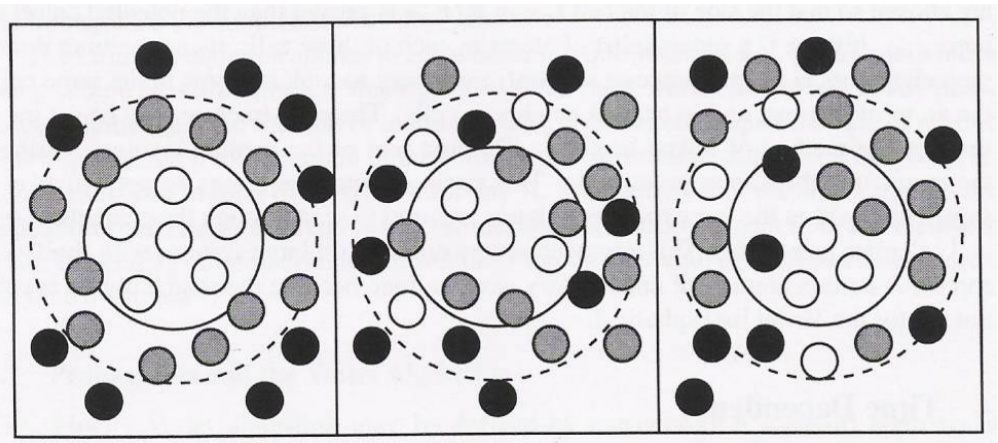
O que o algoritmo de Verlet propõe é que invés de se calcular a interação de uma partícula com as outras N-1, tome-se apenas uma lista das partículas mais próximas (vizinhas) e calcule-se a interação apenas com uma parte delas, é equivalente a propor que as outras partículas mais distantes não interajam significativamente com esta. Mas, dessa forma, como definir o que são as partículas vizinhas?
Para poder definir as partículas vizinhas, volta-se para a forma com que as partículas interagem entre si. Analisa-se então a Figura 1 que apresenta o potencial de interação (Potencial de Lennard-Jones), que tem características peculiares: é repulsivo para valores menores que 1.12 r/σ tendendo ao infinito para distâncias muito pequenas entre partículas (poderia se pensar como uma definição de "tamanho" para a partícula). Já para distâncias maiores que 1.12 r/σ entre as partículas, o potencial é atrativo e diminui com a distância, tendendo a zero para distâncias grandes. Perceba ainda que para distâncias não tão grandes assim (aproximadamente 2.5 vezes o "tamanho" da partícula, 2.5 r/σ) o potencial já é praticamente nulo em relação às forças de interação de distâncias próximas ao mínimo do potencial.
Neste ponto se percebe uma das grandes limitação da otimização proposta. Para uma simulação de interações de longo alcance, como objetos sob ação de campos gravitacionais por exemplo, esta técnica não se aplica de forma eficaz, pois ao se desprezar as pequenas interações das partículas distantes, pela grande quantidade dessas partículas, perde-se uma quantidade muito grande de informação que poderia agir sob a partícula de referência.
O procedimento é feito então definindo-se os vizinhos, para isso é feita uma análise das distâncias de cada partícula com as outras (processo de tempo ~ N2), a questão principal é saber quais partículas são vizinhas em todos os momentos e, principalmente, quais deixam de ser. É fácil perceber que se for necessário atualizar a lista em cada passo de tempo, se terá a mesma proporção temporal que o cálculo direto sem otimização (N2). Por este motivo, define-se um segundo círculo em torno da partícula, de raio Rv tal que Rv > Rc, onde Rv é o raio de vizinhos da partícula e Rc o raio crítico de interação (a força é calculada apenas para partículas dentro de Rc).
O algoritmo consiste em enumerar-se as partículas no interior de Rc e as partículas vizinhas entre Rc e Rv, para cada partícula i, e considerar apenas as no interior de Rc como partículas de interação. As partículas no segundo plano (Rc < rj < Rv) regulam quando deve-se atualizar a lista de partículas vizinhas, justamente para que não se necessite fazer este processo a cada iteração. O que se propõe então é que quando alguma partícula se movimentar uma distância maior que |Rc-Rv| a lista deve ser renovada, pois pode deixar de ser vizinha ou entrar na região de interação da partícula referencial. Neste ponto, deve-se pensar na questão de custo benefício pois quanto maiores forem os valores de Rc e Rv, menos vezes será necessário recalcular a lista de vizinhos (que demora ~ N2), em compensação, mais partículas serão consideradas e mais vezes deverá se calcular a força. Então, quais são os valores ideais de Rc e Rv?
Algoritmo
main()
{
inicia_posições();
inicia_velocidades();
define_lista_de_vizinhos();
for(tempo;tempo<tf)
{
testa_vizinhos();
calcula_força();
integra_movimento();
faz_medidas();
}
}
define_lista_de_vizinhos()
{
rc2 = rc*rc;
for(i=0;i<NP;i++)
{
nviz[i] = 0; k = 0;
for(j=0;j<NP;j++)
{
if(i==j) continue;
dx = x[i]-x[j];
dy = y[i]-y[j];
r2 = dx*dx+dy*dy;
if (r2 < rc2) { nviz[i]++; viz[i][k] = j; k++; }
pas_x[i] = x[i];
pas_y[i] = y[i];
}
}
No teste if (r2 < rc2) tem-se que se a distância entre i e j for menor que Rc, a partícula j é interagente e é a k-ésima vizinha de i. Os termos pas_x e pas_y gravam a posição para futuramente analisar o deslocamento e saber se atualiza-se a lista ou não.
testa_vizinhos()
{
rviz2 = (rv-rc)*(rv-rc);
for(i=0;i<NP;i++)
{
dx = x[i]-pas_x[i];
dy = y[i]-pas_y[i];
r2 = dx*dx+dy*dy;
if (r2 > rviz2) define_lista_de_vizinhos();
}
}
No último if analisa-se se alguma partícula andou mais que |Rc-Rv| e atualiza-se ou não a lista.
Simulação - Resultados
Para encontrar os valores ideais de Rc e Rv deve-se testá-los para diferentes parâmetros e os comparar.
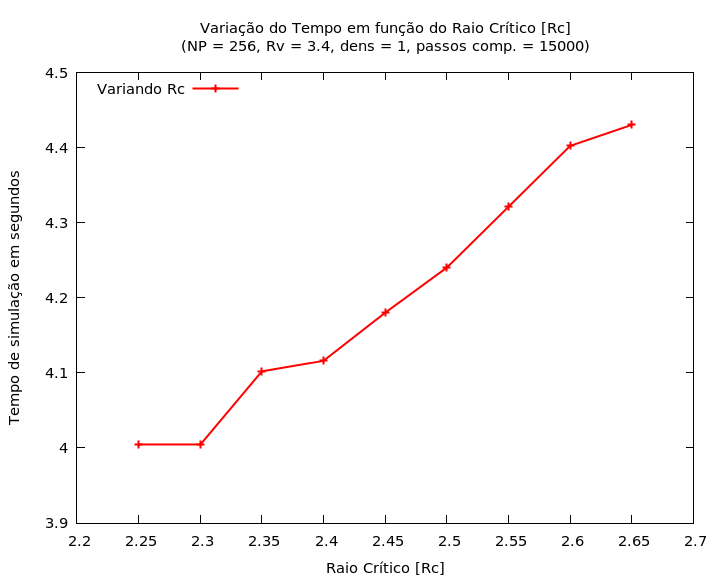
Na Figura 3 pode-se ver os resultados da variação do raio crítico Rc para uma caixa de 256 partículas com densidade igual a 1, 15000 passos computacionais e parâmetro Rv = 3.4 (logo mais se tornará explicito o porque deste valor para Rv). Há de se notar que o raio crítico não possui uma grande liberdade de variação, visto que se muito pequeno, despreza interações relevantes no cálculo, e, se muito grande, proporciona uma faixa muito estreita para partículas vizinhas (isto para Rv fixo). No gráfico em questão, lê-se tempo de simulação por tamanho de raio crítico, e observa-se ainda que a variação relativa no tempo é muito pequena (em termos de tempos totais de simulação), de apenas 0.4 segundos. Assim, assume-se como um valor razoável Rc = 2.5 r/σ, o valor intermediário dos pontos testados.
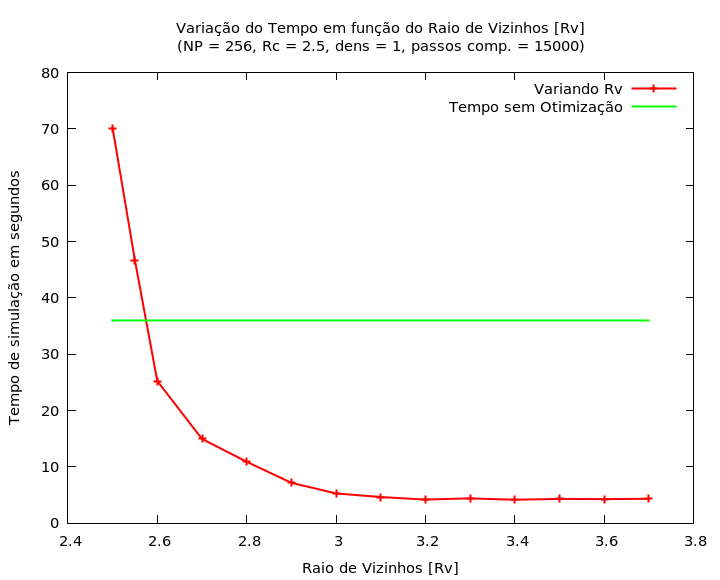
Para o raio de vizinhos, Rv, toma-se a mesma abordagem e obtém-se resultados muito mais significativos. O que se faz é variar o valor de Rv e acompanhar algum parâmetro de medida (no caso os gráficos de G de R) para garantir que o sistema ainda represente uma situação física. Na Figura 4 pode-se observar os efeitos de variação do Rv em termos de r/σ sob o tempo de simulação para um sistema com 256 partículas, Rc = 2.5, densidade = 1 e 15000 passos computacionais. Percebe-se que para valores muito próximos de Rc, a faixa de vizinhos é muito estreita e o tempo de simulação praticamente dobra, em relação ao tempo sem otimizações, e conforme aumenta-se o raio, o tempo diminui no que aparenta ser de forma exponencial até um valor constante próximo de 4.3 segundos para 3.5 < Rv < 3.7. A partir de Rv > 3.7 o gráfico de G de R se distorce e o sistema deixa de ser físico. Desta forma, assume-se Rv = 3.4 (meio do intervalo) como um tamanho razoável para o raio das partículas vizinhas.
Por fim então comparam-se os tempos de medidas para variação no número de partículas, onde espera-se que para uma grande quantidade de partículas os efeitos da otimização sejam extremamente significantes.
Para a otimização da Lista de Verlet, por mais que ainda se tenham alguns cálculos de ordem N2, espera-se que sejam muito menos frequentes que no cálculo não otimizado.
Onde o parâmetro c tem relação com a forma em que o programa foi escrito, podendo melhorar ou piorar o desempenho a partir de algumas especificidades do programador, N é o número de partículas, nv é o número de vizinhos de todas as partículas e α é um parâmetro relacionado com a difusão das partículas de um sistema uma vez que quanto mais as partículas andarem, mais terão de ser atualizadas suas listas. Assim, o primeiro termo a direita da Equação 2 remete ao cálculo das forças e o segundo à atualização das listas de vizinhos.
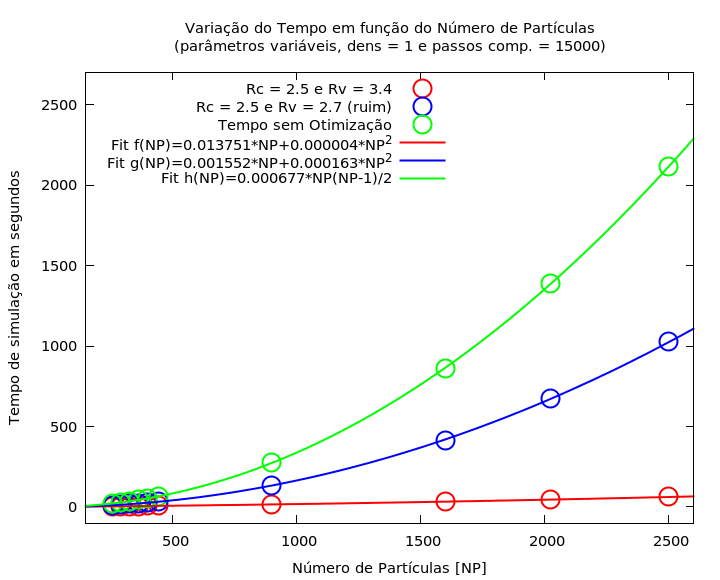
Na Figura 5 pode-se analisar então o comportamento do tempo de simulação em relação à quantidade de partículas no sistema, onde os círculos aberto representam as simulações para o programa sem otimização, o programa otimizado com parâmetros ruins e o programa otimizado com parâmetros ótimos; já os traços contínuos representam os ajustes lineares das Equações 1 e 2 para os respectivos programas. O fator flagrante que se marca no gráfico é o fato de que as simulações otimizadas, mesmo para parâmetros ruins, são bem mais rápidas que as efetuadas sem qualquer otimização. Além disso, a partir dos ajustes, obtém-se os coeficientes, já discutidos, das equações de tempo de simulação onde pode-se notar que quando tomam-se parâmetros ruins para a otimização de Verlet (no caso Rv), o programa acaba tendo uma dependência muito maior no parâmetro quadrático (aproximadamente 40x maior) e consequentemente isso afeta de forma drástica o tempo de simulação. No caso particular abordado, em que o sistema tem densidade 1, a difusão das partículas é quase inexistente, isso permite que o fit com parâmetros ótimos quase não possua dependência quadrática, não sendo esta uma regra geral de bons parâmetros. Marcam-se então as diferenças para 2500 partículas, em que os tempos sem otimização, com otimização ruim e com otimização ótima são, respectivamente, τSem-Otim = 2115.340s, τVerl-ruim = 1025.992s e τVerl-bom = 62.564s.
Desta forma pode se ver a grande importância das otimizações para cálculos de simulações numéricas e, mais que isso, o bom acerto dos parâmetros de otimização que são fortemente controlados pelo tipo de interação utilizada e por outros parâmetros do sistema. É importante também lembrar da limitação do método quanto a interações de longo alcance e de potenciais não diretamente limitados.
Otimização por células
Método
Neste método divide-se o espaço em células (normalmente, mas não necessáriamente quadradas) e calcula-se, para cada partícula, somente a interação com aquelas que se encontram em uma célula vizinha à sua. Desta forma, ao invés de o tempo computacional escalar com N2, escala segundo a lei:
onde a e b são constantes, n é o número de partículas nas células vizinhas e N é o número total de partículas. Assim como na Lista de Verlet, o método possui muitas operações com matrizes e, por isso, torna-se mais eficiente somente para valores de N grandes.
Em geral, para uma célula Ci,j, considera-se vizinhas as células: Ci,j, Ci+/-1,j, Ci,j+/-1, Ci+/-1,j+/-1, Ci+1,j-1 e Ci-1,j+1, totalizando nove. É importante ressaltar que o tamanho linear de uma célula deve, pelo menos, conter o intervalo em que a interação entre duas partículas é relevante.
Algoritmo
Deve-se inicialmente construir uma matriz indicando as células vizinhas de cada célula.
//NC=numero linear de celulas - celulas totais=NC*NC
//cviz[i][j] = j-esima celula vizinha da i-esima celula
void init_viz(int **cviz)
{
int i, j, ind, ip, im, jp, jm;
for(i=0; i<NC; i++)
for(j=0; j<NC; j++)
{
ip=(i+1)%NC; im=(i+NC-1)%NC; //condicoes de contorno periodicas em i
jp=(j+1)%NC; jm=(j+NC-1)%NC; //condicoes de contorno periodicas em j
ind=i*NC+j;
cviz[ind][0]=i*NC+jm;
cviz[ind][1] =i*NC+jp;
cviz[ind][2]=im*NC+jm;
cviz[ind][3]=im*NC+jp;
cviz[ind][4]=im*NC+j; //define vizinhos
cviz[ind][5]=ip*NC+jm;
cviz[ind][6]=ip*NC+jp;
cviz[ind][7]=ip*NC+j;
cviz[ind][8]=ind;
}
}Então, para cada passo de tempo, deve-se determinar quais partículas estão em quais células.
//ncell[i]=numero de particulas na celula i
//cell[i][j]=indice da j-esima particula na i-esima celula
void celula(double *xx, double *yy, int *ncell, int **cell)
{
int i, cx, cy, ind=0;
double xtmp, ytmp;
for(i=0; i<NC*NC; i++)
ncell[i]=0;
for(i=0; i<N; i++)
{
xtmp=fmod(xx[i], L); ytmp=fmod(yy[i], L); //PBC
if(xtmp<0) xtmp+=L;
if(ytmp<0) ytmp+=L;
cx=(int)(xtmp/(1.*L/NC)); //determina a celula da particula
cy=(int)(ytmp/(1.*L/NC));
ind=cx*NC + cy;
cell[ind][ncell[ind]]=i;
ncell[ind]++;
}
}
Calcula-se então as forças entre cada partícula e suas vizinhas.
void comp_forces(double *ffx, double *ffy, double *xx, double *yy, int **cell, int *ncell, int **cviz)
{
int i, j, k, cx, cy;
double x1, y1, x2, y2, dxx, dyy, r2, r2i, r6, ff;
int numPart=0, celulaVizinha;
for(i=0; i<N; i++)
{
ffx[i]=0.; //zera as forcas
ffy[i]=0.;
}
int ind, p2;
double xtmp, ytmp;
for(i=0; i<N; i++) //para cada particula
{
x1=xx[i]; y1=yy[i];
xtmp=fmod(x1, L); ytmp=fmod(y1, L); //PBC
if(xtmp<0) xtmp+=L;
if(ytmp<0) ytmp+=L;
cx=(int)(xtmp/(1.*L/NC));
cy=(int)(ytmp/(1.*L/NC));
ind=cx*NC+cy; //determina indice da celula
for(j=0; j<9; j++) //para cada celula vizinha
{
celulaVizinha=cviz[ind][j];
numPart=ncell[celulaVizinha];
for(k=0; k<numPart; k++) //para cada particula na celula vizinha
{
p2=cell[celulaVizinha][k];
if(p2!=i)
{
\\calculos usuais das forcas
}
}
}
}
}
Em geral, o algoritmo fica:
init_viz(...)
loopTemporal
celula(...)
comp_forces(...)
integra(...)
endLoopTemporal