Medidas estáticas: mudanças entre as edições
Sem resumo de edição |
|||
| (43 revisões intermediárias pelo mesmo usuário não estão sendo mostradas) | |||
| Linha 1: | Linha 1: | ||
Em dinâmica molecular, medidas estáticas são medidas | Em dinâmica molecular, medidas estáticas são medidas realizadas em um passo específico de tempo da simulação. | ||
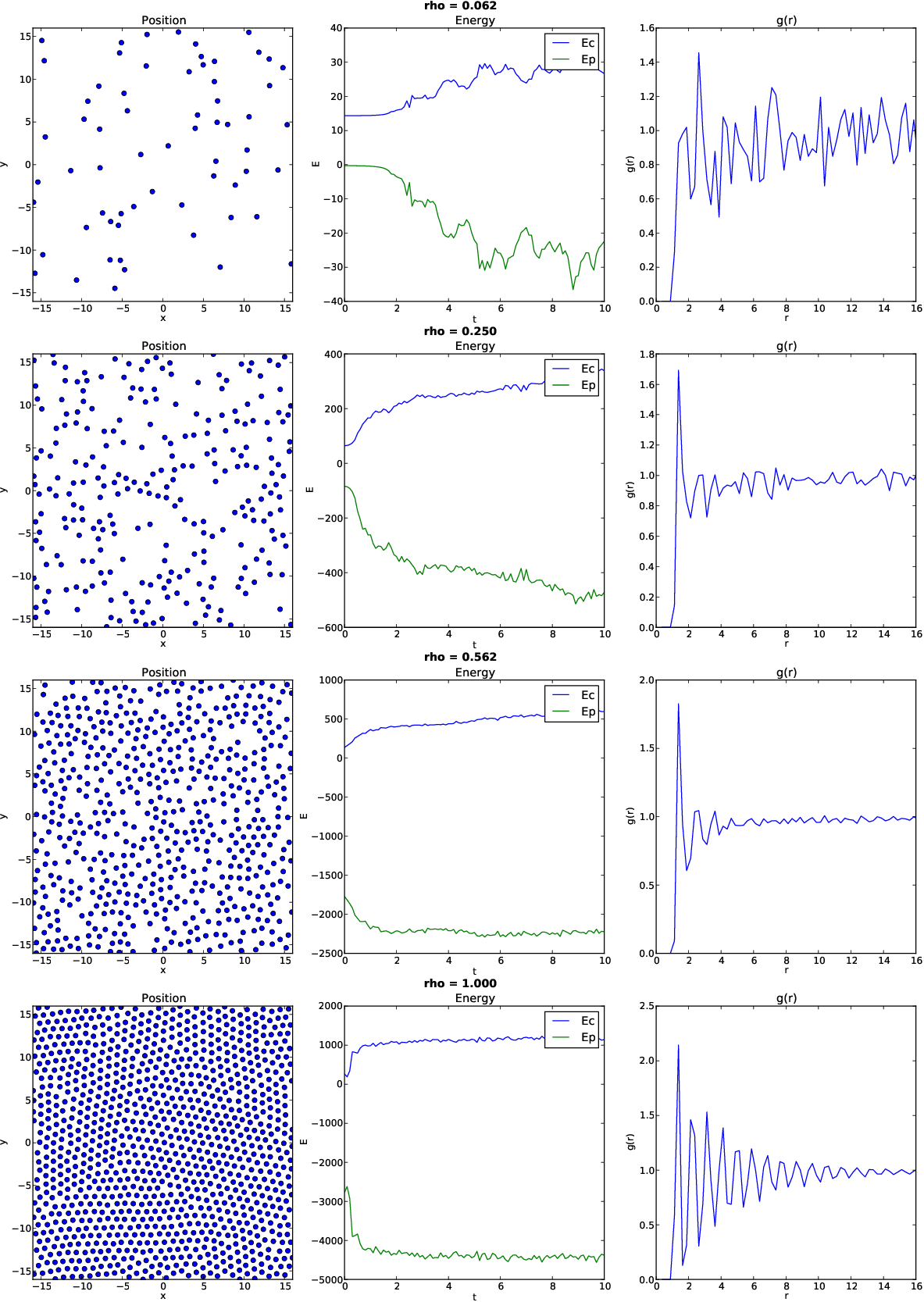
No estudo do agrupamento de pontos equidistantes em um espaço 2D, é possível provar matematicamente que o formato formado pelos pontos que maximiza a utilização do espaço é o '''padrão hexagonal'''. Para dinâmicas moleculares com potenciais de Lennard-Jones com densidade suficientemente alta (rho ~) é possível observar que o padrão formado após o relaxamento (tempo suficiente para a rede se estabilizar) é de fato o padrão hexagonal. | =Pair Distribution Function= | ||
[[Image:Gr.png|thumb|300px|right|Representação do cálculo numérico de <math>g(r)</math>;]] | |||
A ''Pair Distribution Function'' , ou "<math>g(r)</math>", é uma função que estima o quão provável é encontrar duas partículas a uma distância <math>r</math> dentro de um sistema de várias partículas. | |||
Em um sistema de <math>N</math> partículas, o <math>g(r)</math> é definido como a média do número de partículas a uma distância <math>r</math>: | |||
<center><math> | |||
g(r)=\frac{V}{N^2}\langle\sum_{i=1}^N\sum_{j\neq i}^N\delta(r-|r_i-r_j|) \rangle | |||
</math></center> | |||
Numéricamente pode ser interpretado como a média do número de pares de partículas a uma distância entre <math>r</math> e <math>r+\Delta r</math> pesado pelo volume/área desta região. | |||
<math> | |||
g(r,\Delta r)=\frac{V_t}{N^2}\sum_{i=1}^N\sum_{j\neq i}^N\left[\frac{rect\left(\frac{r-|r_i-r_j|}{\Delta r}\right)}{V\left(r+\frac{\Delta r}{2}\right)-V\left(r-\frac{\Delta r}{2}\right)}\right] | |||
</math></center> | |||
Onde <math>V_t</math> é o/a volume/área total e <math>rect</math> é a função retangular. | |||
Em resumo, o <math>g(r,\Delta r)</math> é a média dos [[Histogramas_e_Densidade_de_Probabilidade|histogramas]] do número de partículas em um ''bin'' de largura <math>\Delta r</math> a uma <math>r</math> feitos para cada partícula no sistema pesado pelo volume/área deste ''bin''. | |||
==== Construção do Código ==== | |||
==== Resultados ==== | |||
[[Image:GR-D.png|400px|Resultado do cálculo de <math>g(r)</math>;]] | |||
=Referências= | |||
*Frenkel, Daan and Smit, Berend (2001). ''Understanding Molecular Simulation''. Academic Press. | |||
= Psi 6 (<math> \psi_6 </math>)= | |||
No estudo do agrupamento de pontos equidistantes em um espaço 2D, é possível provar matematicamente que o formato formado pelos pontos que maximiza a utilização do espaço é o '''padrão hexagonal'''. Para dinâmicas moleculares com potenciais de Lennard-Jones com densidade suficientemente alta (rho ~0.8) é possível observar que o padrão formado após o relaxamento (tempo suficiente para a rede se estabilizar) é de fato o padrão hexagonal. | |||
O psi 6 é uma análise de o quão hexagonal um padrão de posições está em um certo tempo da simulação. É possível associar a cada partícula um valor que varia entre -1 e 1 da hexagonalidade do padrão de posições formado por ele e seus primeiros vizinhos (conjunto de partículas mais próximas). Com essa medida é possível quantificar diferentes regiões da "caixa" em que as partículas estão localizadas e então localizar possíveis "defeitos" no padrão hexagonal. | O psi 6 é uma análise de o quão hexagonal um padrão de posições está em um certo tempo da simulação. É possível associar a cada partícula um valor que varia entre -1 e 1 da hexagonalidade do padrão de posições formado por ele e seus primeiros vizinhos (conjunto de partículas mais próximas). Com essa medida é possível quantificar diferentes regiões da "caixa" em que as partículas estão localizadas e então localizar possíveis "defeitos" no padrão hexagonal. | ||
| Linha 15: | Linha 50: | ||
== Implementação Computacional == | == Implementação Computacional == | ||
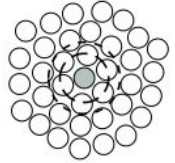
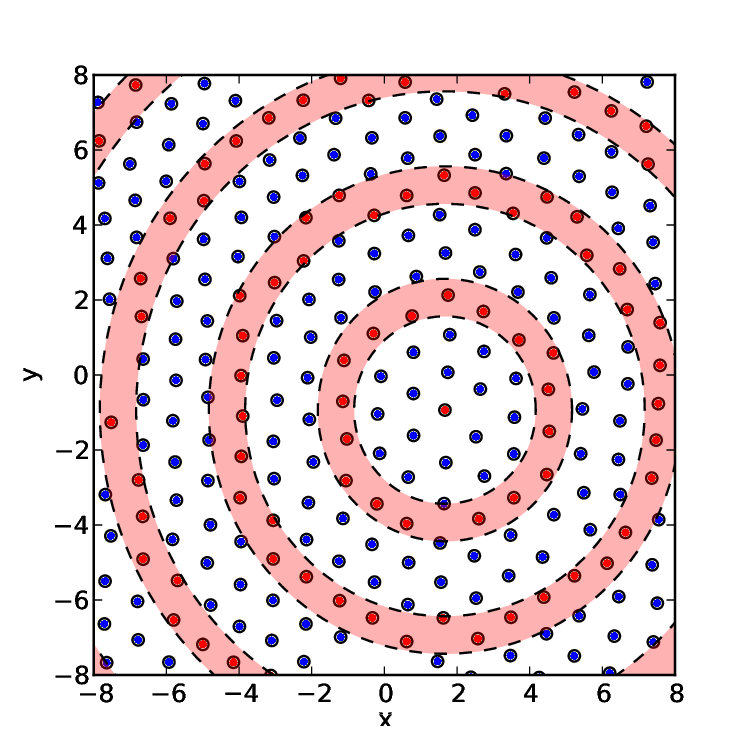
[[Image:Neighbors.jpg|thumb|200px|right|Definição de vizinhança:O primeiro círculo pontilhado delimita os primeiros vizinhos, o segundo círculo pontilhado delimita os segundos vizinhos;]] | |||
Pode-se separar a implementação computacional em dois procedimentos necesssários: Encontrar os primeiros vizinhos para cada partícula e então calcular, de fato, o valor de <math>\psi_6</math> | Pode-se separar a implementação computacional em dois procedimentos necesssários: Encontrar os primeiros vizinhos para cada partícula e então calcular, de fato, o valor de <math>\psi_6</math>. | ||
=== Encontrando vizinhos === | === Encontrando vizinhos === | ||
Por definição, os primeiros vizinhos de uma partícula são aquelas partículas que estão presentes em um anel mais próximo dela, como mostra a | Por definição, os primeiros vizinhos de uma partícula são aquelas partículas que estão presentes em um anel mais próximo dela, como mostra a figura ao lado. | ||
O problema de encontrar primeiros vizinhos é um problema bastante discutido em teoria da computação e diversos métodos foram desenvolvidos para efetuar esta tarefa. Desta forma a maioria dos métodos otimizados são de difícil implementação e então não serão tratados nesse verbete. | |||
O | O método aqui citado é pouco otimizado, pois utiliza-se um for em <math>N^2</math>, porém de implementação razoavelmente simples. O método consiste em encontrar as partículas mais próximas da partícula-teste (partícula em que busca-se calcular o valor de <math>\psi_6</math> baseando-se em outro valor já calculado, a medida do g(r), desta forma o valor de <math>\psi_6</math> resultará em um valor razoável ( >0.8 ) se os vizinhos dentro do raio medido no g(r) são de fato 6 e estão localizados em um padrão quase-hexagonal em torno da partícula-teste. | ||
Para encontrar os primeiros vizinhos baseando-se no g(r), precisa-se observar qual a medida do primeiro pico no gráfico do g(r) e então definir este como uma distância de corte para a partícular ser ou não vizinha (isto é, um circulo com o raio da distância de corte ao redor da partícula-teste de forma que outras partículas dentro deste círculo são suas vizinhas). Neste caso, definem-se três vetores: "neighborsX" (que guarda a posição X do vizinho), "neighborsY"(que guarda a posição Y do vizinho) e "dNeighbors"(que guarda a distância do vizinho)[É recomendado definir estes vetores com tamanho pelo menos de 10, pois é possível que sejam encontrados mais de 6 vizinhos, para isso existe a variável de controle "k" que guarda quantos vizinhos cada partícula tem] e então faz-se o for em <math>N^2</math> buscando-se as distância menores que o raio de corte: | |||
<pre> | <pre> | ||
if(i!=j){ | for(i=0;i<NP;i++){ | ||
k=0; | |||
xOrg=xx[i]; | |||
yOrg=yy[i]; | |||
for(j=0;j<NP;j++){ | |||
x2=xx[j]; | |||
y2=yy[j]; | |||
if(i!=j){ | |||
deltaX=fabs(x2-x1); | |||
deltaY=fabs(y2-y1); | |||
//Condições de contorno | |||
deltaX=deltaX-rint(deltaX/Lx)*Lx; | |||
deltaY=deltaY-rint(deltaY/Ly)*Ly; | |||
// --- // | |||
d=sqrt((deltaX*deltaX)+(deltaY*deltaY)); | |||
if(d<radiusLimit) | |||
{ | |||
neighborsX[k]=x2; | |||
neighborsY[k]=y2; | |||
dNeighbors[k]=d; | |||
k+=1; | |||
} | } | ||
} | } | ||
} | |||
</pre> | </pre> | ||
Portanto após esse algoritmo, temos guardadas as posições das | Portanto após esse algoritmo, temos guardadas as posições das partículas mais próximas. É importante ressaltar que o "for" em partícula-teste ainda não foi fechado, e ainda dentro deste mesmo loop serão calculados os valores de <math> \psi_6 </math>. | ||
=== Calculando o Psi 6 === | === Calculando o Psi 6 === | ||
| Linha 104: | Linha 108: | ||
<pre> | <pre> | ||
for(j=0;j< | for(j=0;j<k;j++){ | ||
delX=neighborsX[j]-xx[i]; | delX=neighborsX[j]-xx[i]; | ||
delY=neighborsY[j]-yy[i]; | delY=neighborsY[j]-yy[i]; | ||
| Linha 120: | Linha 124: | ||
</pre> | </pre> | ||
Agora somente precisa-se realizar um algoritmo que ordene o vetor "angle" do menor para o maior ângulo, desta forma é possível calcular os <math> \theta_i </math> | Agora somente precisa-se realizar um algoritmo que ordene o vetor "angle" do menor para o maior ângulo, utilizando algoritmos de ordenamento (bubblesort, quicksort, etc) desta forma é possível calcular os <math> \theta_i </math>. | ||
Feito o ordenamento do vetor, basta calcular a média do valor do <math> \psi_6 </math> de cada vizinho. Para isto, precisamos calcular os valores de <math> \theta_i </math> em função dos <math> \phi </math>, basta realizar a subtração do próximo vizinho no vetor pelo valor do ângulo do vizinho atual, dessa forma: <math> \theta_i = \psi_{i+1} - \psi_i </math> com exceção do último vizinho, que será o ângulo dele menos o do primeiro, desta forma a implementação fica: | Feito o ordenamento do vetor, basta calcular a média do valor do <math> \psi_6 </math> de cada vizinho. Para isto, precisamos calcular os valores de <math> \theta_i </math> em função dos <math> \phi </math>, basta realizar a subtração do próximo vizinho no vetor pelo valor do ângulo do vizinho atual, dessa forma: <math> \theta_i = \psi_{i+1} - \psi_i </math> com exceção do último vizinho, que será o ângulo dele menos o do primeiro, desta forma a implementação fica: | ||
| Linha 128: | Linha 131: | ||
firstAngle=angle[0]; | firstAngle=angle[0]; | ||
for(j=0;j< | for(j=0;j<k-1;j++){ | ||
angle[j]=cos(6.*(angle[j+1]-angle[j])); | angle[j]=cos(6.*(angle[j+1]-angle[j])); | ||
} | } | ||
angle[ | angle[k-1]=cos(6.*(firstAngle+2*3.1415-angle[k-1])); | ||
for(j=0;j< | for(j=0;j<k;j++){ | ||
psix[i]+=angle[j]/6.; | psix[i]+=angle[j]/6.; | ||
} | } | ||
| Linha 139: | Linha 142: | ||
</pre> | </pre> | ||
E então está calculado o valor de <math> \psi_6 </math>, para facilitar a implementação é recomendado que este algoritmo seja uma função dentro de seu código e desta forma retorne o | E então está calculado o valor de <math> \psi_6 </math>, para facilitar a implementação é recomendado que este algoritmo seja uma função dentro de seu código e desta forma retorne o vetor "psix". | ||
= | === Exemplos === | ||
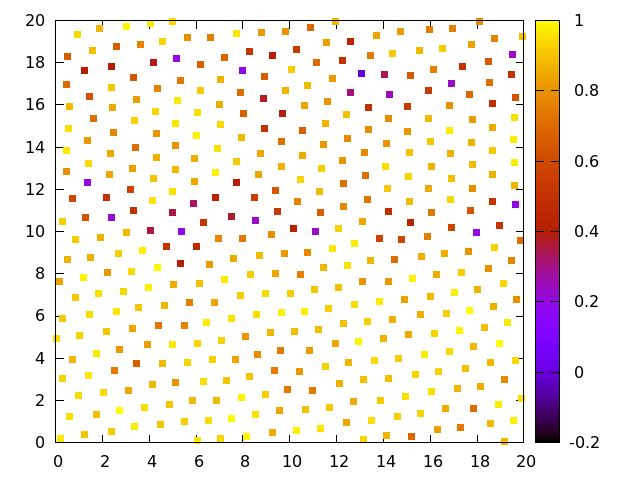
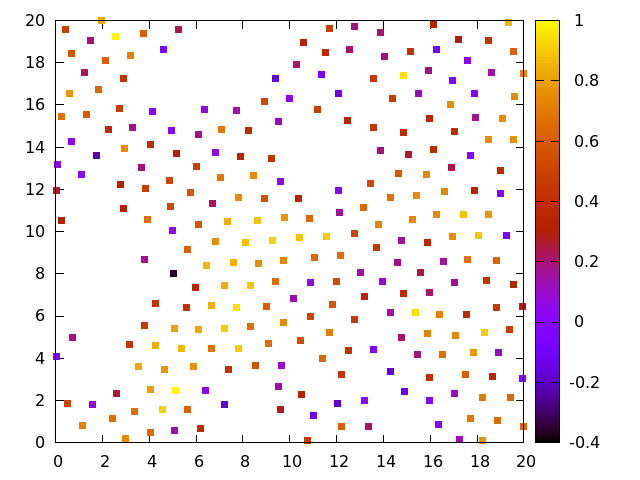
Para o caso de um potencial de Lennard-Jones, é possível observar resultados de mapas de <math> \psi_6 </math>, isto é, um mapa que mostra para cada partícula o seu valor da medida, da seguinte forma para cada valor de rho: | |||
[[Image: | [[Image:Psix_1.png|thumb|400px|right|Resultados do valor de <math>\psi_6</math> (em cores) para o caso de um potencial de Lennard Jones para <math> \rho=1.000 </math>]] | ||
[[Image:Psix_0360.png|thumb|400px|left|Resultados do valor de <math>\psi_6</math> (em cores) para o caso de um potencial de Lennard Jones para <math> \rho=0.360 </math>.]] | |||
[[Image:Psix_0640.png|thumb|400px|center|Resultados do valor de <math>\psi_6</math> (em cores) para o caso de um potencial de Lennard Jones para <math> \rho=0.640 </math>]] | |||
== Aplicações em outras áreas == | |||
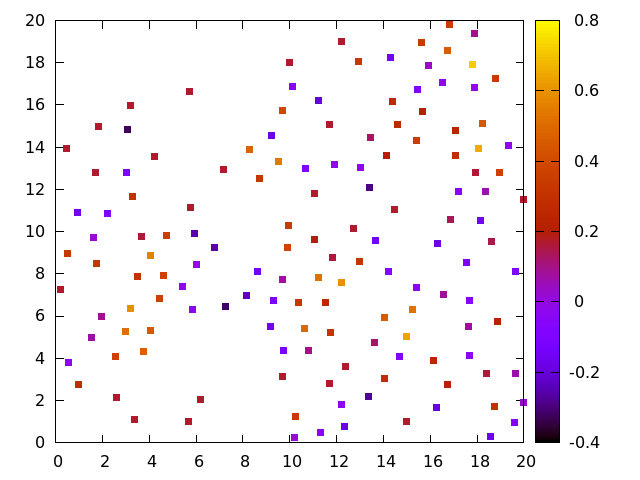
[[Image:Celulasdegrade.png|thumb|300px|right|Mapa neural de células de grade em que o valor de <math> \psi_6 </math> equivale 0,99.]] | |||
No estudo de como animais processam a sua posição em seu cerébro, são observadas as chamadas "Células de grade", neurônios que disparam em certos locais específicos do espaço e tendem a formar padrões de grade (hexagonal). Em posse das posições de disparo desses neurônios é possível utilizar a medida do <math> \psi_6 </math> para determinar a "hexagonalidade" dos disparos, podendo-se realizar estudos da influência desse formato na interpretação geoespacial do animal. | |||
Em | |||
Edição atual tal como às 18h17min de 1 de agosto de 2016
Em dinâmica molecular, medidas estáticas são medidas realizadas em um passo específico de tempo da simulação.
Pair Distribution Function
A Pair Distribution Function , ou "", é uma função que estima o quão provável é encontrar duas partículas a uma distância dentro de um sistema de várias partículas.
Em um sistema de partículas, o é definido como a média do número de partículas a uma distância :
Numéricamente pode ser interpretado como a média do número de pares de partículas a uma distância entre e pesado pelo volume/área desta região.
Onde é o/a volume/área total e é a função retangular.
Em resumo, o é a média dos histogramas do número de partículas em um bin de largura a uma feitos para cada partícula no sistema pesado pelo volume/área deste bin.
Construção do Código
Resultados
Referências
- Frenkel, Daan and Smit, Berend (2001). Understanding Molecular Simulation. Academic Press.
Psi 6 ()
No estudo do agrupamento de pontos equidistantes em um espaço 2D, é possível provar matematicamente que o formato formado pelos pontos que maximiza a utilização do espaço é o padrão hexagonal. Para dinâmicas moleculares com potenciais de Lennard-Jones com densidade suficientemente alta (rho ~0.8) é possível observar que o padrão formado após o relaxamento (tempo suficiente para a rede se estabilizar) é de fato o padrão hexagonal.
O psi 6 é uma análise de o quão hexagonal um padrão de posições está em um certo tempo da simulação. É possível associar a cada partícula um valor que varia entre -1 e 1 da hexagonalidade do padrão de posições formado por ele e seus primeiros vizinhos (conjunto de partículas mais próximas). Com essa medida é possível quantificar diferentes regiões da "caixa" em que as partículas estão localizadas e então localizar possíveis "defeitos" no padrão hexagonal.
Para um padrão hexagonal perfeito, cada partícula apresenta 6 primeiros vizinhos, cada qual posicionado simetricamente em torno dessa. Analisando a simetria, cada vizinho consecutivo deve apresentar um ângulo de . Buscando uma relação em que esta situação seja a situação de , é possível definir que o vale:
Onde i é o índice do i-ésimo vizinho e n é o número de primeiros vizinhos. Esta relação deixa específico que o caso de 6 primeiros vizinhos e resulte em um valor de .
Implementação Computacional
Pode-se separar a implementação computacional em dois procedimentos necesssários: Encontrar os primeiros vizinhos para cada partícula e então calcular, de fato, o valor de .
Encontrando vizinhos
Por definição, os primeiros vizinhos de uma partícula são aquelas partículas que estão presentes em um anel mais próximo dela, como mostra a figura ao lado.
O problema de encontrar primeiros vizinhos é um problema bastante discutido em teoria da computação e diversos métodos foram desenvolvidos para efetuar esta tarefa. Desta forma a maioria dos métodos otimizados são de difícil implementação e então não serão tratados nesse verbete.
O método aqui citado é pouco otimizado, pois utiliza-se um for em , porém de implementação razoavelmente simples. O método consiste em encontrar as partículas mais próximas da partícula-teste (partícula em que busca-se calcular o valor de baseando-se em outro valor já calculado, a medida do g(r), desta forma o valor de resultará em um valor razoável ( >0.8 ) se os vizinhos dentro do raio medido no g(r) são de fato 6 e estão localizados em um padrão quase-hexagonal em torno da partícula-teste.
Para encontrar os primeiros vizinhos baseando-se no g(r), precisa-se observar qual a medida do primeiro pico no gráfico do g(r) e então definir este como uma distância de corte para a partícular ser ou não vizinha (isto é, um circulo com o raio da distância de corte ao redor da partícula-teste de forma que outras partículas dentro deste círculo são suas vizinhas). Neste caso, definem-se três vetores: "neighborsX" (que guarda a posição X do vizinho), "neighborsY"(que guarda a posição Y do vizinho) e "dNeighbors"(que guarda a distância do vizinho)[É recomendado definir estes vetores com tamanho pelo menos de 10, pois é possível que sejam encontrados mais de 6 vizinhos, para isso existe a variável de controle "k" que guarda quantos vizinhos cada partícula tem] e então faz-se o for em buscando-se as distância menores que o raio de corte:
for(i=0;i<NP;i++){
k=0;
xOrg=xx[i];
yOrg=yy[i];
for(j=0;j<NP;j++){
x2=xx[j];
y2=yy[j];
if(i!=j){
deltaX=fabs(x2-x1);
deltaY=fabs(y2-y1);
//Condições de contorno
deltaX=deltaX-rint(deltaX/Lx)*Lx;
deltaY=deltaY-rint(deltaY/Ly)*Ly;
// --- //
d=sqrt((deltaX*deltaX)+(deltaY*deltaY));
if(d<radiusLimit)
{
neighborsX[k]=x2;
neighborsY[k]=y2;
dNeighbors[k]=d;
k+=1;
}
}
}
Portanto após esse algoritmo, temos guardadas as posições das partículas mais próximas. É importante ressaltar que o "for" em partícula-teste ainda não foi fechado, e ainda dentro deste mesmo loop serão calculados os valores de .
Calculando o Psi 6
Tendo os valores de X e Y dos vizinhos da partícula-teste, podemos proceder para o cálculo do . Para este cálculo, precisamos dos ângulos que cada vizinho tem em relação a partícula-teste, isto é, setamos um referencial X-Y com a origem na partícula-teste e calculamos o ângulo que cada vizinho tem com o eixo X, este ângulo será chamado de e será dado pela seguinte relação:
Na implementação em código fica:
for(j=0;j<k;j++){
delX=neighborsX[j]-xx[i];
delY=neighborsY[j]-yy[i];
//Condições de contorno periódicas
delX=delX-rint(delX/Lx)*Lx;
delY=delY-rint(delY/Ly)*Ly;
// --- //
angle[j]=atan2(delY,delX);
if (angle[j]<0)
angle[j]=2*PI+angle[j];
psix[i]=0;
Agora somente precisa-se realizar um algoritmo que ordene o vetor "angle" do menor para o maior ângulo, utilizando algoritmos de ordenamento (bubblesort, quicksort, etc) desta forma é possível calcular os .
Feito o ordenamento do vetor, basta calcular a média do valor do de cada vizinho. Para isto, precisamos calcular os valores de em função dos , basta realizar a subtração do próximo vizinho no vetor pelo valor do ângulo do vizinho atual, dessa forma: com exceção do último vizinho, que será o ângulo dele menos o do primeiro, desta forma a implementação fica:
firstAngle=angle[0];
for(j=0;j<k-1;j++){
angle[j]=cos(6.*(angle[j+1]-angle[j]));
}
angle[k-1]=cos(6.*(firstAngle+2*3.1415-angle[k-1]));
for(j=0;j<k;j++){
psix[i]+=angle[j]/6.;
}
}
}
E então está calculado o valor de , para facilitar a implementação é recomendado que este algoritmo seja uma função dentro de seu código e desta forma retorne o vetor "psix".
Exemplos
Para o caso de um potencial de Lennard-Jones, é possível observar resultados de mapas de , isto é, um mapa que mostra para cada partícula o seu valor da medida, da seguinte forma para cada valor de rho:
Aplicações em outras áreas
No estudo de como animais processam a sua posição em seu cerébro, são observadas as chamadas "Células de grade", neurônios que disparam em certos locais específicos do espaço e tendem a formar padrões de grade (hexagonal). Em posse das posições de disparo desses neurônios é possível utilizar a medida do para determinar a "hexagonalidade" dos disparos, podendo-se realizar estudos da influência desse formato na interpretação geoespacial do animal.





![{\displaystyle g(r,\Delta r)={\frac {V_{t}}{N^{2}}}\sum _{i=1}^{N}\sum _{j\neq i}^{N}\left[{\frac {rect\left({\frac {r-|r_{i}-r_{j}|}{\Delta r}}\right)}{V\left(r+{\frac {\Delta r}{2}}\right)-V\left(r-{\frac {\Delta r}{2}}\right)}}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ffa4d0a60de5ae56e453b93a941c2973db15928a)