Lennard-Jones e propriedades termodinâmicas
Nesta página apresentamos uma simulação de Dinâmica Molecular utilizando o potencial de Lennard-Jones como potencial de interação entre as partículas.
Potencial de Lennard Jones
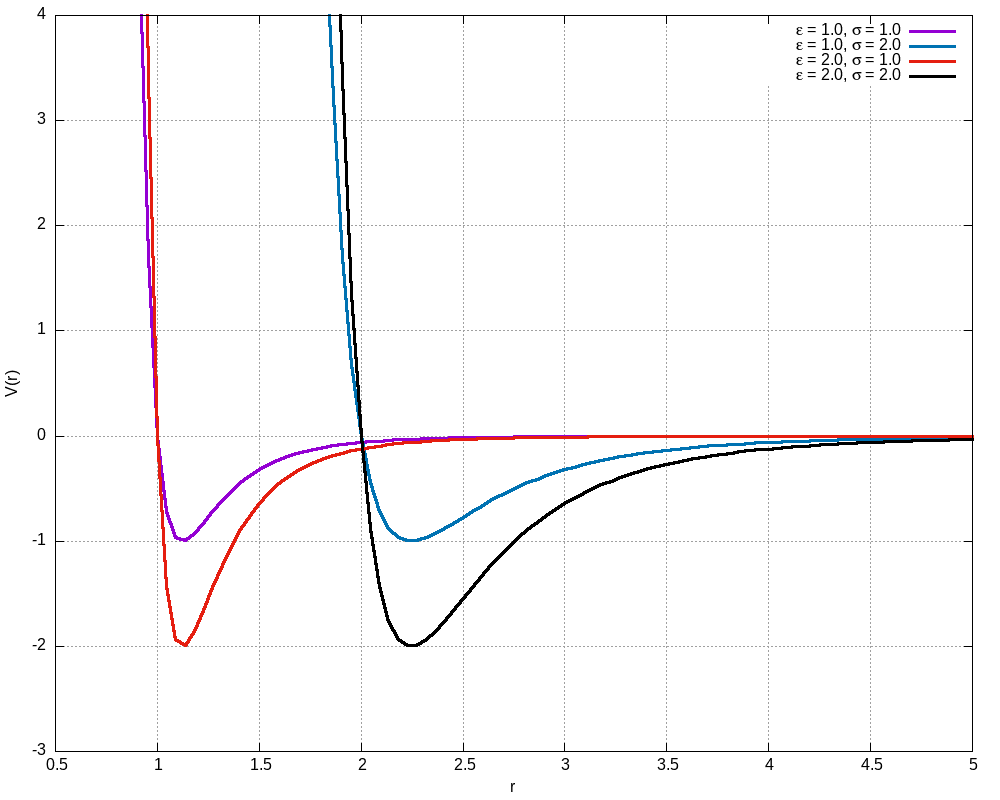
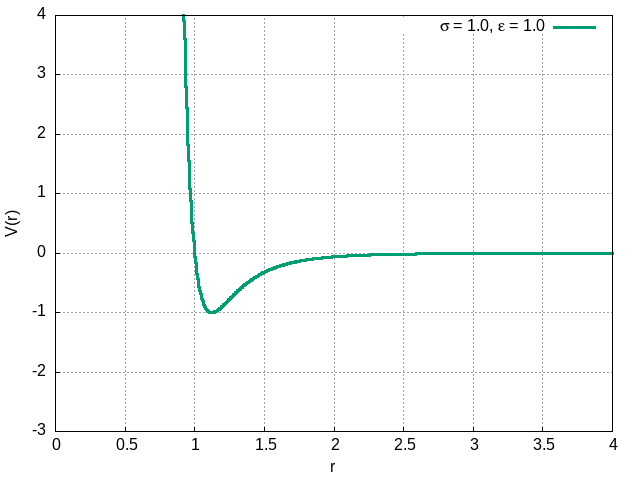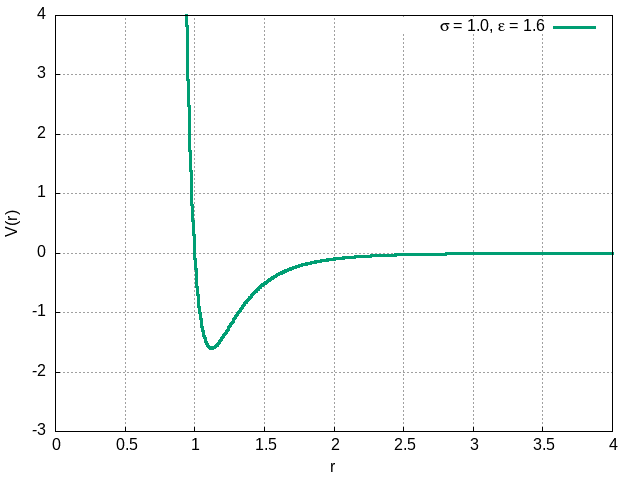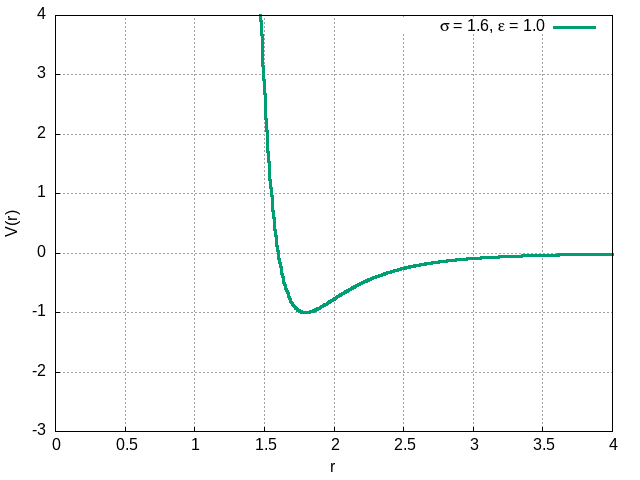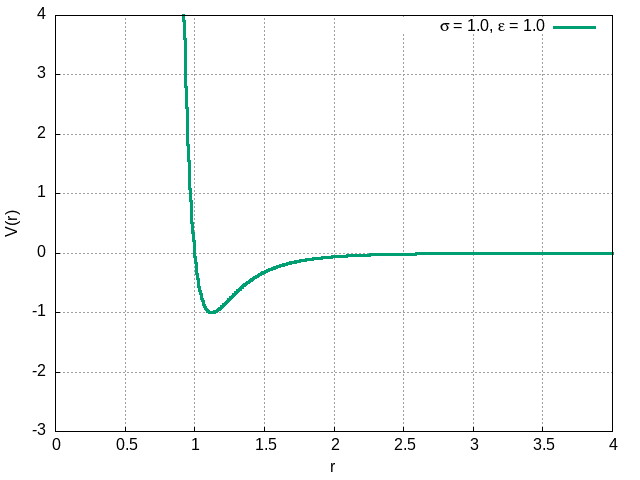
O potencial de Lennard Jones representa a interação entre os àtomos ou moléculas. Possui dois termos distintos: um repulsivo devido às forças de troca do princípio de exclusão de Pauli (origem quântica) e um atrativo devido às forças de van der Walls.
O termo proporcional à é o termo atrativo, enquanto o termo proporcional à é o termo repulsivo, muito mais intenso à curtas distâncias. A constante descreve a profundidade do poço deste potencial, enquanto descreve a posição do ponto de equilíbrio estável. Nas imagens abaixo, podemos ver o comportamento do potencial para diferentes valores de e .
A força que uma partícula sofre, quando sujeita a este potencial é dada por
Dinâmica Molecular
A abordagem da dinâmica molecular se baseia em integrar as equações de movimento newtonianas para todas as partículas do sistema. Abaixo, podemos sintetizar um resumo dos passos para realizar uma simulação de dinâmica molecular.
1. Inicializar a posição e velocidade das partículas do sistema.
2. Calcular as forças de interação entre todas as partículas do sistema.
3. Integrar as equações de movimento utilizando algum algoritmo integrador.
4. Medir grandezas físicas, como energia cinética, energia potencial, temperatura e pressão.
A seguir, vamos explicitar cada um dos passos.
Inicialização
O primeiro passo é inicializar a posição inicial das partículas. Podemos iniciar a simulação com as partículas distribuídas em qualquer forma que desejarmos, entretanto a forma mais comum de arranjar as partículas é em um formato cúbico ou em um formato triangular. O formato triangular é mais denso do que o formato cúbico. Além disso, a densidade calculada em uma simulação é a densidade numérica, ou seja, quanto de área, ou volume se for em três dimensões, da caixa está ocupada. Esta densidade é calculada como a soma da área ocupada por cada partícula dividida pela area total da caixa. É importante mencionar que as partículas são pontuais, entretanto como o Potencial de Lennard-Jones tende à infinito quanto a distância entre as partículas diminui, cada partícula é como se tivesse um raio efetivo, na qual outra partícula não consegue atravessar. É como se partículas fossem esferas que sofrem colisões umas com as outras. Além disso, podemos inicializar a velocidade das partículas de diferentes formas.
a) Velocidade nula:
Se dermos velocidade nula para todas as partículas, a simulação vai demorar mais, uma vez que elas começarão o movimento somente devido à distância de interação entre elas. Se elas estiverem muito afastadas, a força entre elas será muito pequena e demorará muito para que o sistema comece a evoluir e equilibrar, que é o nosso objetivo.
b) Velocidade aleatória:
A opção mais utilizada. Ao iniciarmos as velocidades de forma aleatória, as partículas não demoram tanto para interagir e conseguimos identificar a parte não-equilibrada e a equilibrada do sistema.
c) Distribuição de Maxwell-Boltzmann de velocidades:
Já esperamos que quando o sistema for equilibrar, a distribuição de velocidades das partículas seja a distribuição de Maxwell-Boltzman. Iniciar as velocidades desta forma acelera a simulação de forma significativa, entretanto se você tem algum interesse no sistema antes dele entrar em equilíbrio, você perderá informações sobre.
Cálculo das forças
Esta etapa é a mais custosa do ponto de vista computacional, uma vez que temos que calcular a força sofrida por cada partícula do sistema devido à todas as outras N-1 partículas, e com isso precisamos computar N(N-1) forças. Entretanto, se a interação exercida em i por j for a mesma que a exercida em j por i, então este número cai pela metade. Ainda é possível reduzir o tempo e o gasto computacional se considerarmos apenas as partículas dentro de um "raio crítico" . Este raio crítico é geralmente tomado como metade do tamanho da caixa. Em nossa simulação, contabilizamos todas as interações pois o número de partículas não é muito grande.
Integração das equações de movimento
Uma vez que nós obtemos a força sofrida por cada partícula, estamos preparados para integrar as equações de movimento. Para isso, precisamos escolher algum integrador que atenda à nossas necessidades. O integrador mais adequado aos nossos propósitos é um algoritmo do tipo Verlet. Em particular, utilizamos em nossa simulação o algoritmo de Velocity-Verlet.
As vantagens desse algoritmo são que ele preserva a área do espaço de fase, é reversível temporalmente, como são as euqações de Newton, conserva a energia para alcances curtos, é de fácil implementação e é rápido. A desvantagem é que ele não é um dos métodos mais precisos. Devemos nos lembrar que a precisão está diretamente relacionada ao tempo e gasto computacional, além é claro, da escolha adequada do algoritmo.
Medidas
Nesta etapa, calculamos as grandezas termodinâmicas de interesse. A primeira grandeza relevante é a energia mecânica do sistema, uma vez que gostaríamos de saber se ela está sendo conservada. Para isso, precisamos calcular a energia cinética e a energia potencial do sistema. Naturalmente, a energia mecânica deste sistema será
Outra grandeza importante é a temperatura. Podemos calcular a temperatura através da energia cinética utilizando o teorema da equipartição da energia da seguinte forma
onde estamos considerando nosso sistema com 2 dimensões. Como a energia cinética flutua durante a simulação, a temperatura também o fará.A temperatura será
onde é o número de graus de liberdade para um número fixo de partículas com momento total fixo.
Por último, gostaríamos de medir a pressão do nosso sistema, que será dada por
onde é a densidade numérica, é a constante de Boltzmann, é a temperatura, é a dimensionalidade do sistema (em nosso caso 2) e é a aŕea da caixa. Devemos nos lembrar que estamos considerando em nossa simulação como sendo 1, pois como ela é uma constante muito pequena, poderia acumular erros numéricos. Setamos como 1 e todas as unidades das grandezas ficam em termos de .
Por último, devemos nos lembrar que todas estas grandezas estão flutuando no tempo. Para obter, por exemplo, a temperatura média do sistema, deveríamos fazer uma média sobre todas essas flutuações.
Convenção da imagem mímina e PBC
Existem algumas características que devem ser levadas em consideração quando fazemos uma simulação de dinâmica molecular. Como integrar as equações de movimento requer um poder computacional elevado para muitas partículas, fazemos uma simulação de um "pedaço" do sistema de interesse. Construímos este "pedaço" como uma caixa de lado L e confinamos nossas partículas neste espaço. O número de partículas permanecesse constante e quando uma partícula atravessa a fronteira da caixa, ela retorna do outro lado. Isso simula um sistema infinito. Esta condição de contorno é chamada de condição de contorno periódica (Periodic Boundary Condition - PBC).
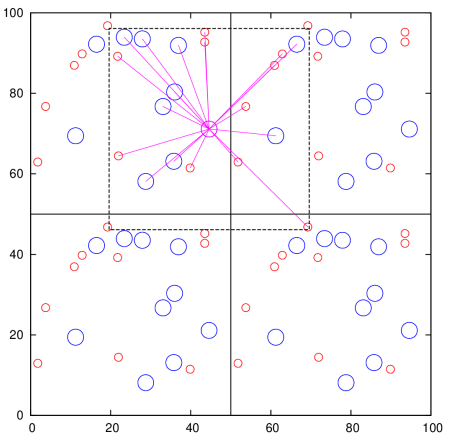
Outra característica importante é a convenção da imagem mínima. Esta característica diz respeito à interação entre as partículas. Como cada partícula interage com todas as outras, se a distância entre as partículas for maior do que a metade da caixa, ou seja, L/2, então devemos imaginar que ela vai interagir com a partícula correspondente na caixa "ao lado". Isso é uma extensão do conceito de sistema infinito discutido anteriormente. Ao lado temos uma figura que esquematiza este conceito.
Função Distribuição Radial
Outra grandeza importante é a função distribuição radial (FDR), que nos diz como é a organização local ao redor de um átomo. A FDR é um histograma e pode ser calculada como
onde A é a área da caixa e N é o número de partículas.
Simulação
Nesta simulação, utilizamos L = 25 e N = 108, onde N é o número de partículas. Neste contexto, obtemos .
Bibliografia
D. Frenkel, B. Smit. Understanding Molecular Simulation: From Algorithms to Applications. Academic Press. 2002.
![{\displaystyle V(r)=4\epsilon \left[\left({\frac {\sigma }{r}}\right)^{12}-\left({\frac {\sigma }{r}}\right)^{6}\right].}](https://wikimedia.org/api/rest_v1/media/math/render/svg/59bc3c3370e9df2d0df07c5bb4d66628b064d5b1)





![{\displaystyle {\boldsymbol {F}}({\boldsymbol {r}})=4\epsilon \left[12{\frac {\sigma ^{12}}{r^{13}}}-6{\frac {\sigma ^{6}}{r^{7}}}\right]{\hat {\boldsymbol {r}}}.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d6e0a4e3f4a59aa712b64752201ec970d71473d8)





![{\displaystyle E={\frac {1}{2}}m(v_{x}^{2}+v_{y}^{2})+\sum _{i<J}4\epsilon \left[\left({\frac {\sigma }{r_{ij}}}\right)^{12}-\left({\frac {\sigma }{r_{ij}}}\right)^{6}\right].}](https://wikimedia.org/api/rest_v1/media/math/render/svg/fce274b401044e108178c959799c08fcec6435f9)