Potencial de Lennard-Jones
Nome: André Bracht Burmeister
Fluidos estão em todos os lugares e, portanto, estudá-los é muito importante. Como são objetos de estudo extremamente complicados, se fazem necessários modelos e simplificações. Uma simplificação muito usada é a análise do sistema em duas dimensões apenas, excluindo a terceira dimensão espacial.
O Modelo
Um fluido bidimensional pode ser modelado como partículas em um plano com densidade (partículas por ). O plano é um quadrado de lado . Para escolher a posição inicial de uma partícula, são escolhidas coordenadas e aleatórias entre e .
O Método de Monte Carlo
Para evoluir o sistema, usaremos caminhadas aletórias, escolhendo as partículas com o método de Monte Carlo[[1]]. O método de Monte Carlo consiste em escolher uma partícula aleatoriamente entre as . Então escolhemos aleatoriamente um deslocamento proposto para a partícula , onde e estão entre e . A partícula só é realmente deslocada em de acordo com o Algoritmo de Metropolis-Hasting.
Algoritmo de Metropolis-Hasting
O Algoritmo de Metropolis-Hasting consiste em escolher se o movimento será ou não aceito usando a diferença de energia potencial entre o estado após o movimento e anterior ao movimento . A probabilidade de aceitar o movimento é dada por:
Onde , é a temperatura e é a constante de Boltzmann.
O Potencial de Lennard Jones
Para que o algoritmo funcione, precisamos então encontrar uma maneira de calcular a energia potencial de cada partícula. O potencial que uma partícula causa na outra, pode ser aproximado a uma distribuição em termos da distância entre elas:
Esse potencial é conhecido como o potencial de Lennard-Jones, primeiro apresentado por ele em seu artigo [1].
Unidades Reduzidas
Como usar unidades do sistema internacional seria muito custoso computacionalmente, precisaremos usar unidades reduzidas em termos de , e a constante de Boltzmann . As unidades utilizadas foram:
| Grandeza | Comprimento | Temperatura | Energia | Densidade |
|---|---|---|---|---|
| Unidade |
Usando as novas unidades o potencial de Lennard-Jones fica:
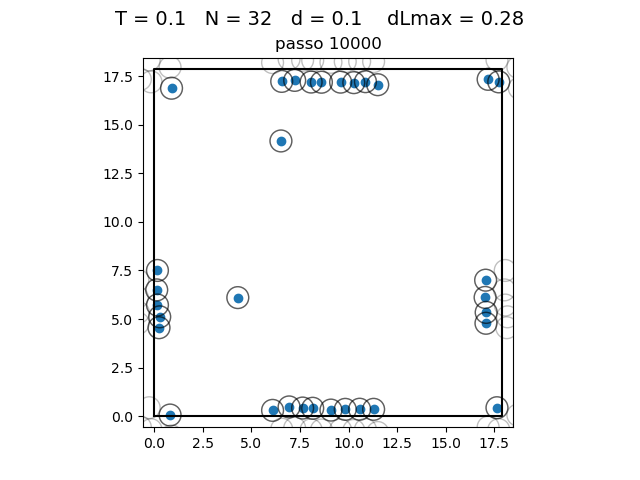
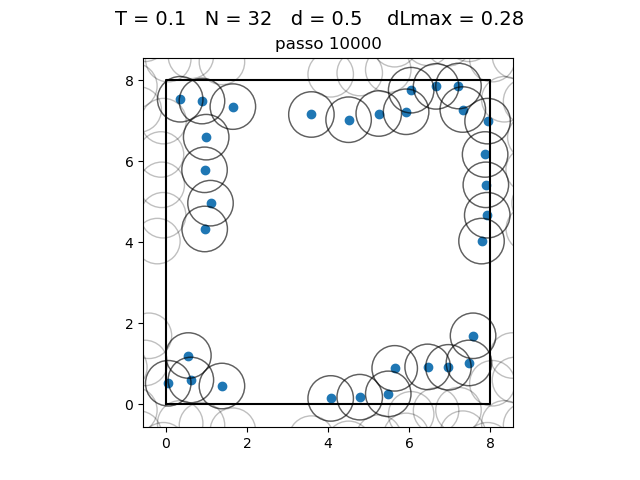
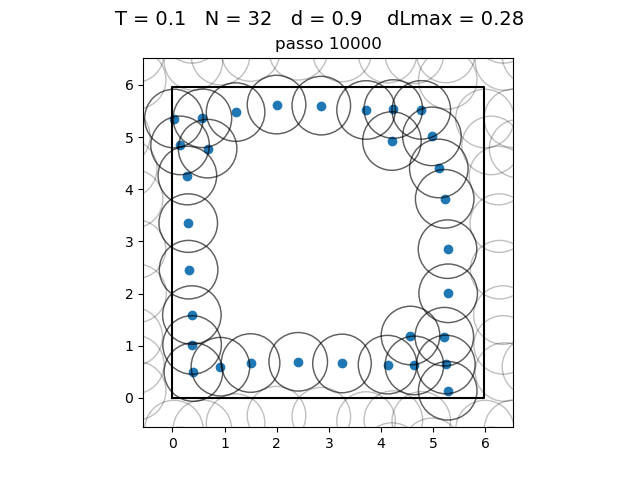
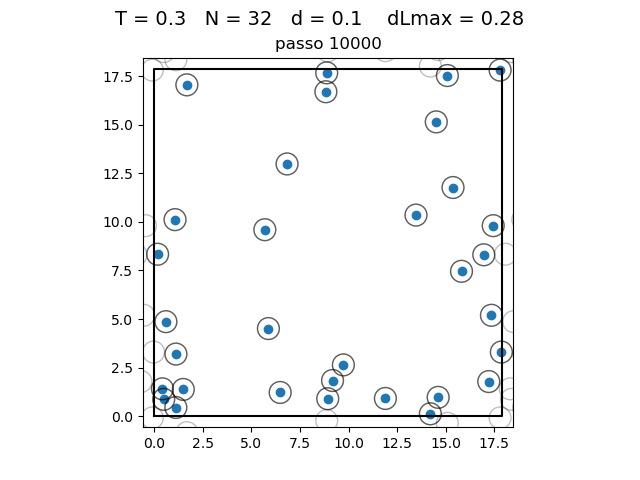
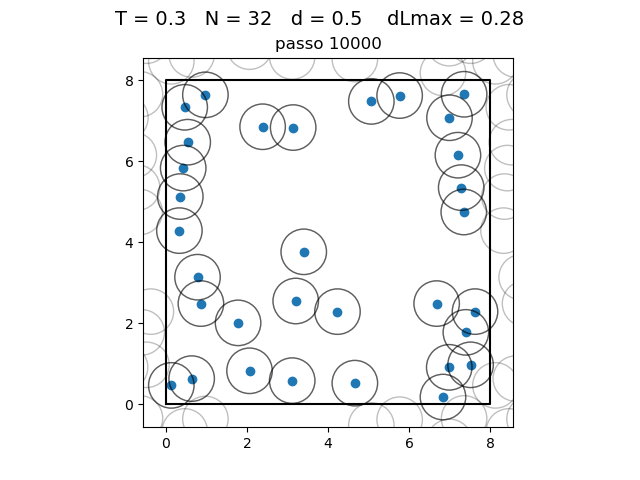
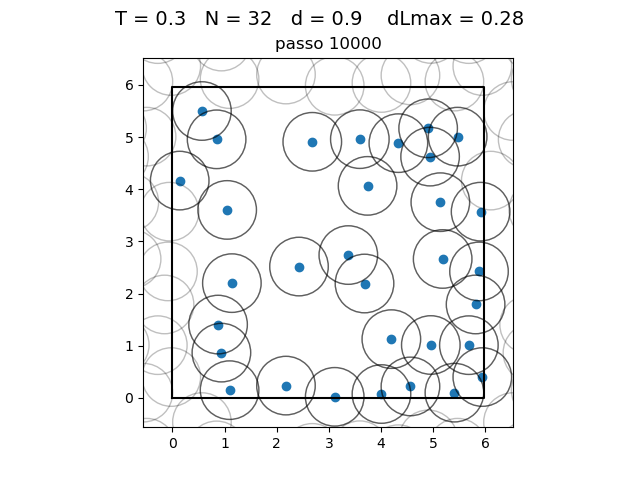
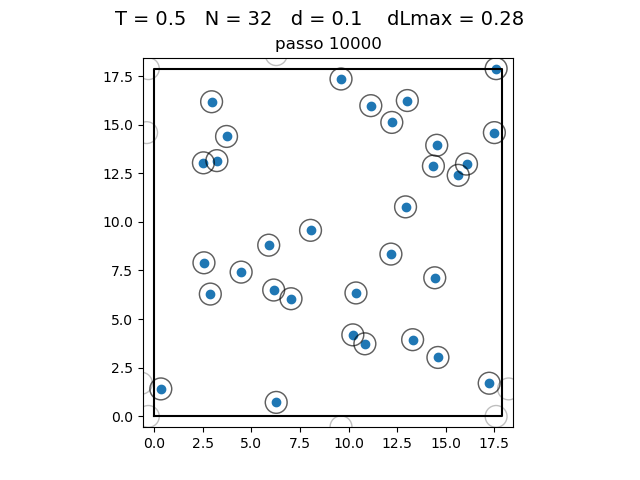
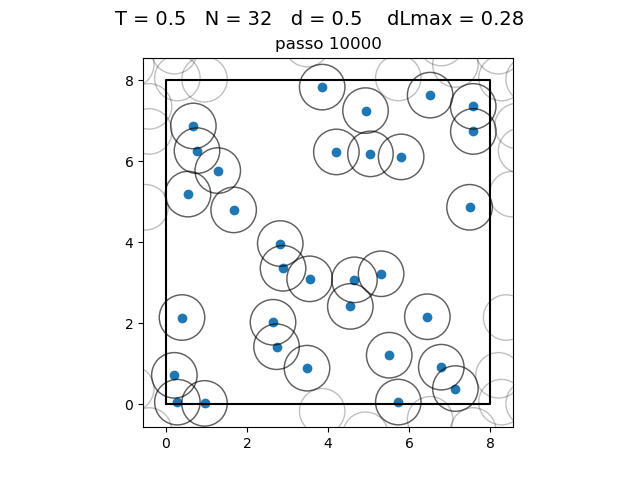
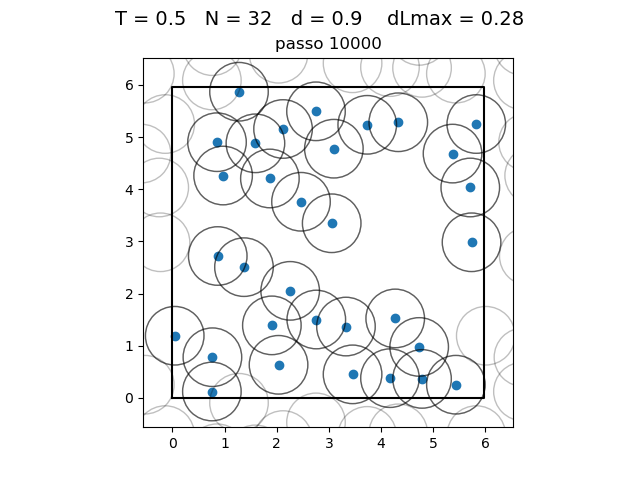
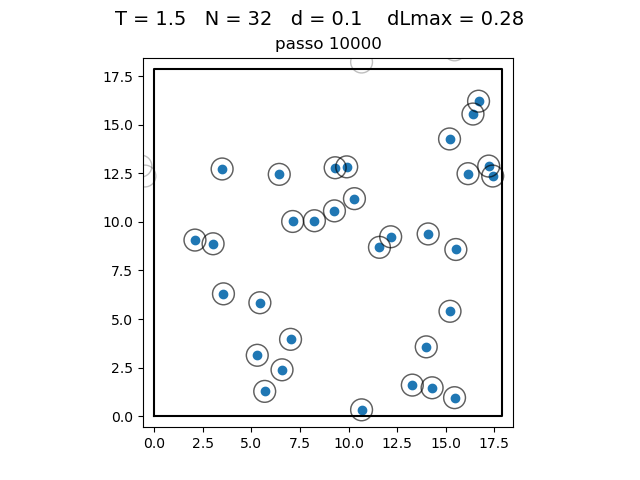
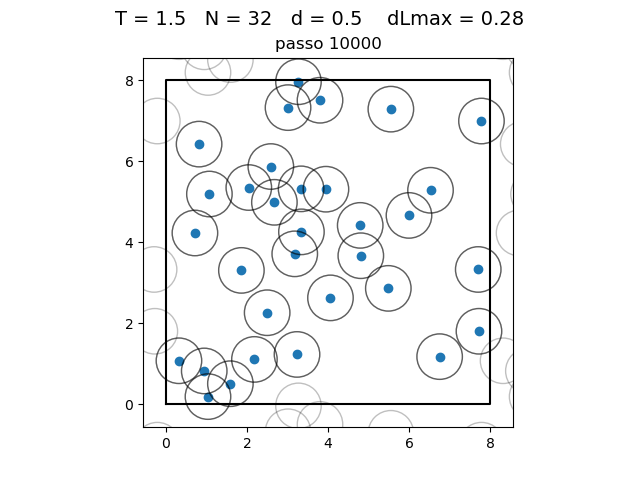
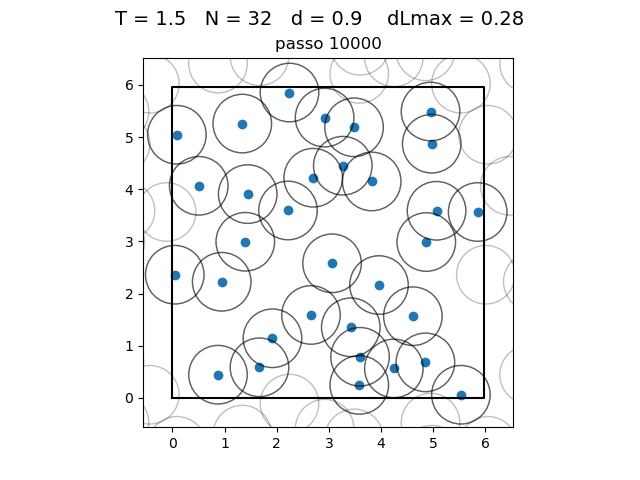
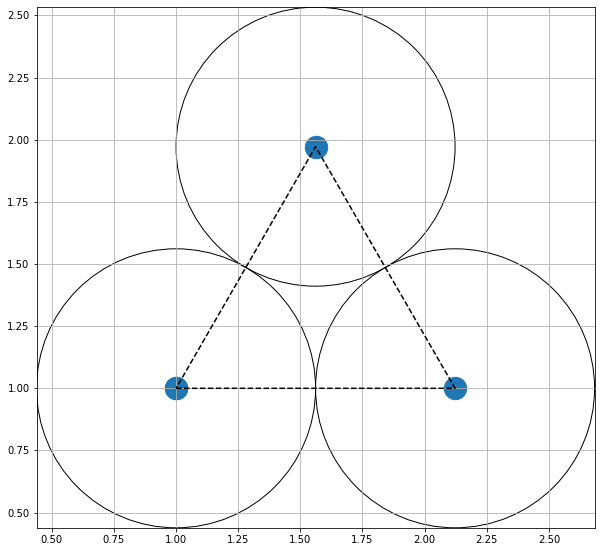
O formato do potencial faz com que para raios grandes, a interação entre partículas seja muito pequena e, para raios muito pequenos, haja uma força de repulsão que tende ao infinito. Além disso, a região de potencial mínimo se encontra a do centro da partícula. Ou seja, as partículas tendem a se organizar em uma configuração similar a da figura à direita, onde o raio dos círculos é
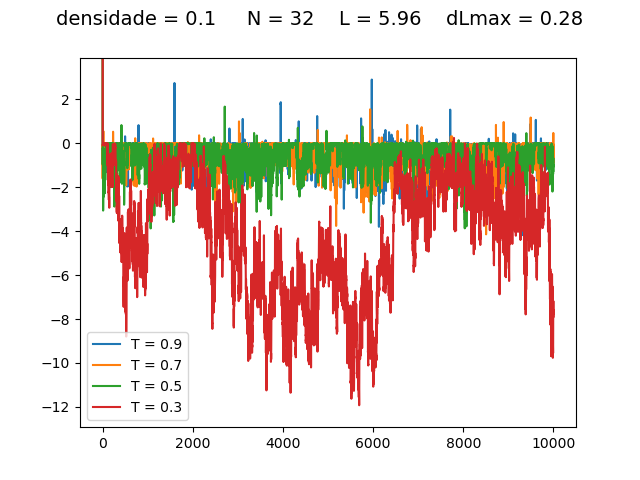
Escolha de
Foi escolhido um deslocamento máximo , pois isso faz com que o deslocamento não seja maior que metade da distância ao mínimo do potencial.
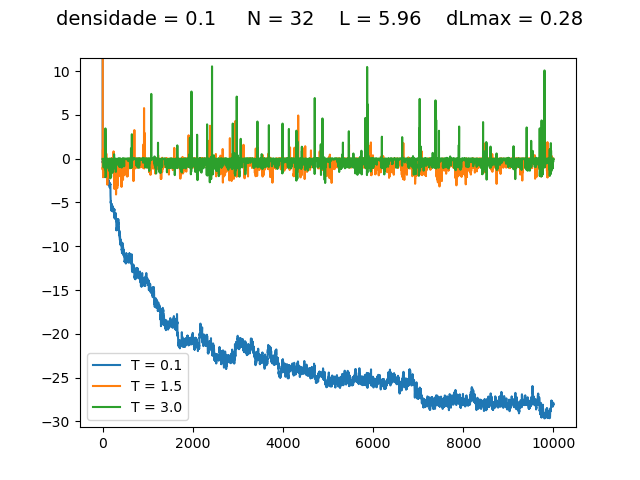
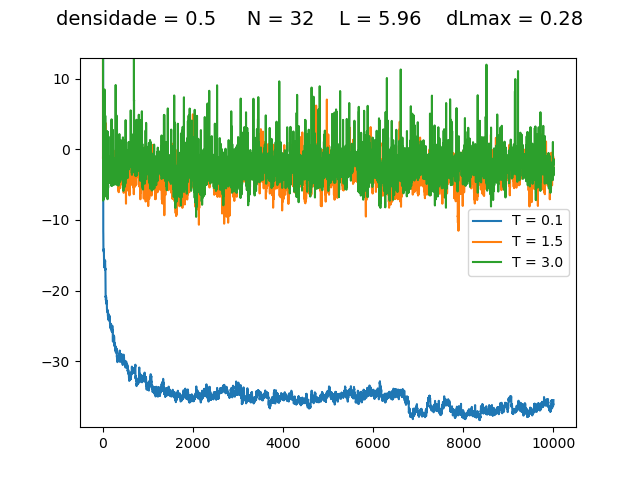
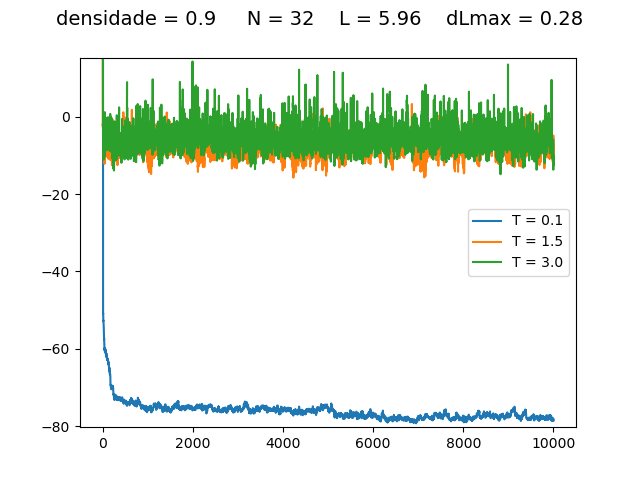
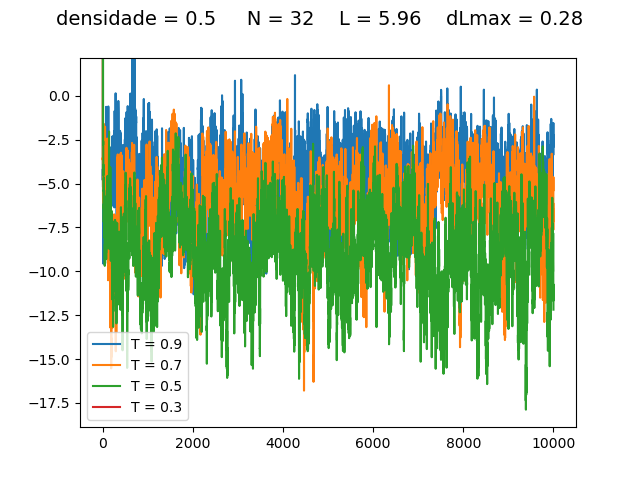
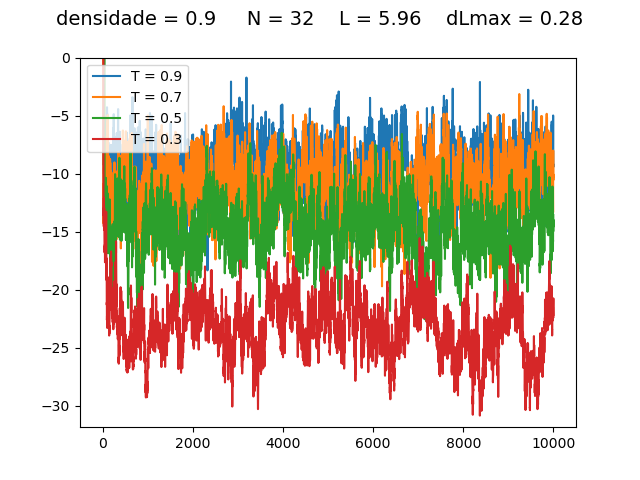
Resultados
Energia
| Medidas de energia a cada para diferentes densidades e temperaturas | ||
|---|---|---|
Transição de fase
| Posição das partículas ao fim da simulação com diferentes densidades e temperaturas | ||
|---|---|---|
Código
"""lennard_jones.ipynb
Automatically generated by Colaboratory.
Original file is located at
https://colab.research.google.com/drive/1jSPjBz0RXXWLEAzf3zcHgdlQpzjyO_ni
# Potencial de Lennard-Jones
# Monte Carlo no contínuo
#### Delete variables and import packages
"""
import numpy as np
from matplotlib import pyplot as plt
from matplotlib.animation import FuncAnimation
import random
from scipy.optimize import bisect as find_root
"""## Simulação:
### Definições Numéricas:
"""
densidade = 0.1 #(2 ** (2/3)) * (3 ** (-1/2)) # partícula(s) por sigma^2 (partículas por área)
densidade_round = round(densidade, 2)
n_particles = 64
L = np.sqrt(n_particles / densidade) # unidade de sigma
print(L)
dLmax = 0.1
MCS = 10000
eps = 1
T = .1
k = 1
beta = 1/(k*T)
raio = 2**(-5/6)
"""### Funcões:"""
def lennard_jones(r1, r2, L): # r1 e r2 em unidades de sigma (na vdd seria r1/sig e r2/sig)
dr = np.absolute(np.subtract(r2, r1))
for i in np.arange(2):
if dr[i] > 0.5:
dr[i] = L - dr[i]
r2 = (dr[0] ** 2) + (dr[1] ** 2)
sr2 = 1/r2
return 4 * (((sr2) ** 6) - ((sr2) ** 3))
def initial_energy(pos_particles, n_particles, L):
U0 = 0
for xxx in range(n_particles - 1):
for yyy in range(xxx+1, n_particles):
U0 += lennard_jones(pos_particles[:, xxx], pos_particles[:, yyy], L=L)
return U0
def energy_var(p, pos_particles, n_particles, L, new_pos=np.zeros(2)):
U1 = 0
U2 = 0
for i in range(n_particles - 1):
j = (i + p + 1) % n_particles # j vai ser todas as partículas
# exceto a partícula p (porque p interage com todas, menos com ela mesma)
U1 += lennard_jones(pos_particles[:, j], pos_particles[:, p], L=L)
U2 += lennard_jones(pos_particles[:, j], new_pos, L=L)
return U2 - U1
def main_loop(dLmax, beta, n_particles, MCS, L, acceptance_array=False,
last_acceptance=False):
pos_particles = L * np.random.random([2, n_particles])
# Random positions:
pos_MCS = np.zeros([MCS+1, 2, n_particles])
pos_particles = pos_particles
pos_MCS[0] = pos_particles
# Energy:
energy_arr = np.zeros(MCS+1)
energy = initial_energy(pos_particles, n_particles, L=L)
energy_arr[0] = energy
# Acceptance:
accepted = 0
if acceptance_array:
acceptance_arr = np.zeros(MCS)
# Main Loop:
for m in range(MCS):
for _ in range(n_particles):
p = random.randint(0, n_particles - 1)
# change position:
displacement = dLmax * ((2 * np.random.random([2])) - 1)
new_pos = pos_particles[:, p] + displacement
for i in range(2):
if new_pos[i] < 0:
new_pos[i] += L
elif new_pos[i] > L:
new_pos[i] -= L
dU = energy_var(p, pos_particles, n_particles, L, new_pos)
if dU <= 0:
pos_particles[0][p] = new_pos[0]
pos_particles[1][p] = new_pos[1]
energy += dU
accepted += 1
else:
if random.random() < np.exp(-beta * dU):
pos_particles[0][p] = new_pos[0]
pos_particles[1][p] = new_pos[1]
energy += dU
accepted += 1
if not (acceptance_array):
pos_MCS[m+1] = pos_particles
energy_arr[m+1] = energy
if acceptance_array:
acceptance_arr[m] = accepted / (n_particles*(m + 1))
if acceptance_array:
return acceptance_arr
elif last_acceptance:
return accepted / (n_particles*MCS)
else:
return pos_MCS, energy_arr, (accepted / (n_particles*(m + 1)))
def save_data(dLmax=dLmax, n_particles=n_particles, MCS=MCS, T=T, densidade=densidade):
beta = 1 / (k*T)
L = np.sqrt(n_particles / densidade) # unidade de sigma
print(L)
pos, energy, acceptance = main_loop(dLmax=dLmax, beta=beta, n_particles=n_particles,
MCS=MCS, L=L)
densidade_round = round(densidade, 3)
# save positions:
pos_reshaped = pos.reshape(pos.shape[0], -1)
np.savetxt(f"positions_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}_dL_{dLmax}.txt",
pos_reshaped)
# save energy:
np.savetxt(f"energy_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}_dL_{dLmax}.txt",
energy)
return pos, energy, acceptance
def open_pos(file, n_particles):
loaded_pos = np.loadtxt(file)
load_original_pos = loaded_pos.reshape(loaded_pos.shape[0], loaded_pos.shape[1] // n_particles, n_particles)
return load_original_pos
"""### Rodar e salvar dados:
#### Rodar uma vez:
"""
dLmax = round(0.5*raio, 3)
pos, energy, acceptance = save_data(dLmax=dLmax)
print(f'dLmax={dLmax} \n beta={beta} \n n_particles={n_particles} \n MCS={MCS} \n\n pos[0]: \n {pos[0]}')
print(acceptance)
"""#### Rodar para vários T e rô:"""
dLmax = round(0.5*raio, 3)
n_particles = 32
T_arr = np.array([0.1, 1.5, 3])
dens_arr = np.array([0.1, 0.5, 0.9])
for T in T_arr:
print()
print(f'T = {T}')
for densidade in dens_arr:
print(f'd = {densidade}')
pos, energy, acceptance = save_data(dLmax=dLmax, n_particles=n_particles, MCS=MCS, T=T, densidade=densidade)
#print(f'dLmax={dLmax} \n beta={beta} \n n_particles={n_particles} \n MCS={MCS} \n\n pos[0]: \n {pos[0]}')
print(f'acceptance = {acceptance}')
"""
## Testes:
### Encontrar deslocamento máximo:
Testando valores:
"""
def testing_dL(possible_dL, times):
for l in possible_dL:
accept = np.zeros(times)
for d in range(times):
accept[d] = main_loop(dLmax=l*L, last_acceptance=True, acceptance_array=False)
print(f'dLmax = {l}*L, acceptance = {np.mean(accept)}')
# Roda mais geral:
possible_dL = np.round(0.1*np.arange(1, 10), 1)
testing_dL(possible_dL, 5)
# Ajuste fino:
possible_dL = np.round(0.01*np.arange(10, 14), 3)
testing_dL(possible_dL, 50)
# Novo dLmax:
dLmax = 0.12*L
print(dLmax)
"""### Comportamento da acceptance:"""
def acceptance_behaviour(possible_dL, times):
for d in range(times):
fig, axes = plt.subplots(1, len(possible_dL), figsize=(20, 5))
fig.suptitle(f'T = {T} N = {n_particles} d = {densidade_round} dLmax = {round(dLmax, 2)}', fontsize=14)
for l in range(len(possible_dL)):
print(f'{l+1}/{len(possible_dL)}, {d+1}/{times}')
acceptance = main_loop(dLmax=possible_dL[l]*raio, acceptance_array=True)
axes[l].plot(np.arange(len(acceptance)), acceptance)
axes[l].set_ylim([0, 1])
axes[l].set_title(f'dLmax = {possible_dL[l]}*2^(-5/6)')
plt.show()
plt.close()
# Plotando acceptance
possible_dL = np.round(0.2*np.arange(1, 5), 1)
acceptance_behaviour(possible_dL, 1)
# Mais fino:
possible_dL = np.round(np.concatenate([0.0001*np.arange(1, 6)]), 4)
acceptance_behaviour(possible_dL, 1)
"""
## Figuras
### Intial and final positions:
"""
def scatter_pos(pos, indices, T, L, circles=True):
graphs = len(indices)
fig, axes = plt.subplots(1,graphs)
fig.suptitle(f'T = {T} N = {n_particles} d = {densidade_round} dLmax = {round(dLmax, 2)}', fontsize=14)
for j in range(graphs):
k = indices[j]
if circles:
# draw the circles where they are
for i in range(n_particles):
c = plt.Circle([pos[k][0][i], pos[k][1][i]], raio, fill=False, edgecolor='black', alpha = 0.5)
axes[j].add_patch(c)
# draw the circle's projections:
for a in range(-1, 2):
for b in range(-1, 2):
for i in range(n_particles):
c = plt.Circle([pos[k][0][i] + a*L, pos[k][1][i] + b*L], raio, fill=False, edgecolor='black', alpha=0.25)
axes[j].add_patch(c)
points = axes[j].scatter(pos[k][0], pos[k][1])
margem = raio
axes[j].plot([0, L, L, 0, 0], [0, 0, L, L, 0], color='black')
axes[j].axis([-margem, L + margem, -margem, L + margem], 'equal')
axes[j].grid(visible=False)
axes[j].set_aspect("equal")
axes[j].set_title(f'passo {k}')
return fig, axes, points
for T in T_arr:
print()
print(f'T = {T}')
for densidade in dens_arr:
print(f'd = {densidade}')
densidade_round = round(densidade, 3)
file_end = f"_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}_dL_{dLmax}"
file_name = f"positions{file_end}.txt"
pos = open_pos(file_name, n_particles=32)
L = np.sqrt(n_particles / densidade) # unidade de sigma
plot = scatter_pos(pos=pos, indices=[0, pos.shape[0] - 1], T=T, L=L, circles=True)
plt.show()
plt.savefig(f"first_last_positions{file_end}.png")
plt.close()
"""### Energy:"""
def y_limits(energy):
energy_hist_range = energy[int(round(0.1*len(energy), 0)):]
return [min(energy_hist_range)-1 , max([0, max(energy_hist_range) + 1])]
"""#### Energy graph:"""
for T in T_arr:
print()
print(f'T = {T}')
for densidade in dens_arr:
print(f'd = {densidade}')
file_end = f"_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}_dL_{dLmax}"
energy = np.loadtxt(f"energy{file_end}.txt")
# Plot:
fig, ax = plt.subplots(1, 1)
titulo = f'T = {T} N = {n_particles} d = {densidade_round} L = {round(L, 2)} \n dLmax = {round(dLmax, 2)} acceptance = {round(acceptance, 2)}'
fig.suptitle(titulo,
fontsize=14)
energy_range = energy
ax.plot(np.arange(len(energy_range)), energy_range)
limites = y_limits(energy)
print(limites)
ax.set_ylim(limites)
plt.show()
plt.savefig(f"energy_graph{file_end}.png")
plt.close()
# Histogram:
fig, ax = plt.subplots(1, 1)
fig.suptitle(titulo,
fontsize=14)
ax.hist(energy[100: ], density=True)
ax.set_xlabel('energy')
plt.show()
plt.savefig(f"energy_histogram{file_end}.png")
plt.close()
"""#### Energy histogram:"""
"""## Animação"""
file_name = f"positions_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}_dL_{dLmax}.txt"
print(file_name)
start = 0
end = len(pos)
pos = open_pos(file_name)[start:end]
fig, ax = plt.subplots(1,1, figsize=(5, 6))
fig.suptitle(f'T = {T} N = {n_particles} d = {densidade_round} L = {round(L, 2)} \n dLmax = {round(dLmax, 2)} acceptance = {acceptance}',
fontsize=14)
margem = raio
ax.axis([-margem, L + margem, -margem, L + margem], 'equal')
ax.grid()
ax.set_aspect("equal")
ax.set_title(f'passo {k}')
c = []
for i in range(n_particles):
c.append(plt.Circle([pos[0][0][i], pos[0][1][i]], raio, fill=False, color='lightgrey'))
ax.add_patch(c[i])
scatter_points = ax.scatter(pos[0][0], pos[0][1], s=1000)
def animate(i):
for k in range(n_particles):
c[k].center = (pos[i][0][k], pos[i][1][k])
scatter_points.set_offsets(pos[i].T)
return scatter_points
anim = FuncAnimation(fig, animate, interval = 10, frames = end-start-1)
anim_file = f'animacao_T{T}_N{n_particles}_d{densidade_round}_MCS{MCS}_dL_{dLmax}.mp4'
print(anim_file)
anim.save(anim_file, writer = 'ffmpeg', fps = 20)
"""## Imagens:
### Raio:
"""
x0 = 1
y0 = 1
l = 2 * (2 ** 1/6)
triX = [x0, x0+l, x0+(l/2)]
h = np.sqrt(3)*l/2
triY = [y0, y0, y0 + h]
triX.append(triX[0])
triY.append(triY[0])
fig, ax = plt.subplots()
for i in range(3):
c = plt.Circle([triX[i], triY[i]], l, alpha=0.5)
c = plt.Circle([triX[i], triY[i]], l, alpha=0.5)
c = plt.Circle([triX[i], triY[i]], l, alpha=0.5)
ax.add_patch(c)
ax.plot(triX, triY, color = 'b')
ax.axis([x0-(l/2), x0 + (3*l/2), y0 - (l/2), y0 + h + l/2], 'equal')
ax.grid()
ax.set_aspect("equal")
plt.show()
plt.close()
"""### Potencial Lennard Jones:"""
def lennard_jones1d(r):
sr6 = 1/(r ** 6)
return 4 * (((sr6) ** 2) - (sr6))
xx = np.linspace(0, 4, 100)
yy = lennard_jones1d(xx)
fig, ax = plt.subplots(1, 1, figsize=(10, 4))
ax.plot([-10, 10], [0, 0], color='black')
ax.plot(xx, yy)
ax.set_ylabel('U(r)')
ax.set_xlabel('r')
ax.set_ylim([-1.5, 5])
ax.set_xlim([0, 3])
plt.show()
fig.savefig('lennard_jones_potential.png')
plt.close()
- ↑ J E Lennard-Jones 1931 Proc. Phys. Soc. 43 461





















![{\displaystyle U'(r')=4\epsilon \left[\left({\frac {\sigma }{r'}}\right)^{12}-\left({\frac {\sigma }{r'}}\right)^{6}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/7ef84cdd8901e3feec058279a5ff0f497a5fbf51)





![{\displaystyle U(r)=4\left[\left({\frac {1}{r}}\right)^{12}-\left({\frac {1}{r}}\right)^{6}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b6cc1c7619b98414369331ae46a5d8eab347f25c)