Modelos Epidemiológicos
Em construção
Grupo: Gabriel Schmökel, Julia Remus e Luis Gustavo Lang Gaiato
O objetivo do trabalho é realizar a implementação do modelo SIR e de um modelo simplificado do proposto pelo artigo "An epidemiological model with voluntary quarantine strategies governed by evolutionary game dynamics" [1] utilizando Monte Carlo.
Será apresentada uma breve introdução sobre o tema e as equações que envolvem o desenvolvimento dos cálculos, as implementações e seus respectivos resultados.
Introdução
O objetivo do trabalho é realizar a implementação do modelo SIR e de um modelo simplificado do proposto pelo artigo "An epidemiological model with voluntary quarantine strategies governed by evolutionary game dynamics" [1] utilizando Monte Carlo.
O modelo apresentado por Marco Amaral, et al [1] propõe que o indivíduo escolha fazer a quarentena dependendo do risco que observa a partir da quantidade de infectados no sistema. Acoplada à escolha pela quarentena está o modelo SIR com parâmetros distintos para indivíduos não isolados e isolados. Para realizar a simulação os autores utilizam a teoria de campo médio, obtendo ondas de infecção ao longo do tempo.
Neste trabalho, a escolha do indivíduo pela quarentena se dará de acordo com o estado do seus vizinhos mais próximos - utilizado o dilema do prisioneiro -, enquanto a evolução dos infectados no sistema será realizada com o modelo SIR tal como na referência citada. Para a simulação foi utilizado o método de Monte Carlo.
O esquemático do modelo SIR com a escolha da quarentena pode ser visto na seção Modelo SIR com quarentena voluntária.
Modelos
Modelo SIR
Em 1927 Kermack e McKendrick elaboraram o modelo SIR que tinha como função descrever o comportamento de uma pandemia [2]. O modelo SIR é um dos mais simples modelos comportamentais, o qual descreve a variação de três parâmetros ao longo do tempo:
- Susceptível (S): Número de indivíduos suscetíveis. Quando um indivíduo suscetível e um infectado entram em contato, o indivíduo suscetível tem uma probabilidade de contrair a doença, caso contraia o indivíduo deixa de ser suscetível e torna-se infectado.
- Infectado (I): Número de indivíduos infectados. Indivíduos infectados tem uma probabilidade de infectar indivíduos suscetíveis quando em contato, e uma probabilidade de tornar-se indivíduos removidos a medida que tempo avança.
- Recuperado ou Removido (R): Número de indivíduos removidos (recuperados ou mortos pela doença). Indivíduos que foram infectados, tornam-se recuperados ou mortos pela doença, desta forma entram na classificação de indivíduos removidos, já que não são mais suscetíveis a pegar a doença, pois adquiriram imunidade. Assumindo que o número de mortos é irrelevante ao se comparar com a população total, então o parâmetro R recebe o nome de indivíduos recuperados ou resistentes.
A dinâmica de uma epidemia geralmente é mais rápida do que os números de nascimentos e óbitos, por essa razão se omite estás quantidades no modelo comportamental simples do SIR. Usando uma população fixa, Kermack e McKendrick chegaram nas seguintes equações diferencias que descrevem a dinâmica de uma epidemia [2]:
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle \frac{dS}{dt} = - \beta IS \qquad (1) }
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle \frac{dR}{dt} = \gamma I \qquad (3) }
O sistema descrito pelas equações acima é não linear, porém é possível derivar uma solução analítica de forma implícita [3].
A população total no SIR é constante, e pode ser expressa pela soma total dos indivíduos suscetíveis, infectados e removidos. Abaixo segue a expressão:
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle S + I + R = N \qquad (4) }
Na forma normalizada a expressão (4) é representada por:
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle s + i + r = 1 \qquad (5) }
A taxa de transmissão da doença é dada pela variável beta, está depende de outras duas variáveis. A primeira indica a "transmissibilidade", definida por Tau, a segunda indica o "número médio de contatos" entre infectados e suscetíveis, indicada por "c barra". Portanto, pode-se escrever a taxa de transmissão pela expressão:
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle \beta = \tau \bar c \qquad (6) }
A taxa de remoção da doença, indicada por γ, é o inverso do período infeccioso, indicada por d, que o indivíduo permanecerá doente. Note, que quanto maior o período infeccioso, mais demorada é a remoção de Infectados para Removidos.
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle d = \frac {1}{\gamma} \qquad (7) }
Dinâmica do Modelo SIR e o Número de Reprodução
O número básico de reprodução, Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle R_0 } , corresponde ao número médio de pessoas que serão contagiadas pela doença quando um indivíduo infectado é introduzido em uma população completamente suscetível [4].
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle R_0 = \frac{\beta}{\gamma} S_0 \qquad (8) }
Conforme o valor de Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle R_0 } temos o seguinte comportamento [4]:
- Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle R_0 > 1 \Rightarrow } epidemia crescente;
- Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle R_0 = 1 \Rightarrow } equilíbrio endêmico;
- Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle R_0 < 1 \Rightarrow } epidemia decrescente.
Podemos interpretar melhor o comportamento do número de reprodução analisando o comportamento da curva I(S).
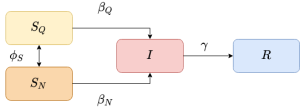
Modelo SIR com quarentena voluntária
No esquemático pode-se ver o modelo utilizado no trabalho, ele é uma simplificação do artigo escrito por Amaral, et al [1]. Nele temos:
- Componentes suscetíveis podem estar ou não em quarentena. A probabilidade Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle \phi_S} para a escolha da quarentena ocorrer depende de quantos infectados há nos vizinhos.
- Ao escolher estar ou não em quarentena, esses componentes tem probabilidades diferentes de adquirir a doença (Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle \beta_Q} e Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle \beta_N} ) e se tornar infectados.
- Infectados possuem a mesma probabilidade de se tornarem recuperados.
- Após recuperados da doença, as pessoas não conseguem adquiri-lá novamente.
- Todos os indivíduos escolhem estar ou não isolados, modificando a escolha do vizinho, mas somente a quarentena dos suscetíveis afeta a evolução SIR.
Para simular a quarentena voluntária é utilizado o jogo definido pelo Dilema do Prisioneiro. Esse dilema descreve a situação onde dois condenados (A e B) precisam decidir se colaboram ou não sem saber a decisão do seu par. Para isso temos 4 possibilidades: A e B colaboram (ambos saem com uma recompensa R), A colabora mas B não colabora (B ficaria com o valor da tentação T e A com o custo do sonso S), o caso contrário onde A não colabora e B colabora e A e B não colaboram (ambos ganham com uma penalidade P). [5]
Essas possibilidades podem ser resumidas em uma matriz de perdas e ganhos (matriz de payoff):
| A coopera | A não coopera | |
|---|---|---|
| B coopera | R / R | S / T |
| B não coopera | T / S | P / P |
Os valores da matriz payoff devem obedecer a ordem T > R > P > S. Além disso, para simulações com várias iterações deve ser obedecido que 2R > T + S. [5]
A proposta de observar a evolução da infecção dependendo da quarentena precisa utilizar mais de um par de interagentes, por isso é definido uma rede onde cada componente possui um ponto fixo. A partir disso, cada um desses componentes interagem com seus quatro vizinhos mais próximos (para os pontos do contorno é utilizado que as bordas são unidas por condições de contorno periódicas). [5]
Segundo Hauert e Szabó [5], os colaboradores tendem a ser extintos em jogos que consideram a interação aleatória, independente da sua concentração inicial, ou seja, todos tendem a não ganhar nada (não colaborar mutuamente) a fim de reduzir custos. Enquanto isso, se for proposto que um componente só escolhe uma estratégia conforme seus vizinhos, é visto é a formação de clusters de cooperadores e de não cooperadores; com isso, os componentes que estão na borda desses espaços, ganham na interação com os vizinhos colaboradores e perdem com a outra interação.
Esse jogo de quarentena é acoplado ao sistema SIR anteriormente descrito. A única diferença para o SIR sem o jogo é que as probabilidades de um componente S se tornar infectado mudam a partir do seu estado de quarentena.
Implementação
Os códigos da simulação foram implementados na linguagem C, para a visualização foi utilizada a linguagem Python.
Implementação modelo SIR
Talvezzzz: Usar essa Analytical solution of SIR-model [6] pra verificar se o código funciona
Implementação modelo SIR com quarentena voluntária
Algumas modificações são realizadas no código anterior: a probabilidade do indivíduo passar de suscetível a infectado depende da escolha pela quarentena e é adicionada a parte da escolha individual.
Inicialmente é definida a matriz de payoff, os parâmetros diferentes para indivíduos suscetíveis e os vetores que conterão os estados de quarentena.
A função do jogo (escolha pelo isolamento) é definido abaixo.
void jogo(int t){
/*
Dinâmica da escolha voluntária do quarentenado ou não
*/
int sitio, i, j, k;
double prob;
double sums, sumv;
int vizinho, dir;
for (i=0;i<L2;i++){
sitio = FRANDOM * L2;
dir = FRANDOM * 4 + 1;
vizinho = viz[sitio][dir];
sums = 0;
sumv = 0;
for(j=1 ; j < 5 ; j++) {
sums += payoff[dmc_quarentena[sitio]][dmc_quarentena[viz[sitio][j]]];
sumv += payoff[dmc_quarentena[vizinho]][dmc_quarentena[viz[vizinho][j]]];
}
prob = 1.0 / (1.0 + exp(-(sumv - sums) / TEMP ));
if(FRANDOM > prob) {
dmc_quarentena[sitio] = dmc_quarentena[vizinho];
}
}
// Escreve o estado de quarentena no arquivo, cada tempo uma linha
FILE *dmcfile;
char output2[10000];
sprintf(output2, "output_dinamica_loc.txt");
dmcfile = fopen(output2,"a");
for(k=0; k<L2; k++){
fprintf(dmcfile," %d ", dmc_quarentena[k]);
}
fprintf(dmcfile," \n ");
fclose(dmcfile);
return;
}
Visualização
#!/usr/bin/env python3
# -*- coding: utf-8 -*-
"""
Graficos - Trabalho SIR e SIR modificado
"""
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
from matplotlib import animation
#%% Read data
data = pd.read_csv('./output.txt',
sep=" ", index_col=False) # sir x tempo
data.sort_values(by='t', ascending=True, inplace=True)
data.set_index('t', inplace=True)
data_j = pd.read_csv('./output_jogo.txt',
sep=" ", index_col=False) # sir x tempo
data_j.sort_values(by='t', ascending=True, inplace=True)
data_j.set_index('t', inplace=True)
data['total'] = data['r'] + data['i'] + data['s']
L2 = data['s'][0]
#%% SIR versus time
plt.title('Evolução temporal da doença')
data = data / L2 # normalização da população
# data_j = data_j / data_j['s'][0] # normalização da população
plt.plot(data['r'], label='R')
plt.plot(data['s'], label='S')
plt.plot(data['i'], label='I')
# data_j = data_j / data_j['s'][0] # normalização da população
# plt.plot(data_j['r'], label='R jogo')
# plt.plot(data_j['s'], label='S jogo')
# plt.plot(data_j['i'], label='I jogo')
plt.ylabel('Fração da população')
plt.xlabel('Tempo')
plt.grid()
plt.legend()
#%% Dinamica da quarentena voluntaria
dmc_loc = np.loadtxt('./dados/output_dinamica_loc.txt') # lê o arquivo
#%%
L = int(np.sqrt(np.shape(dmc_loc)[1]))
# forma uma malha LxL, pra poder plotar como se fosse x - y - z
x = y = np.arange(0, L, 1)
xv, yv = np.meshgrid(x, y)
def animate(i):
plt.clf() # limpa a figura, pra nao ficar sobrepondo figs
# # titulos
plt.suptitle('Evolução da dinâmica de quarentenados', fontsize=14)
plt.title(f'Tempo: {i}', fontsize=10)
quarentena = np.zeros(shape=(L, L))
for j in range(L):
quarentena[j] = dmc_loc[i, j*L : (j+1) * L]
# # plot
graph = plt.scatter(xv,yv, c= quarentena, marker='.', vmax=1.0, vmin=0.0)
return graph
fig, ax = plt.subplots()
ani = animation.FuncAnimation(fig, animate, frames = len(dmc_loc), repeat=False, interval=5)
#%% Reorganiza para plot da quantidade por tempo
quarentena, nao_quarentena = [], []
for i in range(len(dmc_loc)):
quarentena.append(len(np.where(dmc_loc[i] == 0)[0])/L2)
nao_quarentena.append(len(np.where(dmc_loc[i] == 1)[0])/L2)
#%% Evolução da quantidade de quarentenados e infectados
fig, ax = plt.subplots()
ax2 = ax.twinx()
plt.title('Evolução da quantidade de quarentenados e infectados')
ax.plot(quarentena, label='QT', color='black', alpha=0.8)
# ax.plot(nao_quarentena, label='QN', color='red', alpha=0.6)
# ax.set_ylim(0, 1600*1.5)
ax2.plot(data['i'], label='I')
# ax.plot(data['r'], label='I')
# ax.plot(data['s'], label='I')
ax.legend(loc='lower left')
# ax2.legend(loc='lower right')
Resultados e Discussão
- ?
- evolução temporal monte carlo SIR com jogo
- evolução temporal monte carlo SIR com jogo e quantidade de quarentenados por passo
- evolução temporal monte carlo SIR com jogo e SIR normal
- animações com quarentenados e sir (acho que o pessoal gosta)
Os resultados obtidos mostram que uma quarentena voluntária diminuem significativamente o número de infectados em um primeiro momento, o que era esperado e comprovado por dados da Covid em países que conseguiram obter níveis de isolamento adequados [referencia pra essa frase].
Ao realizar as simulações, foi observado que tanto os parâmetros da matriz de payoff e os da dinâmica SIR quanto o sistema inicial da quarentena interferem na evolução da doença. Mesmo assim, não foi possível alcançar o conjunto de parâmetros ideais para obter as ondas de infecção ao longo do tempo.
Abaixo são comparadas as curvas do modelo SIR com quarentena voluntária variando o estado inicial do sistema em relação ao isolamento dos indivíduos.
- % de quarentenas random - todos eles de um lado - todos eles no centro
Referências
- ↑ 1,0 1,1 1,2 1,3 AMARAL, Marco; OLIVEIRA, Marcelo de; JAVARONE, Marco, An epidemiological model with voluntary quarantine strategies governed by evolutionary game dynamics. arXiv:2008.05979v2 [physics.soc-ph] .
- ↑ 2,0 2,1 MCKENDRICK, A.G.; KERMACK, W. O.. Mathematical Theory of Epidemics. Disponível em: https://gallica.bnf.fr/ark:/12148/bpt6k56208r/f728.item.langFR
- ↑ Harko, Tiberiu; Lobo, Francisco S. N.; Mak, M. K. (2014). "Exact analytical solutions of the Susceptible-Infected-Recovered (SIR) epidemic model and of the SIR model with equal death and birth rates". Applied Mathematics and Computation. 236: 184–194. arXiv:1403.2160. Bibcode:2014arXiv1403.2160H. doi:10.1016/j.amc.2014.03.030. S2CID 14509477
- ↑ 4,0 4,1 M.G.Roberts. The pluses and minuses of r 0 . Journal of the Royal Society interface4, 2007.
- ↑ 5,0 5,1 5,2 5,3 HAUERT, Christoph; SZABÓ, György. Game theory and physics. DOI: 10.1119/1.18485144 .
- ↑ Analytical solution of SIR-model