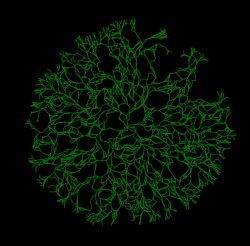
Simulação de Micélio de Fungo
Grupo: Arthur Dornelles, Bruno Zanette, Gabriel De David e Guilherme Hoss
O objetivo deste trabalho é modelar computacionalmente o desenvolvimento de micélios em fungos com base em mecanismos gerais pré-estabelecidos. O progresso e a complexidade dos modelos cresceram de maneira gradual ao longo do trabalho através de três modelos de crescimento diferentes. O trabalho foi inspirado - principalmente - nos dois primeiros capítulos do artigo de Steven Hopkins [1].
Motivação e Introdução aos Fungos
Fungos estão integrados em grande parte dos ecossistemas do planeta e cumprem importantes funções na manutenção e sobrevivência dos mesmos. De maneira geral, eles produzem enzimas que são responsáveis pela decomposição de matéria orgânica e - portanto - a reciclagem de diversos nutrientes do ambiente ao seu redor [2]. Em muitos casos, fungos formam fusões simbióticas com plantas ou algas e interagem de diferentes maneiras com diferentes organismos vivos. Neste trabalho, todavia, não faremos a análise dessas interações e focaremos no comportamento individual de crescimento de fungos.
Anatomicamente, fungos são compostos por células que se assemelham a tubos microscópicos, denominadas de hifas. Essas hifas então se ramificam e se fundem umas com as outras em um processo chamado de anastomose, formando uma complexa rede chamada de micélio (figura 1).
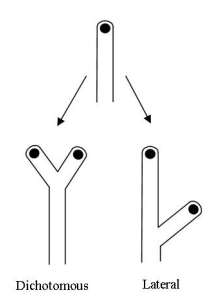
A criação de novas hifas, em geral, ocorre ao longo do tempo através de dois processos principais: o primeiro, denominado de ramificação dicotômica, consiste na ponta de uma hifa já existente se dividindo ao meio. O segundo processo é chamado de ramificação lateral, no qual, como o nome sugere, formam-se novos ramos e hifas na lateral de uma hifa já existente, como pode ser visto na figura 2.
Tendo em vista o objetivo deste trabalho, é importante também entender o porquê e quando as ramificações citadas acimas ocorrem, para que possamos programá-las em nosso modelo computacional. A ramificação, portanto, é atribuída ao acúmulo de partículas de nutrientes e materiais no ambiente, o que estimula a extensão das hifas dos fungos em sua direção. Dessa maneira, o crescimento e desenvolvimento dos fungos são altamente dependentes e influenciados pela disponibilidade de nutrientes e materiais no ambiente ao seu redor. Apesar desse fato, fungos podem continuar se desenvolvendo até em ambientes com poucos nutrientes, devido ao processo de translocação, no qual os nutrientes previamente absorvidos pelo fungo podem ser transportados internamente, bancando o crescimento do mesmo em locais com deficiência de nutrientes [1].
Mecanismos Gerais dos Modelos
Devido à grande complexidade e exigência computacional, algumas características típicas do desenvolvimento de fungos citadas na primeira seção foram deixadas de lado durante a construção de nosso modelo computacional. No entanto, os mecanismos essenciais para o bom funcionamento da simulação foram mantidos e serão abordados mais profundamente nessa seção.
Crescimento e divisão

Fungos possuem - nas pontas de suas hifas - uma estrutura chamada de Spitzenkörper (corpo superior em alemão). Essa estrutura possui a função de orientar a hifa em seu crescimento e de transformar todo o material de crescimento (nutriente) encontrado em material para as paredes de seus tubos, o que leva à expansão de tamanho do fungo [3]. Por isso, tipicamente, as hifas dos fungos crescem de maneira apical (em suas pontas) e em linha reta, possuindo baixa variação em sua orientação e, quando há variação, as mudanças no ângulo de crescimento ocorrem devido a uma deslocação do Spitzenkörper. Estudos anteriores demonstram que essas variações na angulação são - para certas espécies - normalmente distribuídas [4]. A espécie Mucor hiemalis, por exemplo, possui uma angulação de 56º e desvio padrão de 17º [5].
Os dois tipos de divisões - ou ramificações - citadas anteriormente na seção introdutória variam entre as diferentes espécies de fungo e - da mesma maneira que o crescimento por expansão das hifas - são altamente dependentes da alta concentração de nutrientes em seu ambiente. Além disso, a absorção de novos nutrientes através do Spitzenkörper resulta em uma pressão de Turgor (ou pressão hidrostática) nas paredes internas do fungo, o que também influencia diretamente o processo de ramificação das hifas [1]. Conforme o crescimento do fungo ocorre e suas ramificações acontecem, o processo de encontro e fusão das pontas das hifas dos fungos citado na primeira seção (anastomose) começa a ocorrer. Este processo também pode ocorrer de diferentes maneiras, como pode ser visto na figura 3. Após a ocorrência de ambos os processos, as hifas - agora juntas umas das outras - passam a permitir a transmissão de substâncias internamente entre si.
Crescimento com diferentes distribuição de nutrientes
A distribuição de nutrientes no ambiente influencia diretamente o comportamento das hifas de um fungo.
Construção do modelo computacional
Primeiro modelo de Fungo
Branching na ponta / Computar só os da ponta / Crescimento ocorre só com o nutriente que o ponto final está
Utilizando as informações biológicas apresentadas nas seções anteriores, é possível criar funções matemáticas e computacionais para a programação dos três aspectos fundamentais de uma simulação de micélio de fungos:
Ramificação nas pontas
Extensão/Crescimento
Mapa de Nutrientes
Segundo modelo de Fungo
Após termos realizado o primeiro modelo do fungo com sucesso, partimos para uma nova etapa, na qual passamos a incluir o processo de anastomose juntamente do código original.
Implementação
O Crescimento e divisão dependem do ângulo do segmento originário, uma forma de consegui-lo é através da equação:
É importante lembrar que para x negativos deve-se somar . Esse cálculo, nas duas funções, implementa-se assim:
x = x2 - x1
y = y2 - y1
theta= np.arctan(y/x)
if (x<0) :
theta= theta+ math.pi
aleatorio_theta = random.normalvariate(0, math.pi/4) # angulo de
theta=theta+aleatorio_theta
Crescimento
Soma-se este ângulo com um número aleatório normalmente distribuído com média zero e sigma dependente de dados estatísticos advindos da observação do fungo que pretende-se modelar. Agora com o ângulo modificado podemos adquirir um x e y final para o novo segmento com e respectivamente.
def crecimento (x1,y1,x2,y2):
x = x2 - x1
y = y2 - y1
theta= np.arctan(y/x)
if (x<0) :
theta= theta+ math.pi
aleatorio_theta = random.normalvariate(0, math.pi/4) # angulo de
theta=theta+aleatorio_theta
addx = r * math.cos(theta)
addy = r * math.sin(theta)
fx = x2 + addx
fy = y2 + addy
return (fx,fy)
Divisão
def divisao (x1,y1,x2,y2):
x = x2 - x1
y = y2 - y1
theta= np.arctan(y/x)
if (x<0) :
theta= theta + math.pi
angulodivisao= random.normalvariate(0, math.pi/4)
angulo1= theta- angulodivisao/2
angulo2= theta+ angulodivisao/2
addx1 = r * math.cos(angulo1)
addy1 = r * math.sin(angulo1)
addx2= r * math.cos(angulo2)
addy2= r * math.sin(angulo2)
Ax = x2 + addx1
Ay = y2 + addy1
Bx = x2 + addx2
By= y2 +addy2
return (Ax,Ay,Bx,By)
Intersecção de linhas
def check_anastomose(eventos,xf,yf,xi,yi):
flag = True
for evento in eventos:
if (xi == evento[2] and yi == evento[3]):
continue
if (xi == evento[0] and yi == evento[1]):
continue
D = (evento[3]-evento[1])*(xf-xi) - (evento[2]-evento[0])*(yf-yi)
uA = ((evento[2]-evento[0])*(yi-evento[1]) - (evento[3]-evento[1])*(xi-evento[0]))/D
uB = ((xf-xi)*(yi-evento[1])-(yf-yi)*(xi-evento[0]))/D
flag = True
if (0<=uA<=1) and (0<=uB<=1):
flag = False
if not flag:
xf, yf = get_intersection_point(uA,xf,yf,xi,yi)
break
return xf,yf, flag
Após se encontrar usa a func:
def get_intersection_point(uA,xf,yf,xi,yi):
xf = xi + (uA * (xf-xi))
yf = yi + (uA * (yf-yi))
return xf,yf
Mapa de Nutriente
def find_nearest(array, value):
array = np.asarray(array)
idx = (np.abs(array - value)).argmin()
return array[idx]
def check_nutri(x,y,xmapa,ymapa,mapa_nutri):
near_x = find_nearest(xmapa,x) ## encontra valor mais próximo em x
near_y = find_nearest(ymapa,y) ## encontra valor mais próximo em y
index_x = np.where(xmapa == near_x) ## encontra o index do valor de x
index_y = np.where(ymapa == near_y) ## encontra o index do valor de y
return (mapa_nutri[index_y[0][0]][index_x[0][0]])
def atualiza_mapa_nutri(x,y,xmapa,ymapa,mapa_nutri,preco):
near_x = find_nearest(xmapa,x) ## encontra valor mais próximo em x
near_y = find_nearest(ymapa,y) ## encontra valor mais próximo em y
index_x = np.where(xmapa == near_x) ## encontra o index do valor de x
index_y = np.where(ymapa == near_y) ## encontra o index do valor de y
mapa_nutri[index_y[0][0]][index_x[0][0]] -= preco
return (mapa_nutri)
Referências
- ↑ 1,0 1,1 1,2 1,3 1,4 S. Hopkins. A Hybrid Mathematical Model of Fungal Mycelia: Tropisms, Polarised Growth and Application to Colony Competition, tese de doutorado, 2011.(https://core.ac.uk/download/pdf/6117416.pdf)
- ↑ P. da Silva. Reino Fungi. InfoEscola (2018). Disponível em: https://www.infoescola.com/biologia/reino-fungi. Acesso em: 15 de Maio de 2021.
- ↑ G. Steinberg (2007). "Hyphal growth: a tale of motors, lipids, and the Spitzenkörper". Eukaryotic Cell. 6 (3): 351–360. doi:10.1128/EC.00381-06
- ↑ Molin, P., P. Gervais, J. Lemiere, and T. Davet (1992). Direction of hyphal growth: a relevant parameter in the development of filamentous fungi. Research in Microbiology 143, 777–784. doi: 10.1016/0923-2508(92)90106-x.
- ↑ Hutchinson, S. A., P. Sharma, K. R. Clarke, and I. Macdonald (1980). Control of hyphal orientation in colonies of Mucor hiemalis. Transactions of the British Mycological Society 75, 177–191. doi: 10.1016/S0007-1536(80)80078-7
- ↑ Line to line intersection. Wikipedia, the free encyclopedia. Disponível em: https://en.wikipedia.org/wiki/Line%E2%80%93line_intersection. Acesso em: 20 de Maio de 2021.