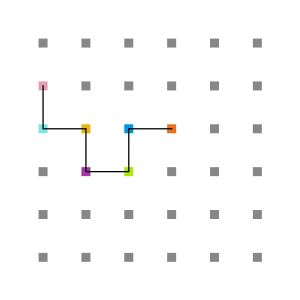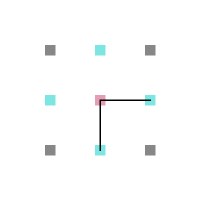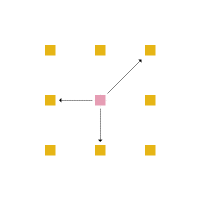Simulação de Enovelamento de Proteína: mudanças entre as edições
| Linha 63: | Linha 63: | ||
== Resultados == | == Resultados == | ||
Em todas as simulações, a posição inicial da cadeia foi esticada horizontalmente no meio da matriz da rede. Os valores de <math>J_{ij}</math> foram sorteados dentro do intervalo [-4, -2], como utilizado no livro. As energias foram medidas em unidades de kB por simplicidade. | Em todas as simulações, a posição inicial da cadeia foi esticada horizontalmente no meio da matriz da rede. Os valores de <math>J_{ij}</math> foram sorteados dentro do intervalo [-4, -2], como utilizado no livro. As energias foram medidas em unidades de kB por simplicidade. | ||
Na figura 1, observamos o gráfico da energia em função do tempo durante a simulação para uma proteína com 15 aminoácidos à temperatura T = 10. As flutuações de energia são grandes pois a temperatura é alta, o que aumenta a probabilidade de alterar a estrutura da proteína. Já na figura 2, temos também a energia em função do tempo para uma proteína com 15 aminoácidos, mas à temperatura T = 1. Nesse caso, as flutuações diminuem consideravelmente e a proteína passa a maior parte do tempo em estados de menor energia. | Na figura 1, observamos o gráfico da energia em função do tempo durante a simulação para uma proteína com 15 aminoácidos à temperatura T = 10. As flutuações de energia são grandes pois a temperatura é alta, o que aumenta a probabilidade de alterar a estrutura da proteína. Já na figura 2, temos também a energia em função do tempo para uma proteína com 15 aminoácidos, mas à temperatura T = 1. Nesse caso, as flutuações diminuem consideravelmente e a proteína passa a maior parte do tempo em estados de menor energia. | ||
Edição das 11h36min de 16 de maio de 2022
Carolina Lenzi
Este trabalho tem como objetivo utilizar o método de Monte Carlo para simular o enovelamento de proteínas.
Proteínas
As proteínas são moléculas com papel fundamental para os seres vivos. Elas atuam em diversos processos biológicos, como o transporte de oxigênio, a catálise de reações químicas, a defesa do organismo, a formação dos hormônios e formação de ossos e tendões. As proteínas são formadas por centenas de aminoácidos ligados em cadeia por ligações covalentes. Existem 20 aminoácidos proteinogênicos que dão origem às proteínas conhecidas e encontradas na natureza. Cada sequência de aminoácidos ligados covalentemente gera uma proteína diferente, com uma função específica no organismo. Essa sequência, bem determinada, é chamada de estrutura primária da proteína.
A proteína tende a se enovelar para minimizar a energia da sua estrutura, relativa às interações não covalentes (como ligações de hidrogênio, ponte salina, interações hidrofóbicas, interações iônicas…) entre os aminoácidos e com o meio. Isso é possível porque as ligações químicas possuem uma certa flexibilidade, que permite a rotação das moléculas. O enovelamento da estrutura primária gera regiões com padrões regulares na proteína, chamadas de estrutura secundária. Essas regiões regulares também tendem a se enovelar, dando origem a estrutura terciária da proteína. A estrutura terciária de menor energia é, geralmente, a conformação funcional da proteína, ou seja, sua configuração em estado biologicamente ativo, que é conhecida como forma nativa. Em alguns casos as estruturas terciárias podem se aglomerar e formar as estruturas quaternárias.
Como a função da proteína está diretamente relacionada com a sua forma nativa, e esta está relacionada com a sequência de aminoácidos que dá origem à proteína, é interessante conseguir prever a estrutura terciária a partir da estrutura primária, com a finalidade de desenvolver proteínas com características específicas. Esse é um dos grandes problemas da bioinformática atualmente. Além da complexidade de compreender a termodinâmica e o mecanismo do enovelamento, a proteína encontra a estrutura ótima em fração de segundo, mas o tempo que seria necessário para testar aleatoriamente as estruturas possíveis para encontrar a de menor energia é maior que a idade do universo, como apontado no paradoxo de Levinthal.
Simulação de Monte Carlo
A simulação do enovelamento foi feita com base no livro do Giordano e envolve um modelo muito simplificado do que acontece na realidade. O código foi implementado em C e os gráficos foram gerados no gnuplot.
Consideramos uma cadeia de N aminoácidos, sorteados dentre os 20 possíveis, para montar a estrutura primária da proteína, representada por um vetor de tamanho N. Aminoácidos em posições adjacentes do vetor são considerados ligados covalentemente. A cadeia é colocada em uma rede quadrada de tamanho NxN, para permitir que a proteína esteja completamente desenovelada (esticada).
Em cada posição da rede, um aminoácido tem no máximo 4 aminoácidos vizinhos mais próximos com os quais pode interagir ou estar ligado (figura 4) e no máximo 8 posições vizinhas para as quais ele pode se mover (figura 5).
Para as interações de não covalentes entre os aminoácidos, consideramos as seguintes forças atuantes:
- Forças de van der Waals: força atrativa para aminoácidos próximos, que perde o efeito com o aumento da distancia entre os aminoacidos;
- Ligações de hidrogênio: ligações entre aminoácidos próximos na rede;
- Interação com a água (hidrofilicidade e hidrofobia): aminoácidos hidrofílicos são atraídos pela água presente no meio e, por isso, tendem a manter a cadeia não enovelada. Já os hidrofóbicos repelem a água do meio e preferem uma estrutura enovelada.
Todas essas forças estão competindo no processo de enovelamento da proteína. Para o modelo, agrupamos essas interações em uma energia Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle J_{ij}} , associada a um par de aminoácidos vizinhos na rede, A(m) e A(n) nas posições i e j, e não ligados covalentemente, ou seja, não adjacentes na cadeia. A energia da estrutura é dada pela soma sobre todos os pares de aminoácidos da proteína:
- Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle E = \sum_{<m,n>}\delta_{m,n}J_{A(m), A(n)}}
onde se os aminoácidos m e n são vizinhos na rede e não estão ligados covalentemente, e zero caso contrário.
A energia J pode ser pensada como uma matriz 20x20 contendo as interações relativas a todos os pares de aminoácidos, porém a matriz é simétrica, portanto, podemos guardar apenas 210 valores correspondentes aos pares distintos de aminoácidos.
Com essas definições, podemos partir para a simulação de Monte Carlo, esquematizada a seguir:
- Inicializar a cadeia e posicionar na rede
- Sortear um aminoácido da cadeia e encontrar sua posição na rede
- Calcular a energia inicial (Ei) da estrutura
- Sortear um dos 8 vizinhos da posição do aminoácido
- Verificar se é possível mover o aminoácido para a posição vizinha sorteada
- Se possível, mover o aminoácido e calcular a energia da nova estrutura (Ef)
- Se Ef < Ei, aceitar o movimento
- Se Ef >= Ei, aceitar o movimento com probabilidade do fator de Boltzmann
- Desfazer o movimento se ele não for aceito
- Contar um tempo de Monte Carlo a cada sorteio de vizinho
- Repetir a partir do passo 2.
A simulação consiste em repetir esses passos (a partir do 2) por longos tempos de Monte Carlo.
Resultados
Em todas as simulações, a posição inicial da cadeia foi esticada horizontalmente no meio da matriz da rede. Os valores de Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle J_{ij}} foram sorteados dentro do intervalo [-4, -2], como utilizado no livro. As energias foram medidas em unidades de kB por simplicidade.
Na figura 1, observamos o gráfico da energia em função do tempo durante a simulação para uma proteína com 15 aminoácidos à temperatura T = 10. As flutuações de energia são grandes pois a temperatura é alta, o que aumenta a probabilidade de alterar a estrutura da proteína. Já na figura 2, temos também a energia em função do tempo para uma proteína com 15 aminoácidos, mas à temperatura T = 1. Nesse caso, as flutuações diminuem consideravelmente e a proteína passa a maior parte do tempo em estados de menor energia.