DM: um primeiro programa: mudanças entre as edições
Sem resumo de edição |
Sem resumo de edição |
||
| Linha 161: | Linha 161: | ||
</pre> | </pre> | ||
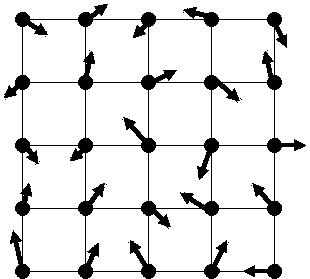
[[Arquivo:initialcondition. | [[Arquivo:initialcondition.jpg]] | ||
A rotina da inicialização das posições recebe os ponteiros das coordenadas <math> x </math> e <math> y </math> das partículas, as coordenadas no tempo anterior <math> x_0 </math> e <math> y_0 </math>, além das velocidades inicias <math> v_x </math> e <math> v_y </math>. Podemos imaginar a disposição inicial das partículas como uma rede quadrada, onde em cada vértice se encontra uma partícula, a distância entre elas é dada por <math> dx=L_x/N_x </math> e <math> dy=L_y/N_y </math>. | A rotina da inicialização das posições recebe os ponteiros das coordenadas <math> x </math> e <math> y </math> das partículas, as coordenadas no tempo anterior <math> x_0 </math> e <math> y_0 </math>, além das velocidades inicias <math> v_x </math> e <math> v_y </math>. Podemos imaginar a disposição inicial das partículas como uma rede quadrada, onde em cada vértice se encontra uma partícula, a distância entre elas é dada por <math> dx=L_x/N_x </math> e <math> dy=L_y/N_y </math>. | ||
Edição das 18h26min de 7 de julho de 2015
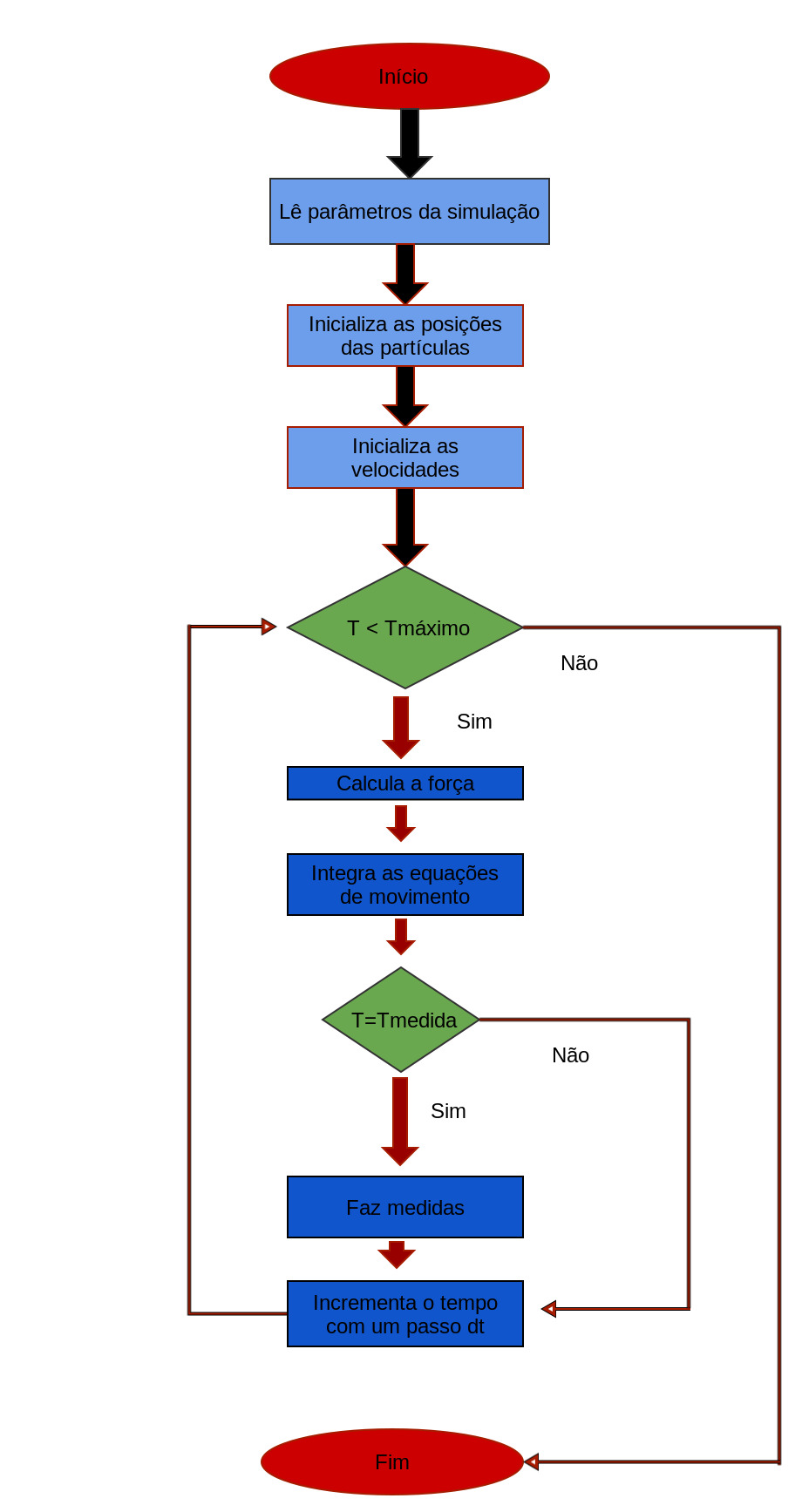
[EM CONSTRUÇÃO] Aqui mostraremos o passo a passo de como montar o primeiro programa para simulação de dinâmica molecular. A linguagem de programação utilizada será o C. Como um primeiro esboço, o programa pode ser organizado como mostra o fluxograma abaixo. Posteriormente, introduziremos partes do código comentando cada etapa. Para o leitor mais familiarizado com DM, ao final da página há o programa completo sem comentários.
A ideia básica da simulação pode ser entendida como: Leitura dos parâmetros da simulação, inicialização das posições das partículas, inicialização das velocidades das partículas, laço temporal onde se calcula a força, integra-se as equações de movimento e, de tempos em tempos, medidas são realizadas.
Abaixo introduzimos partes do código onde começamos declarando as bibliotecas standard utilizadas do C
#include<stdio.h> #include<stdlib.h> #include<math.h>
Está parte é reservada para declarações de constantes que definem os parâmetros da simulação. As constantes podem ser definidas utilizando-se das diretivas de compilação do C através do comando "#define" ou podem ser lidas a partir de um arquivo externo chamado pelo programa. A vantagem do segundo método é que o programa só precisará ser compilado uma única vez para diferentes condições iniciais. Porém, por simplicidade e clareza, optaremos pelo primeiro caso.
#define Nx 16 #define Ny 16 #define Np (Nx*Ny) #define Lx 16 #define Ly 16 #define Rc 4 #define Rc2 (Rc*Rc) #define T 1. #define dt 0.01 #define transient 5. #define total_time 10. #define measure 0.1 #define samples ((total_time-transient)/measure)
Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle N_x \; \mbox{e} \; N_y } representam, respectivamente, o número de partículas nas direções .
representam, respectivamente, o tamanho da rede nas direções .
número total de partículas
define o intervalo de tempo de integração na simulação.
é o raio de corte acima do qual o cálculo da força é desconsiderado.
é o tempo absoluto da simulação.
é o tempo que esperamos até o sistema supostamente equilibrar e, então, começarmos a fazer medidas.
define o intervalo de tempo entre as medições.
nos dá o número total de medidas que serão realizadas
Conforme o programa é construído, novas constantes podem eventualmente aparecer à medida que novas funções são implementadas. Abaixo declaramos as funções essenciais ao programa, onde cada uma delas será detalhadamente explicada no decorrer da implementação.
void initialize_positions(double *xx, double *yy ) ; void initialize_velocities(double *vx, double *vy ) ; void compute_forces( double *ffx, double *ffy, double *xx, double *yy, double *eenergia ) ; void integrate_EqMotion( double *fx, double *fy, double *xx, double *yy, double *xxp, double *yyp, double eenergia, double *energy_pp, double *vxm ) ;
A seguir introduzimos a função principal
void main(void)
{
FILE *file1,*file2;
file1=fopen("energy.dat","w");
file2=fopen("trajectories.dat","w");
unsigned i;
double *x,*y,*xo,*yo,*vx,*vy,*Fx,*Fy,Ep,Ec,Vxm=0.,t;
size_t size=N*sizeof(double);
x= (double*)malloc(size);
y= (double*)malloc(size);
xo=(double*)malloc(size);
yo=(double*)malloc(size);
vx=(double*)malloc(size);
vy=(double*)malloc(size);
Fx=(double*)malloc(size);
Fy=(double*)malloc(size);
Acima são declaradas todas as variáveis essenciais ao programa, assim como a alocação de memória para os vetores como posição, velocidade, força, etc. Com os vetores alocados, podemos agora inicializar as velocidades e posições das partículas na rede
initialize_velocities(vx,vy); initialize_positions(x,y,xo,yo,vx,vy);
Abaixo é mostrada a parte principal do programa, o loop sobre o tempo. A cada passo de tempo, a rotina calcula a força e a energia potencial das partículas a partir de suas posições, na sequência, a rotina atualiza as posições das partículas. O em imprime no arquivo "trajectories.dat" as posições e velocidades de todas as partículas no instante . No testamos se o tempo total é maior que o tempo de equilíbrio e se a medida deve ser feita, caso seja, no arquivo "energy.dat" imprimiremos, respectivamente, o tempo, energia cinética, energia potencial e a energia total.
for(t=0.;t<total_time;t+=dt)
{
compute_forces(Fx,Fy,x,y,&Ep);
integrate_EqMotion(Fx,Fy,x,y,xo,yo,&Ec,&Vxm);
for(i=0;i<N;i++)fprintf(file2,"%lf %lf %lf %lf\n",x[i],y[i],vx[i],vy[i]);
fflush(file2);
rewind(file2);
if( t>transient && (1.-fmod(t/measure,1.) )<0.00001 )
{
fprintf(file1,"%lf %lf %lf %lf\n",t,Ec,Ep,Ec+Ep);
fflush(file1);
}
}
fclose(file1);
fclose(file2);
free(x);
free(y);
free(xo);
free(yo);
free(vx);
free(vy);
free(Fx);
free(Fy);
}
Agora descrevemos detalhadamente todas as rotinas que aparecem na parte principal. Começamos com a inicialização das velocidades, a rotina recebe os ponteiros das velocidades nas direções e . Como condição inicial, todas as partículas recebem uma velocidade aleatória dada por rand()/RAND_MAX que gera um número entre zero e um. Vcx e Vcy são, respectivamente, as componentes da velocidade do centro de massa do sistema nas direções e Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle y } , enquanto V2 é a energia cinética. Para o caso em que as partículas estejam transladando em uma direção preferencial, descontamos suas velocidades pela do centro de massa, além disso, as renormalizamos por um fator de escala que depende da temperatura inicial dada, lembrando que a mesma pode variar no ensamble microcanônico.
void initialize_velocities(double *vx,double *vy)
{
unsigned i;
double Vcx=0.,Vcy=0.,V2=0.,fs;
for(i=0;i<N;i++)
{
vx[i]=1.*rand()/RAND_MAX;
vy[i]=1.*rand()/RAND_MAX;
Vcx+=vx[i];
Vcy+=vy[i];
V2 +=vx[i]*vx[i] + vy[i]*vy[i];
}
Vcx/=N;
Vcy/=N;
V2 /=N;
fs=sqrt(2.*T/V2);
for(i=0;i<N;i++)
{
vx[i]=(vx[i]-Vcx)*fs;
vy[i]=(vy[i]-Vcy)*fs;
}
}
A rotina da inicialização das posições recebe os ponteiros das coordenadas Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle x } e Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle y } das partículas, as coordenadas no tempo anterior Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle x_0 } e Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle y_0 } , além das velocidades inicias Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle v_x } e Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle v_y } . Podemos imaginar a disposição inicial das partículas como uma rede quadrada, onde em cada vértice se encontra uma partícula, a distância entre elas é dada por Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle dx=L_x/N_x } e Falhou ao verificar gramática (MathML com retorno SVG ou PNG (recomendado para navegadores modernos e ferramentas de acessibilidade): Resposta inválida ("Math extension cannot connect to Restbase.") do servidor "https://wikimedia.org/api/rest_v1/":): {\displaystyle dy=L_y/N_y } .
void initialize_positions(double *x,double *y,double *xo,double *yo,double *vx,double *vy)
{
unsigned i;
double dx,dy;
dx=(double)Lx/Nx;
dy=(double)Ly/Ny;
for(i=0;i<N;i++)
{
x[i]=(i%Nx)*dx;
y[i]=(i/Ny)*dy;
xo[i]=x[i]-vx[i]*dt;
yo[i]=y[i]-vy[i]*dt;
}
}