Equação de Langevin
Artur Uhlik Fröhlich e Leonardo Dasso Migotto
O objetivo deste trabalho é resolver computacionalmente a equação de Langevin utilizando o método BAOAB[LEIMKUHLER.] Serão explorados os casos de partículas individuais livres ou sujeitas a um campo potencial, estudando os efeitos da variação do coeficiente de atrito no desvio quadrático médio e na transisão de fases.
Equação de Langevin
Esta equação diferencial estocástica descreve a evolução de um sistema quando sujeito a forças do tipo determinísticas e estocásticas simultâneamente. A sua aplicação mais popular é relativa ao movimento Browniano, o movimento de uma partícula macroscópica imersa em um fluído, sujeita à força de atrito excercida pelas partículas microscópicas do fluído. Neste caso, a equação pode ser escrita como:
Na equação acima, é o coeficiente de atrito e é um ruído estocástico branco, que segue o Teorema Central do Limite com média 0 e desvio padrão relacionado à temperatura, a Constante de Boltzmann, e a massa da partícula. A partir desta expressão, é possível descobrir a relação do coeficiente de difusão do fluído e os valores envolvidos na equação:
Onde é o coeficiente de difusão do meio, é a constante de Boltzmann, é a temperatura e é a massa da partícula macroscópica. Outra relação presente no livro do Frenkel [1], desenvolvida teoricamente, é a do coeficiente de difusão e o deslocamento quadrático médio de uma partícula no meio:
Método BAOAB
O método numérico escolhido para realizar a integração da equação é conhecido como BAOAB, desenvolvido por Leimkuhler e Mattews [2] utilizado para resolver equações diferenciais estocásticas.
Ele é baseado na solução exata para o momentum,
e faz o uso de um método de separação das equações entre as denominadas A, B e O, respectivamente representadas:
O aqui representa um número aleatório Gaussiano que faz o papel do ruído estocástico.
A equação "A" realiza meio passo no tempo da distância, a "B" realiza um meio passo para o momentum e o "O" contabiliza a contribuição estocástica equação.
Essas equações podem formar vários algoritmos de integração mas o utilizado nesse trabalho será o BAOAB:
É importante lembrar que entre os dois últimos passos é necessário atualizar o termo , já que ele pode depender de termos já atualizados como ou .
Implementação do Método BAOAB
Um exemplo de implementação desse método feito foi para a partícula livre. Nota-se que nesse caso a partícula não é afetada por um campo potencial então os passos do método que envolvem a força serão desconsiderados.
A função a seguir se encontra em um código (orientado a objeto) de autoria do integrante Artur e que pode ser acessado aqui.
def baoab_livre(self, dt, exp, sqexp, sqt, G):
'''
dt: discretização do tempo
exp: termo referente a primeira exponencial da eq.3
sqexp: termo da raiz quadrada com exponencial da eq.3
sqt: termo da raiz quadrada com a Temperatura da eq.3
G: o vetor G da eq.3
'''
# 1/2 passo da distância
self.pos[0] += self.vel[0]*(dt/2)
self.pos[1] += self.vel[1]*(dt/2)
# Passo estocástico
self.vel[0] = exp*self.vel[0] + sqexp*sqt*G[0]
self.vel[1] = exp*self.vel[1] + sqexp*sqt*G[1]
# Atualização final da posição
self.pos[0] += self.vel[0]*(dt/2)
self.pos[1] += self.vel[1]*(dt/2)
Essa função representa um passo do laço temporal utilizando o método BAOAB. Os atributos em forma de lista ".pos" e ".vel" representam as componentes x e y respectivamente da posição e velocidade do objeto partícula.
Um exemplo de funcionamento do código é a animação a seguir:
O código foi executado usando a semente de números aleatórios 420.
Implementação do Deslocamento Quadrático Médio
Segue abaixo uma implementação do cálculo do deslocamento quadrático médio. A partir de um array de posições de uma única partícula, o código faz os cálculos necessários retornando um array com o MSD em cada instante de tempo.
A função a seguir se encontra em um código de autoria do integrante Leonardo. Este código abre os dados gerados por outro programa, que salva todas informações em arquvios, e permite visualizar propriedades como o MSD, a distribuição do momentum, entre outras coisas.
if modo == "MSD":
novotamanho = int(1 + ((nsalvos-1)/divisoes))
desviodividido = np.zeros(novotamanho)
indice = novotamanho - 1
xx = xy[:, 0]
yy = xy[:, 1]
for i in range(divisoes):
xini = xx[i*indice]
yini = yy[i*indice]
dx = xx[i*indice : 1+(i+1)*indice] - xini
dy = yy[i*indice : 1+(i+1)*indice] - yini
desviodividido += dx**2 + dy**2
desviodividido = (desviodividido/divisoes)[1:]
eixox = np.linspace(0, int(tmax/divisoes), int((nsalvos - 1)/divisoes) + 1)[1:]
difusivo = desviodividido[int(10/(dt*gaminha)):]
dezao = np.mean(difusivo/eixox[int(10/(dt*gaminha)):])/4
eixox = np.log10(eixox)
desviodividido = np.log10(desviodividido)
balistico = desviodividido[:int(1/(dt*gaminha))]
difusivo = desviodividido[int(10/(dt*gaminha)):]
bal = np.polyfit(eixox[:int(1/(dt*gaminha))], balistico, 1)
dif = np.polyfit(eixox[int(10/(dt*gaminha)):], difusivo, 1)
reta1 = bal[0]*eixox + bal[1]
reta2 = dif[0]*eixox + dif[1]
Para melhorar a precisão do MSD, o programa permite "fatiar" o array original de posições, considerando cada fatia das distâncias como uma execução do programa independente (o número de fatias é a variável "divisoes", definida ao iniciar o programa). Um número maior de fatias resulta em um tempo reduzido percorrido pela partícula em cada fatia.
Após definir a posição de cada fatia, o programa itera sobre elas, calculando os valores de MSD para cada instante de cada uma e somando a um array vazio. Por fim, é feito a média dos MSDs pelo número de partículas neste array. Dado o modo preferido de visuaizar os dados, o array é transformado para escala logarítmica. Em seguida, dois arrays criados a partir do do MSD: um deles contém somente pontos do período de difusão balística, enquanto o outro possui pontos da difusão normal. É feita a regressão linear de cada um destes conjuntos de daods, a fim de encontrar a inclinação das retas ajustadas aos dados (e confirmar que estes períodos possuem, de fato, essas características difusivas, como veremos a seguir).
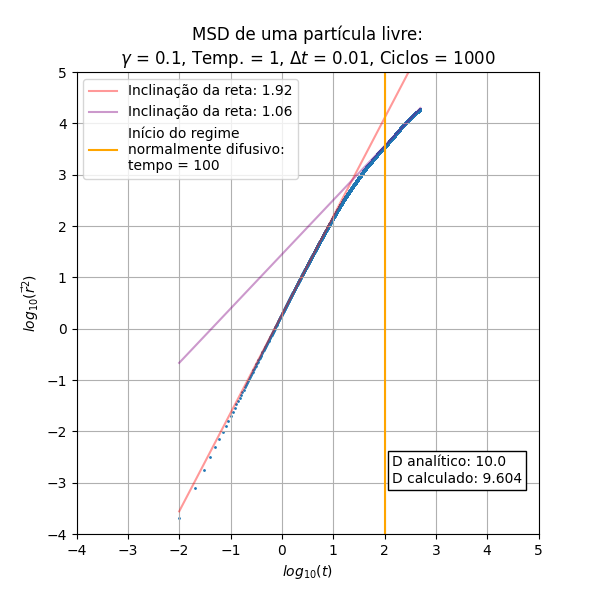
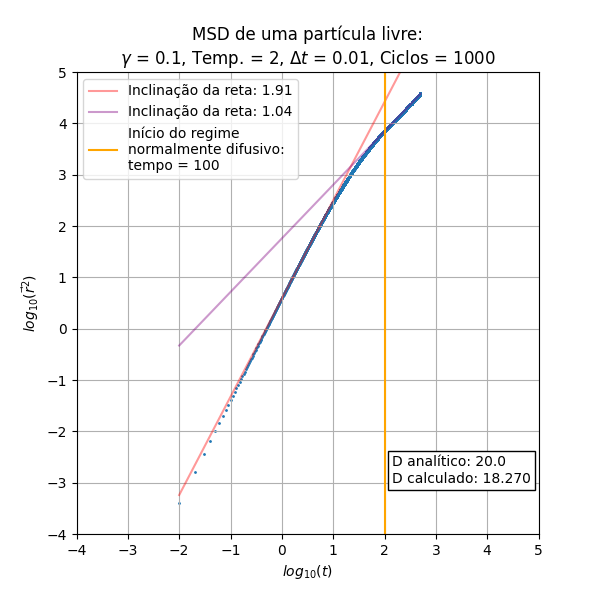
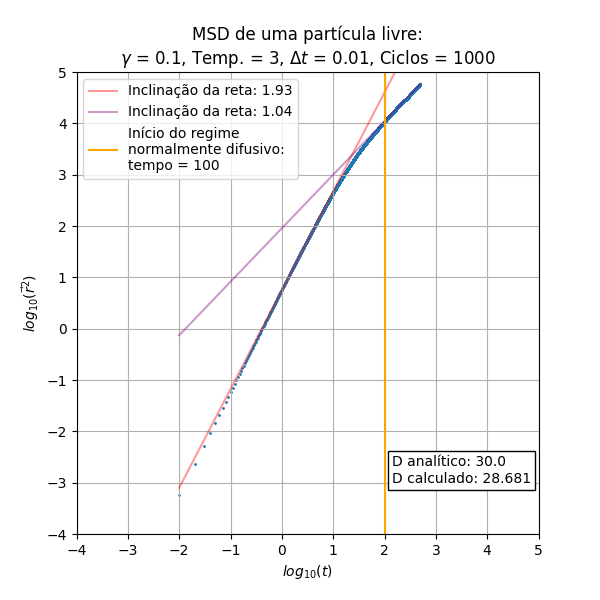
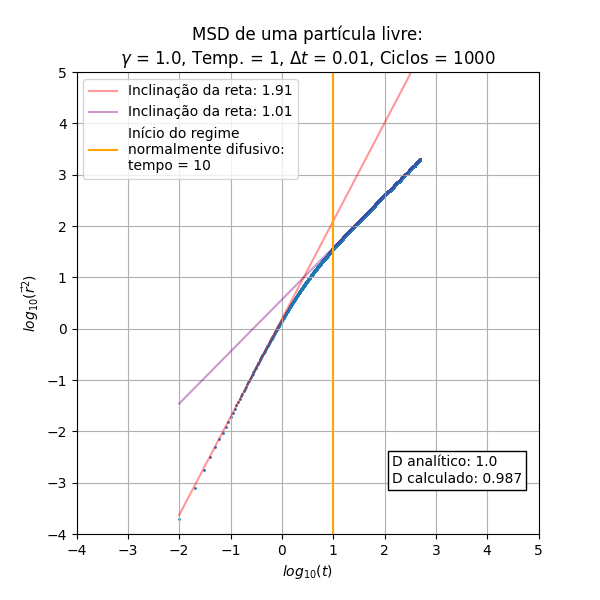
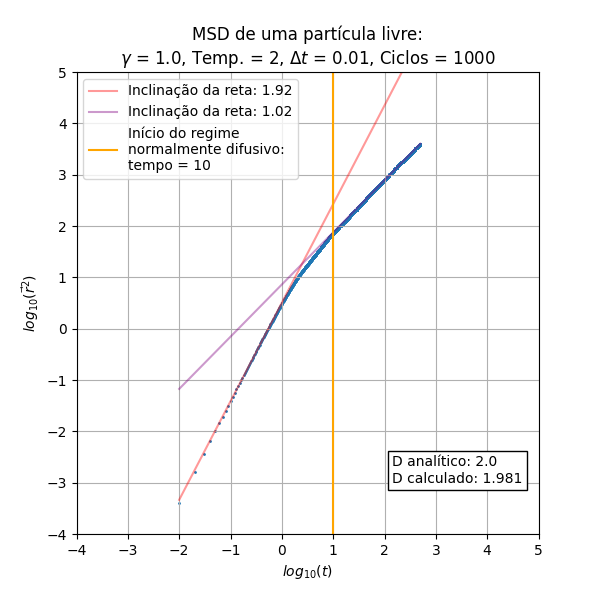
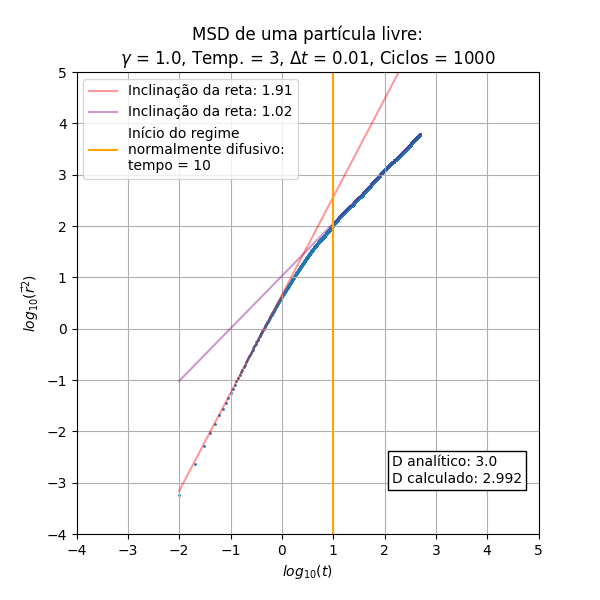
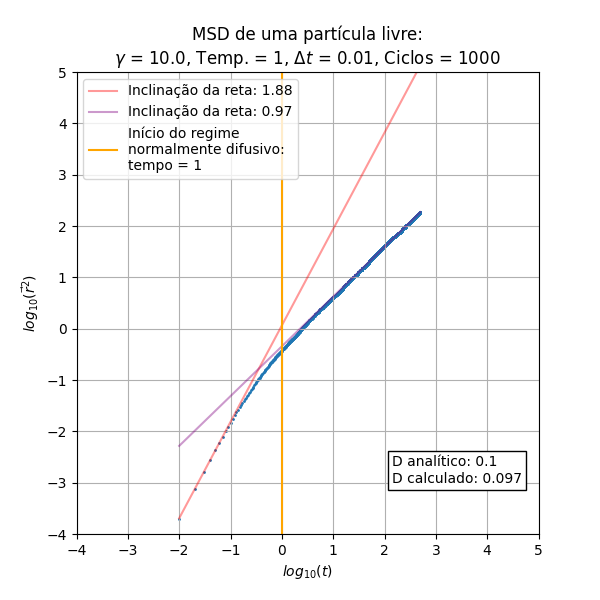
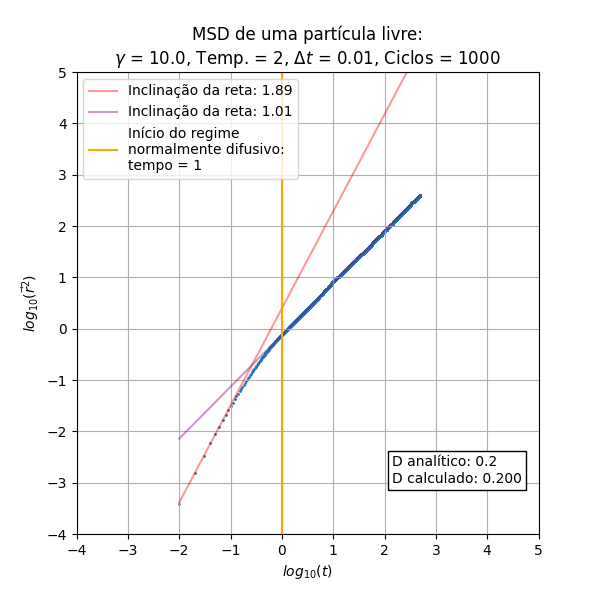
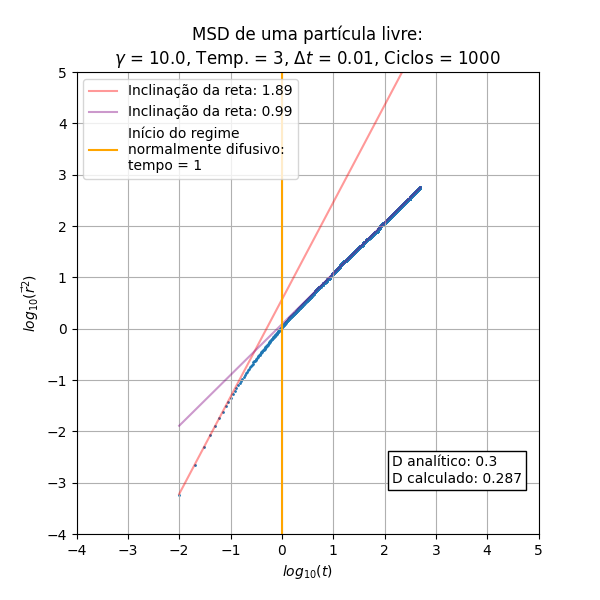
Análise do MSD
Os resultados a serem analisados são originados do código (procedural) do integrante Leonardo.
Foram executadas diferentes vezes a simulação para compararmos a influência do e da temperatura no deslocamento quadrático médio.
Temperaturas de 1-3 e :
Temperaturas de 1-3 e :
Temperaturas de 1-3 e :
De início pode-se notar que o escala de maneira proporcional a (balístico) no início do processo, e depois de um determinado tempo obtemos uma relação linear que é a parte difusiva do processo. Essas inclinações das retas nos regimes balístico e difusivo são constantes em relação ao e à temperatura. Outro aspecto constante é a temperatura, que não gera aparente diferença nos gráficos de MSD
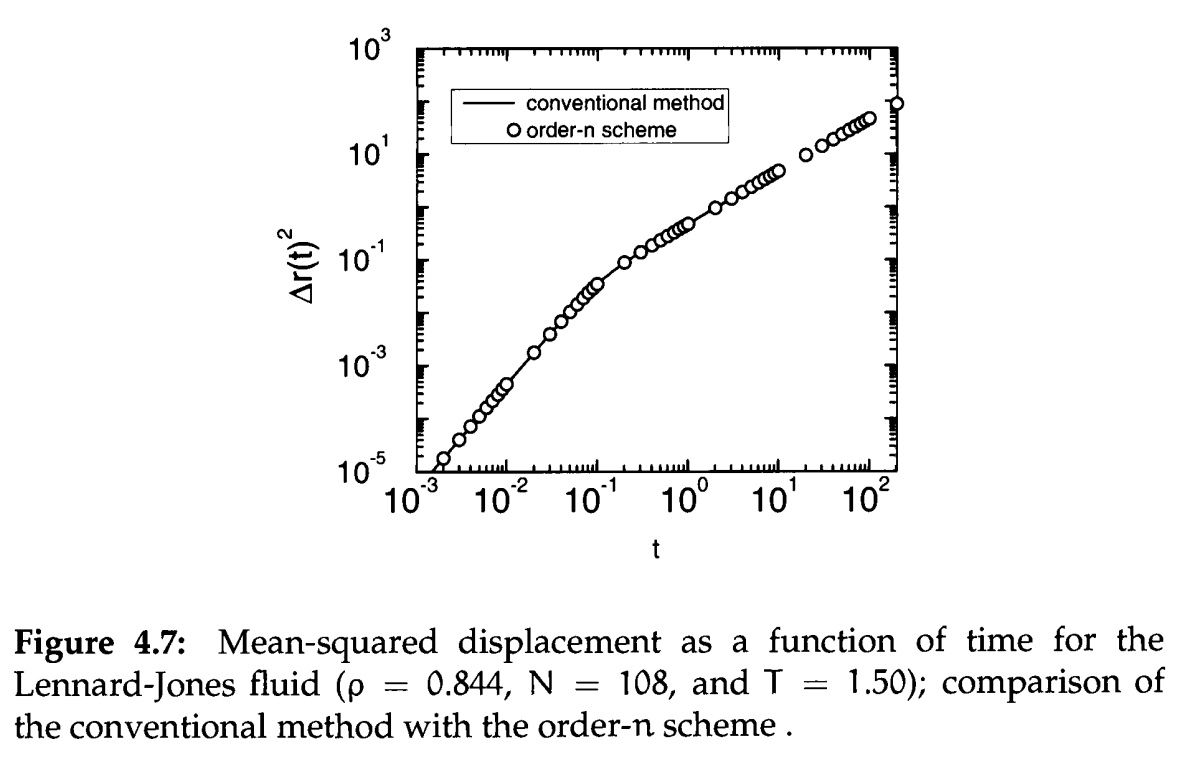
Uma referência da literatura a respeito do MSD (mas no contexto de dinâmica molecular usando o potencial de Lennard-Jones) se encontra no livro do Frenkel, expressa por essa imagem:
Outro aspecto importante a se ressaltar é a concordância do cálculo do coeficiente de difusão através da equação (I) e da relação de Einstein em (II), obtida através dos pontos presentes na parte difusiva.
Uma relação interessante encontrada foi a maneira como escala a mudança de regimes, indicada pela reta perpendicular ao eixo do tempo. Podemos encontrar uma aproximação para o tempo em que o regime difusivo inicia na simulação a partir da relação dependente de :
Referências
- ↑ Daan Frenkel and Berend Smit. 2001. Understanding Molecular Simulation (2nd. ed.). Academic Press, Inc., USA.
- ↑ Leimkuhler, B., & Matthews, C. (2015). Molecular Dynamics: With Deterministic and Stochastic Numerical Methods. (Interdisciplinary Applied Mathematics; Vol. 39). Springer. https://doi.org/10.1007/978-3-319-16375-8